
【研究】RNA-seq01:準備編
こんにちは。あるいは、こんばんは。
次世代シーケンサーを用いたRNA-seqなるものを、Stable Diffusionの片手間に勉強してみました。
備忘録ですので詳細は、他の記事・市販書籍をご確認いただくことをおすすめします。。。
環境構築
データ解析のための基本となる環境構築は、なかなか検索してもベストなものが判断できないものです。
今回はメインマシンのMacBook Pro(14inch,2021), Apple M1 Max, Ventura 13.6にて環境構築とデータ解析にチャレンジしたいと思います(2023/10/7)。
Minicondaをインストールするよ
Anacondaの軽量版「Miniconda」をインストールします。
以下のリンク先から、OSやCPUに対応した適切なインストーラをダウンロードしましょう。

Miniconda3 macOS Apple M1 64-bit bashをダウンロードして、シェルスクリプト(〜.shファイル)を実行します。
ダウンロード先のフォルダを準備しておき、「cd」コマンドにてディレクトリを移動します。
ターミナルAppに以下を入力して実行してみましょう。
cd "/Users/user/Dropbox/RNAseq/01"
wget https://repo.anaconda.com/miniconda/Miniconda3-latest-MacOSX-arm64.sh
source /Users/user/Dropbox/RNAseq/01/Miniconda3-latest-MacOSX-arm64.shWelcome to Miniconda3 py311_23.5.2-0
In order to continue the installation process, please review the license
agreement.
Please, press ENTER to continue
※「wget」コマンドにてダウンロードできるのですが、Homebrewを導入してインストールしておかないと実行できないかも知れません。
※その場合には、リンク先からダウンロードして、保存先のフォルダ("/Users/user/Dropbox/RNAseq/01"など)に入れると良いと思います。
※(おまけ1)Homebrewのインストールが参考になれば幸いです。
ここで注意!!
(「ENTER」連打後のイライラ回避のため・・・)
「ENTER」をひたすら押してライセンス条項を送ることになりますが、ここで注意です!
途中で [yes|no] クエスチョンがあり、誤って「ENTER」を押して飛ばしてしまうと、最初からになってしまいます。
イライラしますよ〜(ーへー;)
Miniconda3 macOS Apple M1 64-bit pkgをダウンロードしてダブルクリックしたほうが良いのかも知れません。。。
Miniconda3 will now be installed into this location:
/Users/user/miniconda3
- Press ENTER to confirm the location
- Press CTRL-C to abort the installation
- Or specify a different location below
デフォルトのインストール先は「/Users/user/miniconda3」となります。
「ENTER」でインストールしましょう。
Do you accept the license terms? [yes|no]
[no] >>>
「yes」を入力しましょう。
・・・
Thank you for installing Miniconda3!
インストール完了です!
お疲れ様(?)でした。
Anacondaのchannelsを設定するよ
Biocondaにて推奨されている順番でコードを実行していきます。
conda config --add channels defaults
conda config --add channels bioconda
conda config --add channels conda-forge
conda config --set channel_priority strictDockerをインストールするよ
Dockerはコンテナと呼ばれる仮想環境を構築するもので、同じデータ解析環境を共有する際にとても便利です。
プロテオーム解析のために準備したことがありますが、パッケージやライブライをふんだんに取り入れたためにパンクして上手く動かなかったのですが。。。
RNA-seqのデータ解析のため、再チャレンジです!
Docker Desktopのサイトから、OS/CPUに適したインストーラを選択してダウンロードしましょう。

Dockerを起動しましょう。
青いクジラのアイコンをクリックして起動します。
右上にある「歯車」アイコンをクリックして設定を変更しておきます。
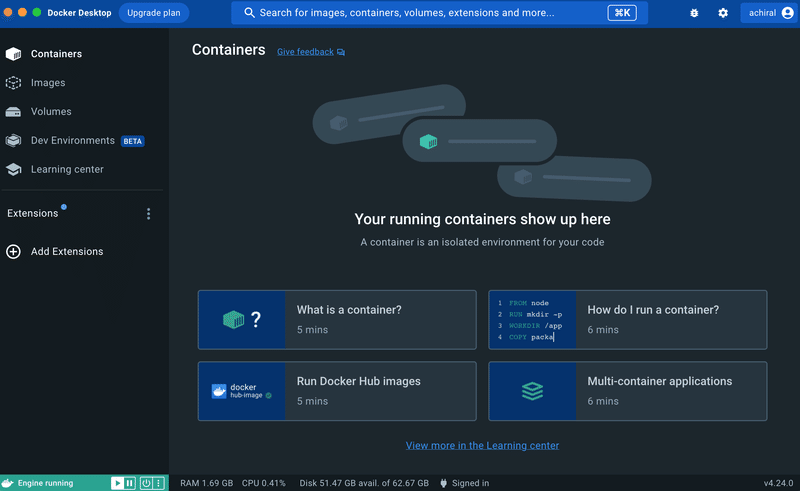
Resources > Advancedにて値を増やしておきます。
・CPU limit:10(デフォルト??)
・Memory limit:48 GB(増やしました)
割当が少ないとプログラムが途中で止まることがあるようです。
Dockerがインストールされているか確認しましょう。
ターミナルAppで以下のコードを実行します。
docker run --rm -it beezu/cmatrix文字が滝のように流れます。
「q」または「Ctrl + C」で停止します。

BLASTをインストールするよ
NCBIのサイトにアクセスします。
「〜.dmg」ファイルをダウンロードします。
ターミナルAppからダウンロードするコードは以下のとおりです。
cd "/Users/user/Dropbox/RNAseq/01"
wget https://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/ncbi-blast-2.14.1+.dmg「ncbi-blast-2.14.1+.dmg」をダブルクリックして、添加したフォルダ内の「ncbi-blast-2.14.1+.pkg」をダブルクリックします。
(バージョンは2023/10/7時点の最新ものになると思います)

インストール先は「/usr/local/ncbi/blast」となります。
コンテンツを調べるには「ls」コマンドを使用します。
ls -R /usr/local/ncbi/blastbin doc
/usr/local/ncbi/blast/bin:
blast_formatter get_species_taxids.sh
blast_formatter_vdb legacy_blast.pl
blast_vdb_cmd makeblastdb
blastdb_aliastool makembindex
blastdbcheck makeprofiledb
blastdbcmd psiblast
blastn rpsblast
blastn_vdb rpstblastn
blastp segmasker
blastx tblastn
cleanup-blastdb-volumes.py tblastn_vdb
convert2blastmask tblastx
deltablast update_blastdb.pl
dustmasker windowmasker
/usr/local/ncbi/blast/doc:
README.txt
(おまけ1)バージョン確認
使用したプログラム等に関しては、再現性を担保するためにバージョン管理が重要になります。
バージョンの確認にはオプションを指定します。
wget --versionGNU Wget 1.21.3 built on darwin22.1.0.
/usr/local/ncbi/blast/bin/blastn -versionblastn: 2.14.1+
Package: blast 2.14.1, build Aug 1 2023 13:52:46
(おまけ2)Anacondaのアップデート
conda update --all(おまけ3)Homebrewのインストール
まず、Mac App StoreからXcodeをインストールします。
とても時間がかかります。
ターミナルAppにて以下を順に実行して、
1)command line developer tools
2)Homebrew
3)wget
をインストールします。
xcode-select --install
/bin/bash -c "$(curl -fsSL https://raw.githubusercontent.com/Homebrew/install/HEAD/install.sh)"
brew install wget詳細はHomebrewのホームページを確認してください。
この記事が気に入ったらサポートをしてみませんか?
