
[Python]健診データを240次元から2次元に圧縮してみた:PCA, MDS, t-SNE, UMAPによる次元削減
はじめに
こんにちは、機械学習勉強中のあおじるです。
以前の記事では、次元削減(次元圧縮)の手法を使って医療費データ(160次元)を2次元に圧縮してみました。今回は、健診データ(240次元)について、同様に2次元に圧縮してみます。
言語はPython、環境はGoogle Colaboratoryを使用しました。
使用するデータ
データは、前回の記事で、全国健康保険協会(協会けんぽ)の生活習慣病予防健診の時系列データ「健診結果基本情報」から作成した、10年度×47都道府県別の健診項目のデータ(性別、年齢階級別の健診項目ごとの平均値)df_yt_K15_sn を使います。
(10年×47都道府県)×(15健診項目×性別2区分×年齢階級8区分)
= 470行 × 240次元
の形のデータです。
$$
\def\arraystretch{1.5}
\begin{array}{c:c|c:c:c:c}
\textsf{y} & \textsf{t} & \textsf{WC\_1\_1} & \textsf{WC\_1\_2} & \cdots & \textsf{eGFR\_2\_8} \\ \hline
2010 & 1 & {} & {} & {} & {} \\
2010 & 2 & {} & {} & {} & {} \\
\vdots & \vdots & \vdots & \vdots & \ddots & \vdots \\
2019 & 47 & {} & {} & {} & {}
\end{array}
$$
y:年度
2010~2019 の10年度分t:都道府県
1:北海道、・・・、47:沖縄 の47都道府県K15_s_n:性別(s)、年齢階級(n)別の15の健診項目の平均値(K15)
K15:15の健診項目(WC、BMI、SBP、DBP、TC、TG、HDLC、LDLC、GOT、GPT、yGTP、FBS、UA、Cr、eGFR)の平均値
s:性別
1:男性、2:女性n:年齢階級
1:35~39歳、2:40~44歳、・・・、7:65~69歳、8:70歳以上
# データ
import pandas as pd
df = pd.read_csv('./df_yt_K15_sn.csv')
print(df.shape)
# (470, 242)
print(df.columns)
# Index(['y', 't', 'WC_1_1', 'WC_1_2', 'WC_1_3', 'WC_1_4', 'WC_1_5', 'WC_1_6',
# 'WC_1_7', 'WC_1_8',
# ...
# 'eGFR_1_7', 'eGFR_1_8', 'eGFR_2_1', 'eGFR_2_2', 'eGFR_2_3', 'eGFR_2_4',
# 'eGFR_2_5', 'eGFR_2_6', 'eGFR_2_7', 'eGFR_2_8'],
# dtype='object', length=242)スケールの異なる健診項目データですので、前回と同じく、スケーリングして用います。
# 数値部分のみ取り出し
X = df.iloc[:,2:]
print(X.shape)
# (470, 240)
# スケーリング
from sklearn import preprocessing
scaler = preprocessing.MinMaxScaler()
X = scaler.fit_transform(X)
print(X.shape)
# (470, 240)次元削減(次元圧縮)
この240次元のデータを医療費データの場合と同様に、2次元へ圧縮します。次元削減(次元圧縮)の方法として次の方法をそれぞれ試しました。
t-SNE(t -distributed Stochastic Neighborhood Embedding;t分布型確率的近傍埋め込み法)
PCA & UMAP(PCAで次元削減した結果をさらにUMAPで次元削減)
結果の表示は、医療費データの場合と同様に、年度(y)ごと、都道府県(t)ごとに色分けして図示しました。色分けにはseabornのカラーパレットを使いました。
%matplotlib inline
import matplotlib.pyplot as plt
import seaborn as sns
from sklearn import preprocessing
le = preprocessing.LabelEncoder()
# 年度y
label_y = le.fit_transform(df['y'])
cp = sns.color_palette('hls', n_colors=10+1)
color_y = [cp[x] for x in label_y]
# 都道府県t
label_t = le.fit_transform(df['t'])
cp = sns.color_palette('hls', n_colors=47+1)
color_t = [cp[x] for x in label_t]0.PCA
比較のために、前回の記事のPCA(Principal Component Analysis;主成分分析)の結果を再掲しておきます。
# PCA
from sklearn.decomposition import PCA
pca = PCA(n_components=2)
PC = pca.fit_transform(X)
print(PC.shape) # (470, 240)
i = 0
j = 1
print('PC{} x PC{}'.format(i+1, j+1))
plt.figure(figsize=(12,5))
plt.subplot(1, 2, 1)
plt.title('PC{} x PC{}, colored by y'.format(i+1, j+1))
plt.xlabel('PC{}'.format(i+1))
plt.ylabel('PC{}'.format(j+1))
plt.scatter(x=PC[:,i], y=PC[:,j], c=color_y)
plt.subplot(1, 2, 2)
plt.title('PC{} x PC{}, colored by t'.format(i+1, j+1))
plt.xlabel('PC{}'.format(i+1))
plt.ylabel('PC{}'.format(j+1))
plt.scatter(x=PC[:,i], y=PC[:,j], c=color_t)
plt.show()
print('PC{} x PC{}'.format(i+1, j+1))
xmin, xmax = min(PC[:,i]), max(PC[:,i])
ymin, ymax = min(PC[:,j]), max(PC[:,j])
plt.figure(figsize=(12,5))
plt.subplot(1, 2, 1)
plt.title('PC{} x PC{}, colored by y'.format(i+1, j+1))
plt.xlabel('PC{}'.format(i+1))
plt.ylabel('PC{}'.format(j+1))
plt.xlim(xmin-(xmax-xmin)/20, xmax+(xmax-xmin)/20)
plt.ylim(ymin-(ymax-ymin)/20, ymax+(ymax-ymin)/20)
for p in range(len(df)):
plt.text(x=PC[p,i], y=PC[p,j], s=df.iloc[p,1],
ha='center', va='center', fontsize=10, color=color_y[p])
plt.subplot(1, 2, 2)
plt.title('PC{} x PC{}, colored by t'.format(i+1, j+1))
plt.xlabel('PC{}'.format(i+1))
plt.ylabel('PC{}'.format(j+1))
plt.xlim(xmin-(xmax-xmin)/20, xmax+(xmax-xmin)/20)
plt.ylim(ymin-(ymax-ymin)/20, ymax+(ymax-ymin)/20)
for p in range(len(df)):
plt.text(x=PC[p,i], y=PC[p,j], s=df.iloc[p,1],
ha='center', va='center', fontsize=10, color=color_t[p])
plt.show()

1.MDS
MDS(Multi-Dimensional Scaling;多次元尺度構成法)を試してみます。
1-1.Metric MDS
使用する距離として、通常のユークリッド距離(euclidean_distances)、マンハッタン距離(manhattan_distances)、コサイン距離(cosine_distances)を試しました。
距離:https://scikit-learn.org/stable/modules/classes.html#pairwise-metrics
# MDS, euclidean_distances
from sklearn.manifold import MDS
from sklearn.metrics.pairwise import euclidean_distances
reducer = MDS(n_components=2, dissimilarity='precomputed', random_state=0)
X_transformed = reducer.fit_transform(euclidean_distances(X))
print(X_transformed.shape) # (470, 2)
alg = 'MDS' # algorithm
param = 'euclidean_distances' # parameter
i = 0
j = 1
print('{}, {}'.format(alg,param))
plt.figure(figsize=(12,5))
plt.subplot(1, 2, 1)
plt.title('{}, {}, colored by y'.format(alg,param))
plt.xlabel('{}_{}'.format(alg,i+1))
plt.ylabel('{}_{}'.format(alg,j+1))
plt.scatter(x=X_transformed[:,i], y=X_transformed[:,j], c=color_y)
plt.subplot(1, 2, 2)
plt.title('{}, {}, colored by t'.format(alg,param))
plt.xlabel('{}_{}'.format(alg,i+1))
plt.ylabel('{}_{}'.format(alg,j+1))
plt.scatter(x=X_transformed[:,i], y=X_transformed[:,j], c=color_t)
plt.show()
print('{}, {}'.format(alg,param))
xmin, xmax = min(X_transformed[:,i]), max(X_transformed[:,i])
ymin, ymax = min(X_transformed[:,j]), max(X_transformed[:,j])
plt.figure(figsize=(12,5))
plt.subplot(1, 2, 1)
plt.title('{}, {}, colored by y'.format(alg,param))
plt.xlabel('{}_{}'.format(alg,i+1))
plt.ylabel('{}_{}'.format(alg,j+1))
plt.xlim(xmin-(xmax-xmin)/20, xmax+(xmax-xmin)/20)
plt.ylim(ymin-(ymax-ymin)/20, ymax+(ymax-ymin)/20)
for p in range(len(df)):
plt.text(x=X_transformed[p,i], y=X_transformed[p,j], s=df.iloc[p,1],
ha='center', va='center', fontsize=10, color=color_y[p])
plt.subplot(1, 2, 2)
plt.title('{}, {}, colored by t'.format(alg,param))
plt.xlabel('{}_{}'.format(alg,i+1))
plt.ylabel('{}_{}'.format(alg,j+1))
plt.xlim(xmin-(xmax-xmin)/20, xmax+(xmax-xmin)/20)
plt.ylim(ymin-(ymax-ymin)/20, ymax+(ymax-ymin)/20)
for p in range(len(df)):
plt.text(x=X_transformed[p,i], y=X_transformed[p,j], s=df.iloc[p,1],
ha='center', va='center', fontsize=10, color=color_t[p])
plt.show()# MDS, manhattan_distances
from sklearn.manifold import MDS
from sklearn.metrics.pairwise import manhattan_distances
reducer = MDS(n_components=2, dissimilarity='precomputed', random_state=0)
X_transformed = reducer.fit_transform(manhattan_distances(X))
print(X_transformed.shape) # (470, 2)# MDS, cosine_distances
from sklearn.manifold import MDS
from sklearn.metrics.pairwise import cosine_distances
reducer = MDS(n_components=2, dissimilarity='precomputed', random_state=0)
X_transformed = reducer.fit_transform(cosine_distances(X))
print(X_transformed.shape) # (470, 2)


だいたい似たような結果になりました。
1-2.Nonmetric MDS
# Nonmetric MDS
from sklearn.manifold import MDS
reducer = MDS(n_components=2, metric=False, random_state=0)
X_transformed = reducer.fit_transform(X)
print(X_transformed.shape) # (470, 2)
これはうまくいきませんでした。
2.t-SNE
t-SNE(t -distributed Stochastic Neighborhood Embedding;t分布型確率的近傍埋め込み法)を試してみます。
パラメータperplexity(デフォルトでは30.0)を変えて実行してみます(小さい方から大きい方へ)。
# t-SNE
from sklearn.manifold import TSNE
list_perplexity = [2.0,3.0,5.0,10.0,15.0,20.0,30.0,50.0,75.0,100.0,
110.0,120.0,130.0,140.0,150.0,200.0,300.0,400.0]
for perplexity in list_perplexity:
reducer = TSNE(n_components=2, perplexity=perplexity, random_state=0)
X_transformed = reducer.fit_transform(X)
print(X_transformed.shape) # (470, 2)
alg = 't-SNE' # algorithm
param = 'perplexity='+str(perplexity) # parameter
i = 0
j = 1
print('{}, {}'.format(alg,param))
plt.figure(figsize=(12,5))
plt.subplot(1, 2, 1)
plt.title('{}, {}, colored by y'.format(alg,param))
plt.xlabel('{}_{}'.format(alg,i+1))
plt.ylabel('{}_{}'.format(alg,j+1))
plt.scatter(x=X_transformed[:,i], y=X_transformed[:,j], c=color_y)
plt.subplot(1, 2, 2)
plt.title('{}, {}, colored by t'.format(alg,param))
plt.xlabel('{}_{}'.format(alg,i+1))
plt.ylabel('{}_{}'.format(alg,j+1))
plt.scatter(x=X_transformed[:,i], y=X_transformed[:,j], c=color_t)
plt.show()
print('{}, {}'.format(alg,param))
xmin, xmax = min(X_transformed[:,i]), max(X_transformed[:,i])
ymin, ymax = min(X_transformed[:,j]), max(X_transformed[:,j])
plt.figure(figsize=(12,5))
plt.subplot(1, 2, 1)
plt.title('{}, {}, colored by y'.format(alg,param))
plt.xlabel('{}_{}'.format(alg,i+1))
plt.ylabel('{}_{}'.format(alg,j+1))
plt.xlim(xmin-(xmax-xmin)/20, xmax+(xmax-xmin)/20)
plt.ylim(ymin-(ymax-ymin)/20, ymax+(ymax-ymin)/20)
for p in range(len(df)):
plt.text(x=X_transformed[p,i], y=X_transformed[p,j], s=df.iloc[p,1],
ha='center', va='center', fontsize=10, color=color_y[p])
plt.subplot(1, 2, 2)
plt.title('{}, {}, colored by t'.format(alg,param))
plt.xlabel('{}_{}'.format(alg,i+1))
plt.ylabel('{}_{}'.format(alg,j+1))
plt.xlim(xmin-(xmax-xmin)/20, xmax+(xmax-xmin)/20)
plt.ylim(ymin-(ymax-ymin)/20, ymax+(ymax-ymin)/20)
for p in range(len(df)):
plt.text(x=X_transformed[p,i], y=X_transformed[p,j], s=df.iloc[p,1],
ha='center', va='center', fontsize=10, color=color_t[p])
plt.show()
















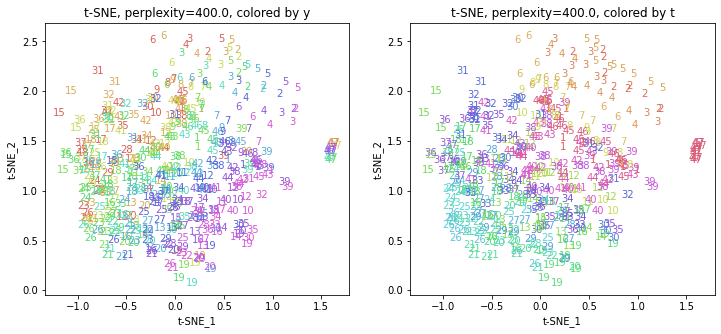
パラメータperplexityの値が小さいと、都道府県ごとにまとまっていて(都道府県が完全に分離)、年度の向きはばらばらで、パラメータperplexityの値を上げていくと、都道府県同士が近づいていき、年度の向きがそろっていくのは、医療費データの場合と同様でした。ただ、医療費データの場合のように完全には向きがそろいませんでした。
また、47沖縄は他の都道府県と離れています。


3.UMAP
UMAP(Uniform Manifold Approximation and Projection)を試してみます。
パラメータn_neighbors(デフォルトでは15)を変えて実行してみます(小さい方から大きい方へ)。
# UMAP
! pip install umap-learn
import umap.umap_ as umap
list_n_neighbors = [2,3,4,5,6,7,8,9,10,11,12,13,14,15,16,17,18,19,20,25,30,35,
40,45,50,60,70,80,90,100,150,200,300,400]
for n_neighbors in list_n_neighbors:
reducer = umap.UMAP(n_components=2, n_neighbors=n_neighbors, random_state=0)
X_transformed = reducer.fit_transform(X)
print(X_transformed.shape) # (470, 2)
alg = 'UMAP' # algorithm
param = 'n_neighbors='+str(n_neighbors) # parameter
i = 0
j = 1
print('{}, {}'.format(alg,param))
plt.figure(figsize=(12,5))
plt.subplot(1, 2, 1)
plt.title('{}, {}, colored by y'.format(alg,param))
plt.xlabel('{}_{}'.format(alg,i+1))
plt.ylabel('{}_{}'.format(alg,j+1))
plt.scatter(x=X_transformed[:,i], y=X_transformed[:,j], c=color_y)
plt.subplot(1, 2, 2)
plt.title('{}, {}, colored by t'.format(alg,param))
plt.xlabel('{}_{}'.format(alg,i+1))
plt.ylabel('{}_{}'.format(alg,j+1))
plt.scatter(x=X_transformed[:,i], y=X_transformed[:,j], c=color_t)
plt.show()
print('{}, {}'.format(alg,param))
xmin, xmax = min(X_transformed[:,i]), max(X_transformed[:,i])
ymin, ymax = min(X_transformed[:,j]), max(X_transformed[:,j])
plt.figure(figsize=(12,5))
plt.subplot(1, 2, 1)
plt.title('{}, {}, colored by y'.format(alg,param))
plt.xlabel('{}_{}'.format(alg,i+1))
plt.ylabel('{}_{}'.format(alg,j+1))
plt.xlim(xmin-(xmax-xmin)/20, xmax+(xmax-xmin)/20)
plt.ylim(ymin-(ymax-ymin)/20, ymax+(ymax-ymin)/20)
for p in range(len(df)):
plt.text(x=X_transformed[p,i], y=X_transformed[p,j], s=df.iloc[p,1],
ha='center', va='center', fontsize=10, color=color_y[p])
plt.subplot(1, 2, 2)
plt.title('{}, {}, colored by t'.format(alg,param))
plt.xlabel('{}_{}'.format(alg,i+1))
plt.ylabel('{}_{}'.format(alg,j+1))
plt.xlim(xmin-(xmax-xmin)/20, xmax+(xmax-xmin)/20)
plt.ylim(ymin-(ymax-ymin)/20, ymax+(ymax-ymin)/20)
for p in range(len(df)):
plt.text(x=X_transformed[p,i], y=X_transformed[p,j], s=df.iloc[p,1],
ha='center', va='center', fontsize=10, color=color_t[p])
plt.show()












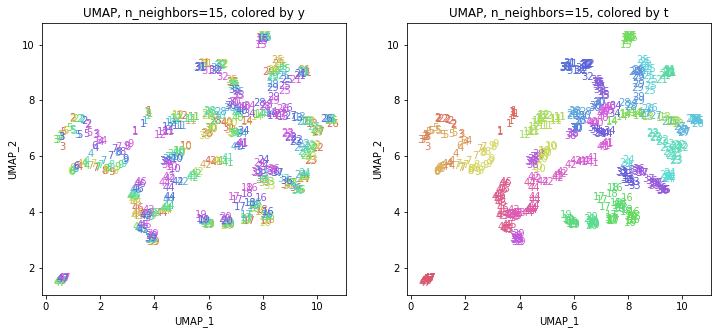
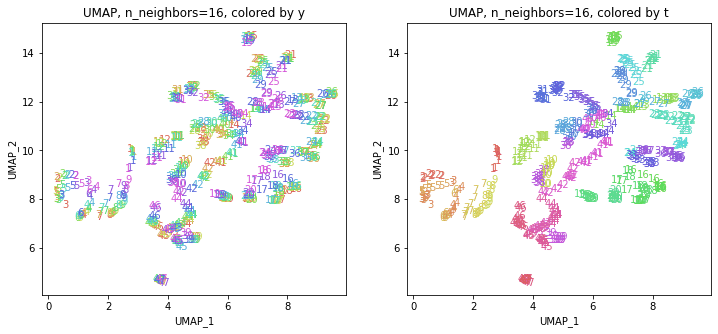







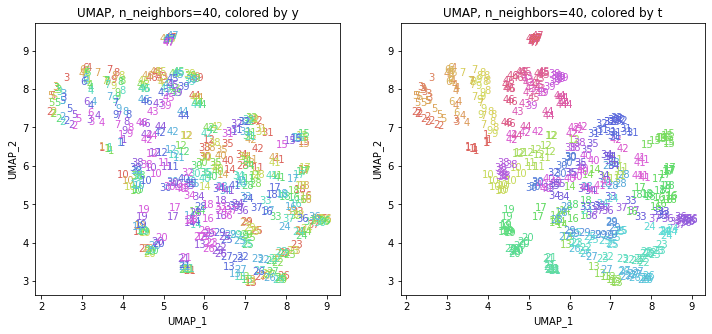











パラメータn_neighborsの値が小さいと、都道府県ごとに分かれていて、パラメータn_neighborsの値を上げていくと、だんだん年度の向きがそろっていくのは、医療費データの場合と同様でした。ただ、医療費データの場合のように完全には向きがそろいませんでした。
また、47沖縄は他の都道府県と離れています。
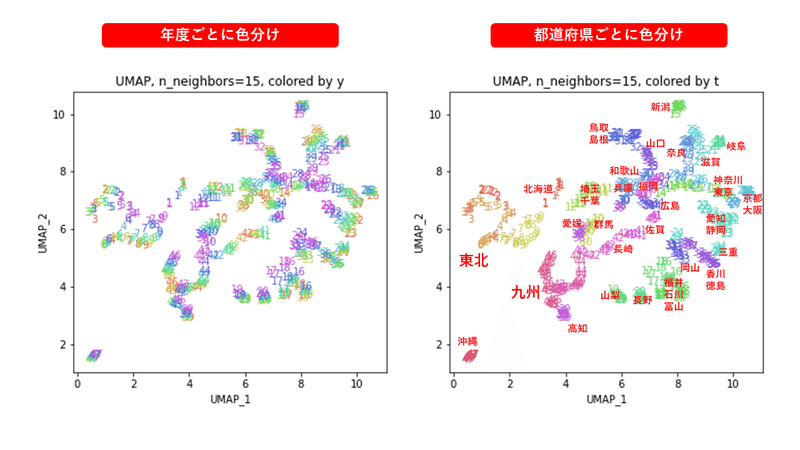


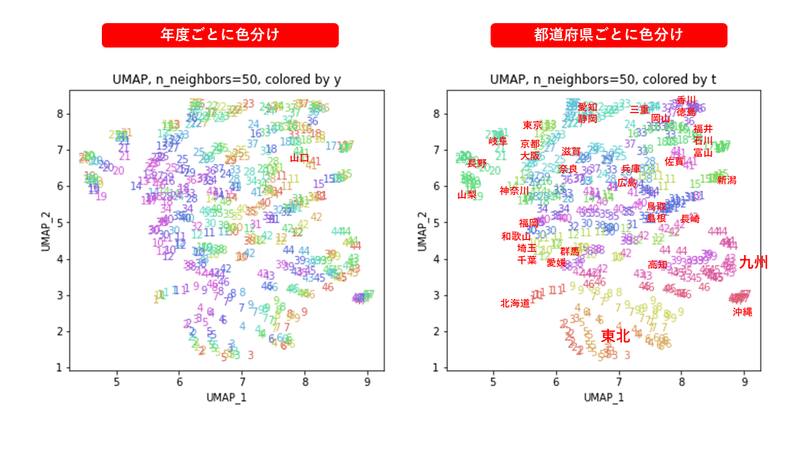
4.PCA & UMAP
PCAの結果、累積寄与率は第8主成分までで8割を超えていましたので、PC1~PC8までを使ってUMAPを実行してみます。
# PCA & UMAP
# PCA
from sklearn.decomposition import PCA
pca = PCA(n_components=8)
PC = pca.fit_transform(X)
print(PC.shape) # (470, 240)
# UMAP
import umap.umap_ as umap
list_n_neighbors = [5,10,15,20,25,30,35,40,45,50,60,70,80,90,100,150,200,300,400]
for n_neighbors in list_n_neighbors:
reducer = umap.UMAP(n_components=2, n_neighbors=n_neighbors, random_state=0)
X_transformed = reducer.fit_transform(PC)
print(X_transformed.shape) # (470, 2)
alg = 'PCA & UMAP' # algorithm
param = 'n_neighbors='+str(n_neighbors) # parameter
i = 0
j = 1
print('{}, {}'.format(alg,param))
plt.figure(figsize=(12,5))
plt.subplot(1, 2, 1)
plt.title('{}, {}, colored by y'.format(alg,param))
plt.xlabel('{}_{}'.format(alg,i+1))
plt.ylabel('{}_{}'.format(alg,j+1))
plt.scatter(x=X_transformed[:,i], y=X_transformed[:,j], c=color_y)
plt.subplot(1, 2, 2)
plt.title('{}, {}, colored by t'.format(alg,param))
plt.xlabel('{}_{}'.format(alg,i+1))
plt.ylabel('{}_{}'.format(alg,j+1))
plt.scatter(x=X_transformed[:,i], y=X_transformed[:,j], c=color_t)
plt.show()
print('{}, {}'.format(alg,param))
xmin, xmax = min(X_transformed[:,i]), max(X_transformed[:,i])
ymin, ymax = min(X_transformed[:,j]), max(X_transformed[:,j])
plt.figure(figsize=(12,5))
plt.subplot(1, 2, 1)
plt.title('{}, {}, colored by y'.format(alg,param))
plt.xlabel('{}_{}'.format(alg,i+1))
plt.ylabel('{}_{}'.format(alg,j+1))
plt.xlim(xmin-(xmax-xmin)/20, xmax+(xmax-xmin)/20)
plt.ylim(ymin-(ymax-ymin)/20, ymax+(ymax-ymin)/20)
for p in range(len(df)):
plt.text(x=X_transformed[p,i], y=X_transformed[p,j], s=df.iloc[p,1],
ha='center', va='center', fontsize=10, color=color_y[p])
plt.subplot(1, 2, 2)
plt.title('{}, {}, colored by t'.format(alg,param))
plt.xlabel('{}_{}'.format(alg,i+1))
plt.ylabel('{}_{}'.format(alg,j+1))
plt.xlim(xmin-(xmax-xmin)/20, xmax+(xmax-xmin)/20)
plt.ylim(ymin-(ymax-ymin)/20, ymax+(ymax-ymin)/20)
for p in range(len(df)):
plt.text(x=X_transformed[p,i], y=X_transformed[p,j], s=df.iloc[p,1],
ha='center', va='center', fontsize=10, color=color_t[p])
plt.show()


















元データからUMAPを実行した結果と似たような結果が得られました。
おわりに
今回は、医療費データと同様に、次元削減のいろいろな手法を使って、健診データを240次元から2次元に圧縮してみました。医療費データの場合ほどきれいな結果ではないものの、地域的な状況をある程度表現できているような結果が得られました。
最後まで読んでいただき、ありがとうございました。
お気づきの点等ありましたら、コメントいただけますと幸いです。
#地域差 , #地域間格差 , #都道府県 , #健診 , #健康診断 , #健診項目 , #健診結果 , #健診データ , #生活習慣病予防健診 , #腹囲 , #BMI , #血圧 , #コレステロール , #中性脂肪 , #血糖 , #尿酸 , #肝機能 , #腎機能 , #クレアチニン , #eGFR , #PCA , #MDS , #tSNE , #UMAP , #次元削減 , #次元圧縮 , #機械学習 , #Python , #協会けんぽ , #noteで数式
この記事が気に入ったらサポートをしてみませんか?
