
実験!岩波データサイエンス1のベイズモデリングをPyMC Ver.5で⑥階層ベイズモデル
「階層ベイズ最初の一歩」章
テキスト「階層ベイズ最初の一歩」の執筆者
久保拓弥 先生
この記事は、テキストの「階層ベイズ最初の一歩」章のモデル2「階層ベイズモデル」の実践を取り扱います。
この章は、給食の変更と児童の身長の関係のベイズ分析を紹介しています。
今回の記事では、前回の「不備なモデル」を改善する「階層ベイズモデル」を取り扱います。
今回のモデルも「給食の変更が児童の身長の伸びに影響する」と推論してしまうのでしょうか?
さあ、光り輝くベイズモデリングの世界へ飛び出しましょう!
前回記事のリンクはこちら。
はじめに
岩波データサイエンスVol.1の紹介
この記事は書籍「岩波データサイエンス vol.1」(岩波書店、以下「テキスト」と呼びます)の特集記事「ベイズ推論とMCMCのフリーソフト」のベイズモデルを用いて、PyMC Ver.5で「実験的」に実装する様子を描いた統計ドキュメンタリーです。
テキストは、2015年10月に発売され、ベイズモデリングの様々なソフトウェアを用いたモデリング事例を多数掲載し、ベイズモデリングの楽しさを紹介する素晴らしい書籍です。
入門的なモデルから2次階差を取り扱う空間モデルまで、幅広い難易度のモデルを満喫できます!
このシリーズは、テキストのベイズモデルをPyMC Ver.5に書き換えて実践します。
引用表記
この記事は、出典に記載の書籍に掲載された文章及びコードを引用し、適宜、掲載文章とコードを改変して書いています。
【出典】
「岩波データサイエンス vol.1」
第9刷、編者 岩波データサイエンス刊行委員会 岩波書店
記事中のイラストは、「かわいいフリー素材集いらすとや」さんのイラストをお借りしています。
ありがとうございます!
PyMC環境の準備
Anacondaを用いる環境構築とGoogle ColaboratoryでPyMCを動かす方法について、次の記事にまとめています。
「PyMCを動かすまでの準備」章をご覧ください。
PythonとPyMC
テキストで利用するツールは、R、BUGS、JAGS です。
この記事では、PythonとPyMCを用いたコードに変換してベイズモデリングを実践いたします。
モデル2 階層ベイズモデル
1. 分析シナリオ(前回の振り返り)
「給食の種類の変更が児童の身長に影響を与えるか」を分析します。
テキストは10県の男子小学生(12歳)平均身長を2回測定した架空データを用いています
1回目は全学校が同じ給食タイプ、1年後の2回目はランダムに選ばれた5県が給食タイプを変更し、残りの5県が1回目と同じ給食タイプを継続します。
架空データは「給食タイプの変更は身長の増減に影響がない」ように生成されたのですが、直線あてはめモデルでは、給食タイプの変更で身長の伸びが小さくなったという誤った推論をしてしまいました。
今回は、ある変数を追加して、ベイズモデルを改善します。
2. 準備(前回と同じ処理です)
「階層ベイズ最初の一歩」章で利用するパッケージをインポートします。
### インポート
# ユーティリティ
import pickle
# 数値・確率計算
import pandas as pd
import numpy as np
# 確率統計
import statsmodels.formula.api as smf
import scipy.stats as stats
# PyMC
import pymc as pm
import arviz as az
# 描画
import matplotlib.pyplot as plt
import seaborn as sns
plt.rcParams['font.family'] = 'Meiryo'
# ワーニング表示の抑制
import warnings
warnings.simplefilter('ignore')3. データの登録と前処理(前回と同じ処理です)
テキストに「岩波データサイエンスのサポートページから例題のデータを格納した,dという名前のdata.frameをダウンロードしてみましょう」と案内があります。
ただ、私には上記データを見つけることができませんでした。
そこで、テキストの表1のデータを引用させていただきます。
### データの登録 ※テキストの表1を引用しました
data_orgn = pd.DataFrame({
'調査県': ['A', 'B', 'C', 'D', 'E', 'F', 'G', 'H', 'I', 'J'],
'給食タイプ': ['乙', '乙', '甲', '乙', '乙', '甲', '甲', '甲', '乙', '甲'],
'標本サイズ1': [55, 53, 55, 53, 58, 55, 58, 59, 56, 56],
'標本サイズ2': [51, 49, 53, 52, 55, 53, 53, 57, 51, 50],
'平均身長1': [151.36, 151.56, 152.22, 153.09, 153.22, 153.31, 152.98,
153.27, 152.67, 155.37],
'平均身長2': [157.27, 156.83, 157.08, 156.00, 157.24, 157.22, 157.81,
158.95, 156.82, 161.71],
'標準偏差1': [2.94, 3.07, 3.2, 2.65, 3.07, 3.1, 2.49, 3.08, 2.82, 3.1],
'標準偏差2': [2.98, 3.14, 3.21, 2.64, 3.03, 3.13, 2.45, 3.06, 2.92, 3.21],
})
display(data_orgn)
# 必要に応じてcsvファイルに保存
# data_orgn.to_csv('./data/kyushoku.csv')【実行結果】
10県のデータです。
給食タイプ甲が1回目の給食と同じ県、乙が2回目に給食の変更があった県です。
標本サイズ・平均身長・標準偏差の末尾1が1回目の測定、末尾2が2回目の測定です。
データの前処理をします。
1回目・2回目の値を縦持ちに変換します。
### データの前処理 1回目・2回目の横持ちから縦持ちへ変換
# コピーの取得
tmp = data_orgn.copy()
# 一部の列名を変更
tmp.rename({'調査県': 'pref', '給食タイプ': 'X'}, axis=1, inplace=True)
# 給食タイプを0:甲、1:乙に変換
tmp['X'] = tmp['X'].apply(lambda x: int(0) if x=='甲' else int(1))
# 標本サイズ、平均身長、標準偏差について、縦持ち変換&値を01の二値変換
tmp1 = pd.melt(tmp, id_vars=['pref', 'X'],
value_vars=['標本サイズ1', '標本サイズ2'],
var_name='Age', value_name='N')
tmp1['Age'] = tmp1['Age'].apply(lambda x: int(0) if x=='標本サイズ1' else int(1))
tmp2 = pd.melt(tmp, id_vars=['pref', 'X'],
value_vars=['平均身長1', '平均身長2'],
var_name='Age', value_name='mean_Y')
tmp3 = pd.melt(tmp, id_vars=['pref', 'X'],
value_vars=['標準偏差1', '標準偏差2'],
var_name='Age', value_name='sd_Y')
# 3つのtmpを結合
data = pd.concat([tmp1, tmp2['mean_Y'], tmp3['sd_Y']], axis=1)
# 列順の変更
data = data[['pref', 'N', 'mean_Y', 'sd_Y', 'Age', 'X']]
display(data)【実行結果】
テキスト23ページの「表1を並びかえたもの」が出来上がりました。
prefは調査県、Nは標本サイズ、mean_Yは平均身長、sd_Yは標準偏差、Ageは測定時期(0は最初の測定、1は1年後:2回目の測定)、Xは給食の変更有無(0:変更なし甲、1:変更あり乙)です。
では、モデル2のPyMCモデリングに進みます。
階層ベイズモデル
1. モデル数式
テキストの数式と事前分布を参照してPyMC的に数式化しました。
$${se}$$は標準誤差であり、観測データdataの標準偏差を観測データの標本サイズの平方根で割り算した値です。
$$
\begin{align*}
\beta_1,\ \beta_2,\ \beta_3 &\sim \text{Uniform}\ (\text{lower}=-10000,\ \text{upper}=10000) \\
\sigma_i &\sim \text{Uniform}\ (\text{lower}=0,\ \text{upper}=10000,\ \text{dims}=Age) \\
r &= \text{Normal}\ (\text{mu}=0,\ \text{sigma}=\sigma_i,\ \text{dims}=(pref,\ Age)) \\
\mu &= \beta_1 + r_{1,j} + (\beta_2 + \beta_3 \times X + r_{2,j}) \times Age \\
y &\sim \text{Normal}\ (\text{mu}=\mu,\ \text{sigma}=se)
\end{align*}
$$
この階層ベイズモデルのポイントは$${r}$$です。
テキストの数式では「県ごとの身長の差$${r_{i,j}}$$」で表されています。
$${1}$$回目の県$${j}$$の$${r{1,j}}$$は県ごとの身長の差、$${2}$$回目の県$${j}$$の$${r{2,j}}$$は県ごとの身長の伸びの差です。
$${r_{i,j}}$$は平均0、標準偏差$${\sigma_i}$$の正規分布に従うと仮定しています。
$$
r_{i,j} \sim \text{Normal}\ (0,\ \sigma_i^2)
$$
標本偏差$${\sigma_i}$$は無情報事前分布に相当する一様分布に従うと仮定しています。
$$
\sigma_i \sim \text{Uniform}\ (0,\ 10000)
$$
階層ベイズモデルの階層は、この$${r_{i,j}}$$の事前分布のパラメータ$${\sigma_i}$$にさらに事前分布が設定されているといった、事前分布が階層構造で複数設定されていることを指すそうです。
そして、階層事前分布は$${r_{i,j}}$$に対する「縛り」として働きます。
個別の「県」に対応する個々の$${r_{i,j}}$$は、当該県だけ、データの一部だけを説明するので「局所パラメータ」と呼ばれます。反対に全データのかなり多くの部分を説明するパラメータは「大域パラメータ」と呼ばれます。
局所パラメータを階層事前分布で縛ることは、事後分布からのサンプリングの収束につながるそうです。
テキストの最終段階において、「統計モデル作りでは、おそらく多くの場合、このような大域・局所パラメーターといった区分が重要であり、それにあわせて適切な事前分布を指定することが、設計のかなめになるのではないでしょうか」と纏められています!
さてさて、県別の身長差$${r_{i,j}}$$を追加したことで、給食タイプの変更が身長の伸びに影響するかどうかを示すパラメータ$${\beta_3}$$はどうなるのでしょう?
前回の不備なモデルでは、パラメータ$${\beta_3}$$の事後平均値が$${-1.74}$$となり、給食タイプの変更が身長の伸びにマイナスの影響があるという、誤った結果を生み出しました。
階層モデルでは、パラメータ$${\beta_3}$$がゼロになること、つまり、給食タイプの変更が身長の伸びに影響しない、という結果が期待されます!
2. モデルの定義
数式モデルどおり実直にPyMCのモデルを定義しましょう。
### モデルの定義
## データの前処理
# 県の値をindex化
pref_idx, pref_label = pd.factorize(data['pref'], sort=True)
# 標準誤差seの算出
se = data['sd_Y'] / np.sqrt(data['N'])
## モデルの定義
with pm.Model() as model2:
### データ関連定義
## coordの定義
model2.add_coord('data', values=data.index, mutable=True)
model2.add_coord('params', values=range(1, 4), mutable=True)
model2.add_coord('pref', values=pref_label, mutable=True)
model2.add_coord('age', values=('1回目', '2回目'), mutable=True)
## dataの定義
# 目的変数:身長の平均値Y
Y = pm.ConstantData('Y', value=data['mean_Y'].values, dims='data')
# 説明変数:給食タイプX 0:甲(変更なし), 1:乙(変更あり)
X = pm.ConstantData('X', value=data['X'].values, dims='data')
# 説明変数:測定回Age 0:1回目, 1:2回目
Age = pm.ConstantData('Age', value=data['Age'].values, dims='data')
# 標準誤差se 尤度関数の正規分布の標準偏差に利用
se = pm.ConstantData('se', value=se, dims='data')
# 県インデックス
prefIdx = pm.ConstantData('prefIdx', value=pref_idx, dims='data')
### 事前分布
## 線形モデル
# 偏回帰係数β
beta = pm.Uniform('beta', lower=-10000, upper=10000, dims='params')
# 県ごとの身長差r
sigmaI = pm.Uniform('sigmaI', lower=0, upper=10000, dims='age')
r = pm.Normal('r', mu=0, sigma=sigmaI, dims=('pref', 'age'))
### 線形予測子
mu = pm.Deterministic(
'mu',
beta[0] + r[prefIdx, 0] + (beta[1] + beta[2]*X + r[prefIdx, 1]) * Age,
dims='data')
### 尤度
y = pm.Normal('y', mu=mu, sigma=se, observed=Y, dims='data')モデルの内容を表示・可視化してみましょう。
### モデルの表示
model2【実行結果】

続いてモデルを可視化します。
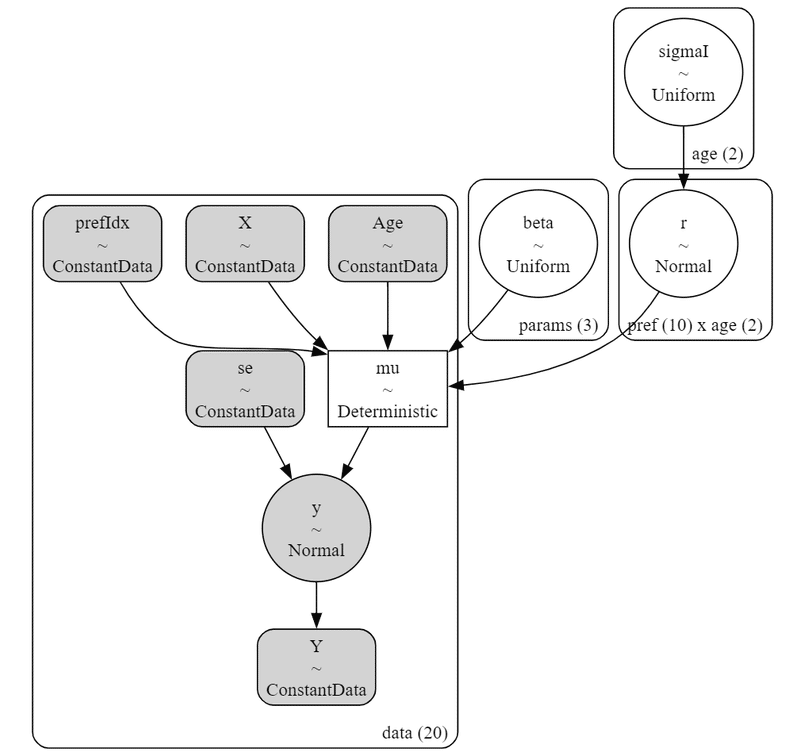
### モデルの可視化
pm.model_to_graphviz(model2)【実行結果】
データやパラメータの繋がりが示されます。
丸い形状の変数には「確率分布」が併記されます。
データ値を示す変数はグレー色が付いています。
白丸のsigmaI の事前分布(一様分布)と r の事前分布(正規分布)が、階層的に連なっている様子がよく分かります。
3. MCMCの実行と収束の確認
■ MCMC
いよいよベイズプログラミングの真骨頂 MCMC の実行です。
マルコフ連鎖=chainsを4本、バーンイン期間=tuneを1000、利用するサンプル=drawを1chainあたり1000に設定して、合計4000個の事後分布からのサンプリングデータを生成します。テキストの指定数とは異なっています。
サンプリングデータは idata2 に格納されます。
サンプル方法に numpyro を指定しています。処理速度が早くなります。
numpyro を使わない場合は「nuts_sampler='numpyro',」を削除します。
### 事後分布からのサンプリング 25秒
with model2:
idata2 = pm.sample(draws=1000, tune=1000, chains=4, random_seed=123,
nuts_sampler='numpyro', target_accept=0.8)【実行結果】
処理時間は25秒です。
■ $${\hat{R}}$$で収束確認
収束の確認をします。
指標$${\hat{R}}$$(あーるはっと)の値が$${1.1}$$以下のとき、収束したこととします。
次のコードでは$${\hat{R}>1.01}$$のパラメータの個数をカウントします。
個数が0ならば、$${\hat{R} \leq1.1}$$なので収束したとみなします。
### r_hat>1.1の確認
# 設定
idata_in = idata2 # idata名
threshold = 1.01 # しきい値
# しきい値を超えるR_hatの個数を表示
print((az.rhat(idata_in) > threshold).sum())【実行結果】
$${\hat{R}>1.01}$$のパラメータは0個でした。
$${\hat{R} \leq1.1}$$なので収束したとみなします。

■ $${\hat{R}}$$の値把握と事後統計量の表示
### 事後統計量の表示
pm.summary(idata2, hdi_prob=0.95)【実行結果】
MCMCでサンプリングデータを生成したすべてのパラメータについて、統計量や診断情報が一覧表示されました。
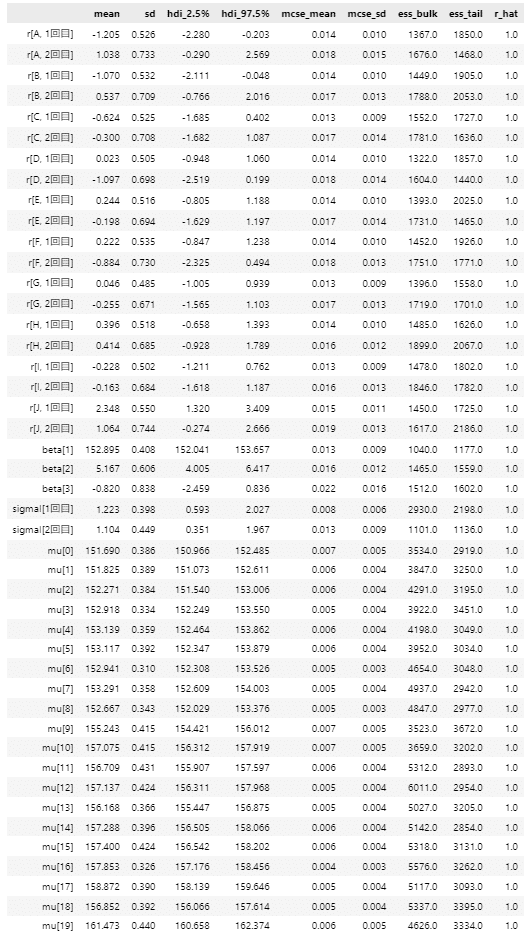
係数$${\beta}$$の平均値なども確認できます。
注目の$${\beta_3}$$は平均値$${-0.820}$$、95%HDI$${[-2.459, 0.836]}$$です。
前のモデルと違って、95%HDIが0をまたいで負の値・正の値の両方を取りうる様子がわかります。
■ トレースプロットで収束確認
トレースプロットを描画します。
こちらの図でも収束の確認を行えます。
### トレースプロットの描画
pm.plot_trace(idata2, compact=True)
plt.tight_layout();【実行結果】
左の曲線は、4本のマルコフ連鎖の分布です。
4本の曲線がほぼ同じ分布を描いているので、サンプリングデータが同一の分布から生成されたことが推察できます。
右のノイズみたいな図は、均等に乱雑に描画されています。
この状態は収束していると推察できます。
均等・乱雑ではなくて、何らかの傾向を示すような描画の場合は収束を疑います。
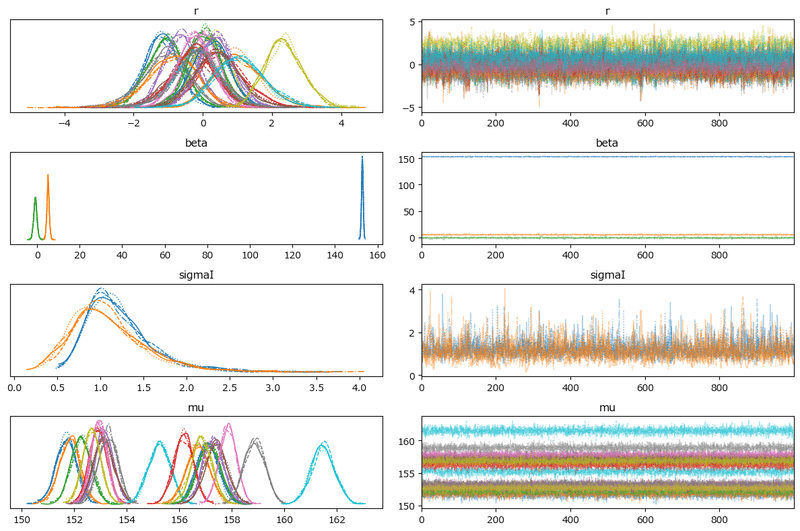
$${\hat{R}}$$とトレースプロットから、分布の収束が確認できましたので、MCMCのサンプリングデータを用いて、推論を継続できます!
やったね!
4. 分析
$${\beta_3}$$の事後分布を描画します。
2つのモデルの推定値を描画します。
テキストの図8に相当します。
前回の直線的あてはめモデルの悪さを再確認しつつ、今回の階層ベイズモデルによる$${\beta_3}$$の推論の適否を確認しましょう。
### β3の事後分布プロットを描画 ※図8に相当
pm.plot_posterior(idata1, hdi_prob=0.95, var_names=['beta'], round_to=3,
coords={'params': [3]}, ref_val=0, figsize=(5, 3))
pm.plot_posterior(idata2, hdi_prob=0.95, var_names=['beta'], round_to=3,
coords={'params': [3]}, ref_val=0, figsize=(5, 3))
plt.tight_layout();【実行結果】
上が前回モデル、下が階層ベイズモデルです。
階層ベイズモデルでは$${\beta_3>0}$$となる確率は$${14.7%}$$になりました。
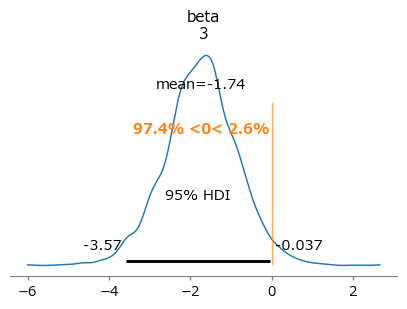
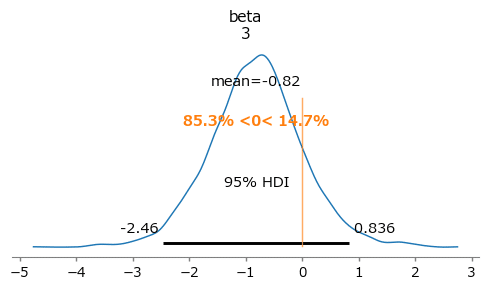
テキストを真似しますと、階層ベイズモデルの「$${\beta_3}$$の事後分布は95%HDIに0を含んでいるので、$${\beta_3}$$は0からそんなに離れているとは言えないね」です。
$${\beta_3}$$の値が0に近いので、給食タイプの変更が身長の伸びに影響を与えていないだろう、と言えそうです。
分析を続けます。
県ごとの身長差$${r_{i,j}}$$を可視化しましょう。
テキストの図9に相当します。
### 県ごとの身長差rの描画 ※図9に相当
## データの準備
# r_1, r_2の取り出し
r_1 = idata2.posterior.r.stack(sample=('chain', 'draw'))[:, 0].data
r_2 = idata2.posterior.r.stack(sample=('chain', 'draw'))[:, 1].data
## σ_iの中央値を標準偏差としたときのr_1, r_2の事前分布の算出
# σ_iの中央値の算出
sigma_i_median1 = (idata2.posterior.sigmaI.stack(sample=('chain', 'draw'))[0]
.median().data)
sigma_i_median2 = (idata2.posterior.sigmaI.stack(sample=('chain', 'draw'))[1]
.median().data)
# 正規分布の確率密度関数の算出
x_val = np.linspace(min(r_1.min(), r_2.min()), max(r_1.max(), r_2.max()), 1001)
y_val1 = stats.norm.pdf(x_val, loc=0, scale=sigma_i_median1)
y_val2 = stats.norm.pdf(x_val, loc=0, scale=sigma_i_median2)
## 描画
fig, (ax1, ax2) = plt.subplots(2, 1, figsize=(8, 7), sharex=True, sharey=True)
# 県の数だけ描画を繰り返し処理
for j, pref in enumerate(pref_label):
# 1回目r_1のKDE曲線を描画
sns.kdeplot(r_1[j], ax=ax1)
ax1.set(title='県ごとの身長差: 1回目 $r_1$')
# 2回目r_2のKDE曲線を描画
sns.kdeplot(r_2[j], ax=ax2)
ax2.set(title='県ごとの身長の伸びの差: 2回目 $r_2$')
# σ_iの中央値を標準偏差とするr_1,r_2の正規分布の確率密度関数の描画
ax1.fill_between(x_val, 0, y_val1, color='lightpink', alpha=0.5, label=None)
ax2.fill_between(x_val, 0, y_val2, color='lightpink', alpha=0.5)
# x=0の垂直線の描画
ax1.axvline(0, color='red', ls='--')
ax2.axvline(0, color='red', ls='--')
# グリッド表示
ax1.grid(lw=0.5)
ax2.grid(lw=0.5)
# x軸の範囲
ax1.set_xlim(-3, 3)
# 凡例
ax1.legend(title='県', labels=pref_label, bbox_to_anchor=(1.04, 0.15))
plt.tight_layout()
plt.show()【実行結果】
県というグループ(質的変数の要素)ごとに事後分布を得られることはベイズプログラミングの魅力ですね!
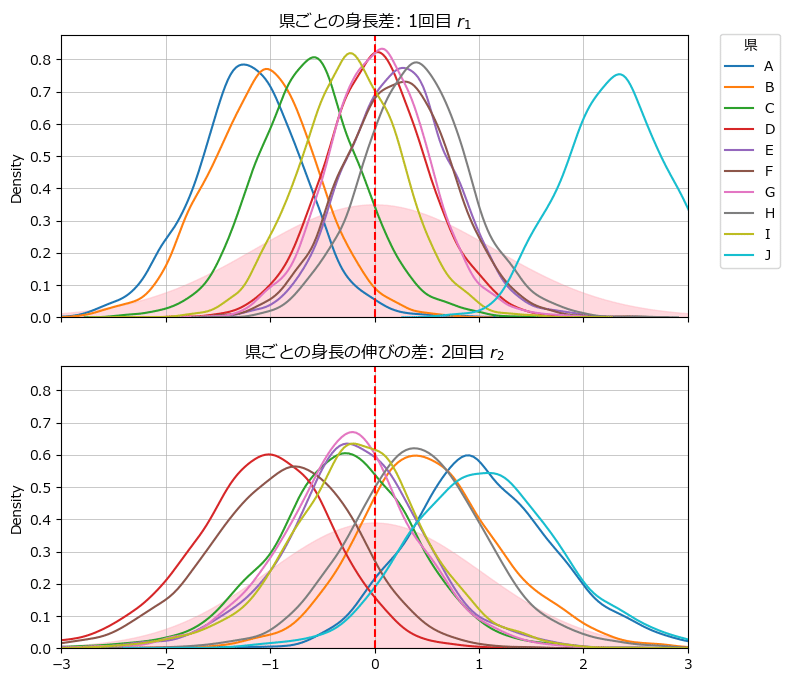
5. 推論データの保存
pickle で MCMCサンプリングデータ idata2 を保存できます。
### 推論データの保存
file = r'idata2_ch2.pkl'
with open(file, 'wb') as f:
pickle.dump(idata2, f)次のコードで保存したファイルを読み込みできます。
### 推論データの読み込み
file = r'idata2_ch2.pkl'
with open(file, 'rb') as f:
idata2_ch2_load = pickle.load(f)ベイズ記事は以上です。
次回予告
「時系列・空間データのモデリング」章
状態空間モデル「スギの年輪幅データ」
結び
階層事前分布で変数を縛る(自由にさせない)、という格言を頂きました。
カテゴリ変数の要素別効果をモデルに導入するとパラメータ数がグンと増える事態になりますが、この格言を思い出しながら、よりよいモデル構築ができるように励みたいです。
おわり
シリーズの記事
次の記事
前の記事
目次
ブログの紹介
note で7つのシリーズ記事を書いています。
ぜひ覗いていってくださいね!
1.のんびり統計
統計検定2級の問題集を手がかりにして、確率・統計をざっくり掘り下げるブログです。
雑談感覚で大丈夫です。ぜひ覗いていってくださいね。
統計検定2級公式問題集CBT対応版に対応しています。
Python、EXCELのサンプルコードの配布もあります。
2.実験!たのしいベイズモデリング1&2をPyMC Ver.5で
書籍「たのしいベイズモデリング」・「たのしいベイズモデリング2」の心理学研究に用いられたベイズモデルを PyMC Ver.5で描いて分析します。
この書籍をはじめ、多くのベイズモデルは R言語+Stanで書かれています。
PyMCの可能性を探り出し、手軽にベイズモデリングを実践できるように努めます。
身近なテーマ、イメージしやすいテーマですので、ぜひぜひPyMCで動かして、一緒に楽しみましょう!
3.実験!岩波データサイエンス1のベイズモデリングをPyMC Ver.5で
書籍「実験!岩波データサイエンスvol.1」の4人のベイジアンによるベイズモデルを PyMC Ver.5で描いて分析します。
この書籍はベイズプログラミングのイロハをざっくりと学ぶことができる良書です。
楽しくPyMCモデルを動かして、ベイズと仲良しになれた気がします。
みなさんもぜひぜひPyMCで動かして、一緒に遊んで学びましょう!
4.楽しい写経 ベイズ・Python等
ベイズ、Python、その他の「書籍の写経活動」の成果をブログにします。
主にPythonへの翻訳に取り組んでいます。
写経に取り組むお仲間さんのサンプルコードになれば幸いです🍀
5.RとStanではじめる心理学のための時系列分析入門 を PythonとPyMC Ver.5 で
書籍「RとStanではじめる心理学のための時系列分析入門」の時系列分析をPythonとPyMC Ver.5 で実践します。
この書籍には時系列分析のテーマが盛りだくさん!
時系列分析の懐の深さを実感いたしました。
大好きなPythonで楽しく時系列分析を学びます。
6.データサイエンスっぽいことを綴る
統計、データ分析、AI、機械学習、Pythonのコラムを不定期に綴っています。
統計・データサイエンス書籍にまつわる記事が多いです。
「統計」「Python」「数学とPython」「R」のシリーズが生まれています。
7.Python機械学習プログラミング実践記
書籍「Python機械学習プログラミング PyTorch & scikit-learn編」を学んだときのさまざまな思いを記事にしました。
この書籍は、scikit-learnとPyTorchの教科書です。
よかったらぜひ、お試しくださいませ。
最後までお読みいただきまして、ありがとうございました。
この記事が気に入ったらサポートをしてみませんか?
