brmsパッケージで共分散分析をベイズ推定してみた。
library(brms)
library(rstan)用量doseを因子型に変換します。
data=ToothGrowth
data=transform(data,dose=as.factor(dose))ベイズの線形回帰パッケージbrmsを使用します。
交互作用を考えるモデルを組みます。
brms.fit=brm(formula= len ~ supp*dose,
family=gaussian(),
data=data)Population-Level Effects:
Estimate Est.Error l-95% CI u-95% CI Rhat Bulk_ESS Tail_ESS
Intercept 13.23 1.18 10.92 15.51 1.00 2269 2697
suppVC -5.25 1.68 -8.59 -1.86 1.00 2184 2572
dose1 9.46 1.67 6.23 12.75 1.00 2184 2863
dose2 12.87 1.65 9.50 16.00 1.00 2371 2992
suppVC:dose1 -0.66 2.36 -5.30 4.03 1.00 2245 2747
suppVC:dose2 5.30 2.35 0.72 10.01 1.00 2260 2750
suppVC:dose1では交互作用を認めませんが、
suppVC:dose2では交互作用を認めています。
層別解析すべきなので、dose:suppで検討します。
eff=conditional_effects(brms.fit,effects="dose:supp")
plot(eff,points=F)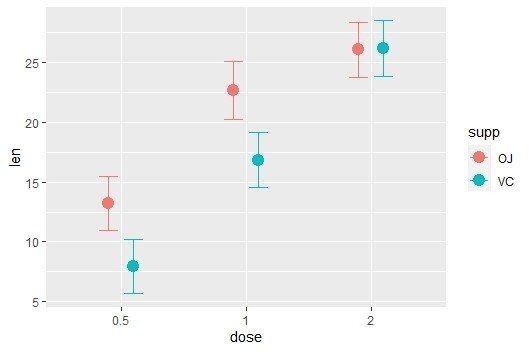
eff0=conditional_effects(brms.fit,effects="supp:dose")
plot(eff0,points=F)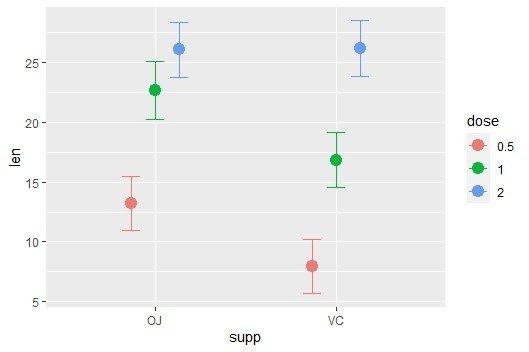
ベイズ推定なので統計学的有意差という表現ではありませんが、
OJ群とVC群でみると、2.0㎎群では差はなく、0.66程度の差でしかない。
また、OJ1.0㎎群とOJ2.0㎎群、VC2.0mg群でも差はありません。
さらにOJ0.5mg群とVC1.0mg群にも差はなさそうです。
この記事が気に入ったらサポートをしてみませんか?
