
【Python】高卒の元シェフによる機械学習を使った毒キノコの判別【機械学習入門】
1.はじめに
自己紹介
こんにちは。
私は飲食業に約10年間従事してきた料理人です。
今回はIT業界への転職を視野に入れAidemyのデータ分析講座を受講し、本項を最終課題として作成させていただきました。
完全初心者の学習成果としてこれから新たに学習する方々への後押しとなれましたら幸いです。
今回の目標
約6万件のデータをもとにキノコの毒の有無を分類する機械学習を実施します。
5種のモデルを使いどのモデルが正確に分類できたかを考察していきます。
2.データについて
データセットと実行環境
以下リンクより取得しました。
今回はこちらのデータデータセットの中の"Secondary data"を使用し分析を行いました。
実行環境はGoogle Colaboratoryです。
データ概要
以下データフォルダ内に格納されていたSecondary dataに対するメタデータです。
1. Title: Secondary mushroom data
2. Sources:
(a) Mushroom species drawn from source book:
Patrick Hardin.Mushrooms & Toadstools.
Zondervan, 1999
(b) Inspired by this mushroom data:
Jeff Schlimmer.Mushroom Data Set. Apr. 1987.
url:https://archive.ics.uci.edu/ml/datasets/Mushroom.
(c) Repository containing the related Python scripts and all the data sets: https://mushroom.mathematik.uni-marburg.de/files/
(d) Author: Dennis Wagner
(e) Date: 05 September 2020
3. Relevant information:
This dataset includes 61069 hypothetical mushrooms with caps based on 173 species (353 mushrooms
per species). Each mushroom is identified as definitely edible, definitely poisonous, or of
unknown edibility and not recommended (the latter class was combined with the poisonous class).
Of the 20 variables, 17 are nominal and 3 are metrical.
4. Data simulation:
The related Python project (Sources (c)) contains a Python module secondary_data_generation.py
used to generate this data based on primary_data_edited.csv also found in the repository.
Both nominal and metrical variables are a result of randomization.
The simulated and ordered by species version is found in secondary_data_generated.csv.
The randomly shuffled version is found in secondary_data_shuffled.csv.
5. Class information:
1. class poisonous=p, edibile=e (binary)
6. Variable Information:
(n: nominal, m: metrical; nominal values as sets of values)
1. cap-diameter (m): float number in cm
2. cap-shape (n): bell=b, conical=c, convex=x, flat=f,
sunken=s, spherical=p, others=o
3. cap-surface (n): fibrous=i, grooves=g, scaly=y, smooth=s,
shiny=h, leathery=l, silky=k, sticky=t,
wrinkled=w, fleshy=e
4. cap-color (n): brown=n, buff=b, gray=g, green=r, pink=p,
purple=u, red=e, white=w, yellow=y, blue=l,
orange=o, black=k
5. does-bruise-bleed (n): bruises-or-bleeding=t,no=f
6. gill-attachment (n): adnate=a, adnexed=x, decurrent=d, free=e,
sinuate=s, pores=p, none=f, unknown=?
7. gill-spacing (n): close=c, distant=d, none=f
8. gill-color (n): see cap-color + none=f
9. stem-height (m): float number in cm
10. stem-width (m): float number in mm
11. stem-root (n): bulbous=b, swollen=s, club=c, cup=u, equal=e,
rhizomorphs=z, rooted=r
12. stem-surface (n): see cap-surface + none=f
13. stem-color (n): see cap-color + none=f
14. veil-type (n): partial=p, universal=u
15. veil-color (n): see cap-color + none=f
16. has-ring (n): ring=t, none=f
17. ring-type (n): cobwebby=c, evanescent=e, flaring=r, grooved=g,
large=l, pendant=p, sheathing=s, zone=z, scaly=y, movable=m, none=f, unknown=?
18. spore-print-color (n): see cap color
19. habitat (n): grasses=g, leaves=l, meadows=m, paths=p, heaths=h,
urban=u, waste=w, woods=d
20. season (n): spring=s, summer=u, autumn=a, winter=w
以下要約
データのソース:書籍と先行研究から生成された仮想キノコデータ
キノコは、食用、毒キノコ、および食用不可の3つのクラスに分類される
データセットには20の変数があり、うち17は質的変数、3は量的変数である
クラス情報は、毒キノコか否かを表すpまたはeの2値データ
20の変数には、キノコのキャップの直径、形状、表面、色、さけやすさ、ひだのつき具合、ひだの間隔、柄の高さ、幅、根の形状、表面、色、蓋の種類、蓋の色、リングの有無、リングの種類、胞子の印刷色、生息地、季節が含まれる
データの読み込みと確認
今回はドライブに保存しドライブにマウントさせファイルを読み込みました。カラム情報が一括でまとめられていたのでsep=';'を引数に渡し分割しています。
#ファイルの読み込み
from google.colab import drive
drive.mount('/content/drive')
df = pd.read_csv("/content/drive/MyDrive/secondary_data.csv",sep=';')
#ライブラリの読み込み
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
import lightgbm as lgbデータを確認してみましょう
#データの確認
print(df.shape)
df.head()
ファイルの確認ができました。数値表記されている列がメタデータで説明のあった量的変数みたいです。
3.実装(前処理)
欠損値の確認と処理
欠損値を処理していきましょう。表示してみます。
#欠損値の表示
print(df.isnull().sum())以下のような結果が出ました
class 0
cap-diameter 0
cap-shape 0
cap-surface 14120
cap-color 0
does-bruise-or-bleed 0
gill-attachment 9884
gill-spacing 25063
gill-color 0
stem-height 0
stem-width 0
stem-root 51538
stem-surface 38124
stem-color 0
veil-type 57892
veil-color 53656
has-ring 0
ring-type 2471
spore-print-color 54715
habitat 0
season 0
dtype: int64まずは欠損値の多い列を削除しました。
#欠損値の多い列の削除
drop=["cap-surface", "gill-attachment", "gill-spacing", "stem-root", "stem-surface" , "veil-type", "veil-color", "spore-print-color"]
df=df.drop(drop, axis = 1)続いて比較的欠損値の少ない列に関しては該当行を削除しました。
#欠損値の比較的少ないring-typeは欠損行を削除
df.dropna(subset=['ring-type'], inplace=True)
print(df.shape)shapeは(58598, 13)となりました。
念の為欠損値の再確認です。
class 0
cap-diameter 0
cap-shape 0
cap-color 0
does-bruise-or-bleed 0
gill-color 0
stem-height 0
stem-width 0
stem-color 0
has-ring 0
ring-type 0
habitat 0
season 0
dtype: int64以上のような結果になりました。大丈夫そうです。
計量尺度への対処
今回の計量尺度はサイズに関する3列でした。まずは列内のデータの詳細を確認しましょう。
今回は箱ひげ図で可視化してみます。
#①cap-diameter
print(df["cap-diameter"].describe())
#箱ひげで表示
plt.boxplot(df["cap-diameter"])
plt.show()
#②stem-height
print(df["stem-height"].describe())
#箱ひげで表示
plt.boxplot(df["stem-height"])
plt.show()
#③stem-width
print(df["stem-height"].describe())
#箱ひげで表示
plt.boxplot(df["stem-width"])
plt.show()


どの要素も上位25%がかなり大きく幅があります。
今回はこのような外れ値が大きく作用しないようそれぞれの列を8つのクラスに分けました。
また、カテゴリを扱いやすい整数クラスにするため引数にlabels=Falseを渡しています。
#要素を8つのカテゴリに分割
df['cap-diameter'] = pd.qcut(df['cap-diameter'], 8, labels=False)
df['stem-height'] = pd.qcut(df['stem-height'], 8, labels=False)
df['stem-width'] = pd.qcut(df['stem-width'], 8, labels=False)グラフで可視化
一旦ここで毒の有無と各カラムの関連性を確認してみましょう。
cols = df_copy.columns
fig, axes = plt.subplots(5, 3, figsize=(50,50))
axes = axes.ravel()
for col, ax in zip(cols, axes):
sns.histplot(data=df_copy, x=col, hue='class', ax=ax, multiple='stack')
plt.show()


このようになりました。一番最初のグラフから有毒の割合が約55%、無毒が約45%で有ることがわかります。
つまりこの割合より差が大きい要素を含むカラムは特徴量の重要性が高く、逆に割合が上記と同様である場合特徴量としての重要性は低いと推測できます。
グラフを確認して重要性の高そうなものとそうでなものを予測してみます。
重要性の高そうなカラム

stem-colorのグラフです。もっともサンプルの多いwの有毒比率がデータ平均より明らかに低いです。これは特徴量の重要性が高そうです。
重要性の低そうなカラム

こちらはdeos-bruise-or-bleedのカラムです。どちらの要素も有毒の割合が若干高く、データの平均と大きな差異はなさそうです。
ではランダムフォレスト(後述)の.feature_importances_の属性を利用して特徴量の重要性を見ていきましょう。
# ランダムフォレストモデルの学習
rfc = RandomForestClassifier()
rfc.fit(X_train, y_train)
#重要な特徴量の可視化
plt.barh(X.columns, rfc.feature_importances_)
このような結果となりました。
上で予想した内容が最も高い(低い)重要性というわけではありませんでしたが大方予想は当たっていたようです。
型の確認と処理
次に全体の型の確認をしてみましょう
df.dtypesclass object
cap-diameter int64
cap-shape object
cap-color object
does-bruise-or-bleed object
gill-color object
stem-height int64
stem-width int64
stem-color object
has-ring object
ring-type object
habitat object
season object
dtype: objectこのような結果になりました。
次は他の列のオブジェクト型の項を整数型に変更していきます。
scikit-learnの LabelEncoderを利用しました。
参考URL
【scikit-learn】カテゴリ変数を数値化するsklearn.preprocessing.LabelEncoder【ラベルエンコーディング】
from sklearn.preprocessing import LabelEncoder
le = LabelEncoder()
# 'object'列を整数エンコーディング
df[["class", 'cap-shape', 'cap-color', "does-bruise-or-bleed", "gill-color", "stem-color", "has-ring", "ring-type", "habitat", "season"]] = df[["class", 'cap-shape', 'cap-color', "does-bruise-or-bleed", "gill-color", "stem-color", "has-ring", "ring-type", "habitat", "season"]].apply(le.fit_transform)
#型の確認
df.dtypesclass int64
cap-diameter int64
cap-shape int64
cap-color int64
does-bruise-or-bleed int64
gill-color int64
stem-height int64
stem-width int64
stem-color int64
has-ring int64
ring-type int64
habitat int64
season int64
dtype: objectこのようにすべてのカラムに対して整数エンコーディングが完了しました。
訓練データとテストデータに分ける
最後にデータを訓練用とテスト用に分けます。
from sklearn.model_selection import train_test_split
#トレーニングデータとテストデータに分割
(X_train, X_test, y_train, y_test) = train_test_split(df.iloc[:,1:], df.iloc[:,0], test_size=0.3, random_state=0)
#データが分割されているか確認
print(X_train.shape,X_test.shape,y_train.shape,y_test.shape)
結果は以下のようになりました。問題なさそうです。

3. 実装(モデルの実施)
モデルについて
それではデータの前処理が完了したので実際にモデルで正答率を求めていきましょう。
今回は以下のモデルで実施致します。
チューニングなしの結果とチューニング後の結果を求めていきます。
ディープラーニングはおまけで実施しました。
ランダムフォレスト
XGBoost
LightGBM
k-NN
ディープラーニング
上から順に実施していきます。
1. ランダムフォレスト
ランダムフォレスト(random forests)は機械学習のアルゴリズムのひとつで、決定木による複数の弱学習器を統合させて汎化能力を向上させる、アンサンブル学習アルゴリズムである。
ランダムフォレストはアンサンブル学習の中でもバギングという手法を使った代表的モデルです。
まずはデフォルトでの結果です。
from sklearn.ensemble import RandomForestRegressor
from sklearn.ensemble import RandomForestClassifier
from sklearn.metrics import accuracy_score
# ランダムフォレストモデルの学習
rfc = RandomForestClassifier()
rfc.fit(X_train, y_train)
#n_estimators=100, random_state=0
# テストデータに対する予測
y_pred = rfc.predict(X_test)
# テストデータに対する正解率の計算
accuracy = accuracy_score(y_test, y_pred)
print('Accuracy:', accuracy)

デフォルトでかなりハイスコアが出ました。
ランダムサーチでベルトパラメーターを検索しましょう。
param_dist = {
'n_estimators': [100, 200],
'max_features': ['auto', 'sqrt'],
'max_depth': [10, 20, None],
'min_samples_split': [2, 5],
'min_samples_leaf': [1, 2],
'bootstrap': [True, False]
}
# モデルのインスタンスを生成
rfc = RandomForestClassifier()
# ランダムサーチで最適なパラメータを探索
rfc_random = RandomizedSearchCV(estimator=rfc, param_distributions=param_dist, n_iter=10, cv=5, random_state=0)
rfc_random.fit(X_train, y_train)
# 最適なパラメータを表示
print(rfc_random.best_params_)
以上の結果が出ました。実際に入力して確認してみます。
from sklearn.ensemble import RandomForestRegressor
from sklearn.ensemble import RandomForestClassifier
from sklearn.metrics import accuracy_score
# ランダムフォレストモデルの学習
rfc = RandomForestClassifier(n_estimators=200, min_samples_split=2, min_samples_leaf=1, max_features="auto", max_depth=None, bootstrap=False, random_state=0)
rfc.fit(X_train, y_train)
#重要な特徴量の可視化
plt.barh(X.columns, rfc.feature_importances_)
#n_estimators=100, random_state=0
# テストデータに対する予測
y_pred = rfc.predict(X_test)
# テストデータに対する正解率の計算
accuracy = accuracy_score(y_test, y_pred)
print('Accuracy:', accuracy)デフォルトでも相当精度が高いことがわかります。
僅かですが、スコアがアップしました。チューニングの効果は得られたようです。
2. XGBoost
続いてXGBoostです
XGBoostとは
XGBoost(eXtreme Gradient Boosting / 勾配ブースティング回帰木)とは、アンサンブル学習の一つで、ブースティングと決定木を組み合わせています。
ブースティングとは、弱いモデル(弱学習器と呼びます)を複数作成し、一つ前の学習器の誤りを次の学習器が修正するという操作を繰り返し行うことで性能を向上させる手法です。
勾配ブースティング回帰木では、浅い決定木を複数作成し、それぞれの決定木はデータの一部に対してしか良い予測を行うことができないため、ブースティングを行うことで性能を向上させています。パラメータ設定に敏感という欠点がありますが、正しく設定すればランダムフォレストよりも良い性能となります。
また、XGBoost(勾配ブースティング回帰木)の名前に「回帰木」とありますが、回帰と分類のどちらでも使用可能です。
とあります。
アンサンブル学習の中でもブースティングという手法に該当します。
ランダムフォレストとの有意な差は出るのでしょうか。
まずはパラメータチューニングなしのコードと結果です。
import xgboost as xgb
from sklearn.metrics import accuracy_score
# データセットをDMatrix形式に変換する
dtrain = xgb.DMatrix(X_train, label=y_train)
dtest = xgb.DMatrix(X_test)
# パラメータを指定してモデルを学習する
param = {}
num_round = 100
bst = xgb.train(param, dtrain, num_round)
# テストデータを予測して精度を評価する
y_pred = bst.predict(dtest)
y_pred = [1 if x > 0.5 else 0 for x in y_pred]
accuracy = accuracy_score(y_test, y_pred)
print("Accuracy:", accuracy)
以上の結果が出ました。なかなかの数値です。
では、パラメーターを検索をしていきましょう。
グリッドサーでは1時間経っても終わらなかったのでランダムサーチで実施しました。(それでも20分くらいはかかりました…)
import xgboost as xgb
from sklearn.model_selection import RandomizedSearchCV
# XGBoostのパラメータ範囲
param_dist = {
'max_depth': [3, 4, 5, 6, 7, 8, 9, 10],
'learning_rate': [0.01, 0.05, 0.1, 0.15, 0.2],
'n_estimators': [100, 200, 300, 400, 500],
'subsample': [0.6, 0.7, 0.8, 0.9, 1.0],
'colsample_bytree': [0.6, 0.7, 0.8, 0.9, 1.0],
'gamma': [0, 1, 2, 3, 4]
}
# XGBoostのインスタンスを生成
xgb = xgb.XGBClassifier()
# ランダムサーチで最適なパラメータを探索
xgb_random = RandomizedSearchCV(estimator=xgb, param_distributions=param_dist, n_iter=10, cv=5, random_state=0)
xgb_random.fit(X_train, y_train)
# 最適なパラメータを表示
print(xgb_random.best_params_)以下のような結果が得られました。早速入力して実行してみます。

import xgboost as xgb
from sklearn.metrics import accuracy_score
# データセットをDMatrix形式に変換する
dtrain = xgb.DMatrix(X_train, label=y_train)
dtest = xgb.DMatrix(X_test)
# パラメータを指定してモデルを学習する
param = {'subsample': 1.0, 'n_estimators': 500, 'max_depth': 10, 'learning_rate': 0.15, 'gamma': 0, 'colsample_bytree': 0.8}
num_round = 100
bst = xgb.train(param, dtrain, num_round)
# テストデータを予測して精度を評価する
y_pred = bst.predict(dtest)
y_pred = [1 if x > 0.5 else 0 for x in y_pred]
accuracy = accuracy_score(y_test, y_pred)
print("Accuracy:", accuracy)
99.37%まで上がりました。結果としてランダムフォレストより高い数字です。
3. LightGBM
続いてLightGBMです。
LightGBMはXGBoostと同じブースティングアルゴリズムを採用しています。
大きな違いはLightGBMはカテゴリ変数の処理方法や特徴量の分割方法を最適化することで高速な学習を実現しており、XGBoostは多くのパラメータを調整することで高速な学習を実現している点です。
まずはデフォルトの結果です。
#LightGBM
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score
# データセットの作成
train_data = lgb.Dataset(X_train, label=y_train)
test_data = lgb.Dataset(X_test, label=y_test)
# パラメータの設定
params = {}
# 学習の実行
num_round = 100
bst = lgb.train(params, train_data, num_round, valid_sets=[test_data])
# 予測の実行
y_pred = bst.predict(X_test)
y_pred_binary = [int(pred >= 0.5) for pred in y_pred]
# 結果の表示
accuracy = accuracy_score(y_test, y_pred_binary)
print('Accuracy: {:.6f}'.format(accuracy))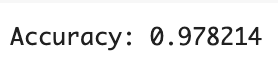
先程の2種よりは低めの数値になります。
適切なパラメータを検索して実行してみます。
#LightGBMのパラメーター調整
import lightgbm as lgb
from sklearn.model_selection import GridSearchCV
params = {'num_leaves': [31, 127],
'max_depth': [4, 8],
'learning_rate': [0.1, 0.05],
'n_estimators': [50, 100]}
lgb_model = lgb.LGBMClassifier()
grid_search = GridSearchCV(lgb_model, param_grid=params, cv=5)
grid_search.fit(X_train, y_train)
print("Best parameters: ", grid_search.best_params_)
#LightGBM
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score
# データセットの作成
train_data = lgb.Dataset(X_train, label=y_train)
test_data = lgb.Dataset(X_test, label=y_test)
# パラメータの設定
params = {
'objective': 'binary',
'metric': 'binary_logloss',
'feature_fraction': 0.9,
'n_estimators': 100,
'num_leaves': 127,
'boosting_type': 'dart',
'learning_rate': 0.2,
'max_depth': 15,
}
# 学習の実行
num_round = 100
bst = lgb.train(params, train_data, num_round, valid_sets=[test_data])
# 予測の実行
y_pred = bst.predict(X_test)
y_pred_binary = [int(pred >= 0.5) for pred in y_pred]
# 結果の表示
accuracy = accuracy_score(y_test, y_pred_binary)
print('Accuracy: {:.6f}'.format(accuracy))
XGboostには劣りますが暫定2位のハイスコアです。パラメータの検索範囲をもう少し広くすればスコアアップが期待できそうです。
4. K-NN(K近傍法)
k-NNはk近傍法とも呼ばれ、教師あり学習の一種で、データが所属するクラスを求めるために使用されます。具体的には、新しいサンプルに対して、最も近いk個の既知のデータ(近傍点)を見つけ、その多数決によってクラスを判定します。k-NNは、単純な手法であり、特に高次元のデータに対しては精度が低下する傾向がありますが、適切な距離尺度を使用することで高い精度を発揮することができます。また、モデルの学習コストがほとんどかからないため、小規模なデータセットに適しています。
早速実装していきましょう
from sklearn.neighbors import KNeighborsClassifier
model = KNeighborsClassifier()
# モデルの学習
model.fit(X_train, y_train)
# 正解率の表示
print(model.score(X_train, y_train))
from sklearn.neighbors import KNeighborsClassifier
from sklearn.model_selection import GridSearchCV
from sklearn.metrics import accuracy_score
# パラメータグリッドを設定
param_grid = {
'n_neighbors': [3, 5, 7, 9],
'weights': ['uniform', 'distance']
}
# グリッドサーチのインスタンスを作成
knn = KNeighborsClassifier()
grid_search = GridSearchCV(knn, param_grid, cv=5)
# モデルの学習と予測
grid_search.fit(X_train, y_train)
y_pred = grid_search.predict(X_test)
# 最適なパラメータとスコアを出力
print('Best parameters: ', grid_search.best_params_)
print('Accuracy: ', accuracy_score(y_test, y_pred))
from sklearn.neighbors import KNeighborsClassifier
model = KNeighborsClassifier(n_neighbors=3, weights='distance')
# モデルの学習
model.fit(X_train, y_train)
# 正解率の表示
print(model.score(X_train, y_train))
今までのモデルで一番のハイスコアが得られました。データの量や性質が最適だったのでしょうか。
5. ニューラル・ネットワーク
最後にニューラル・ネットワークです。
ニューラルネットワークは、神経細胞を模倣して構築された数学的モデルで、複数の層に分かれたニューロンが、それらの入力から重み付けされた信号を受け取り、出力を生成します。このアルゴリズムは、膨大な量のデータを解析し、パターンや関係性を特定するために使用され、機械学習やディープラーニングの分野で広く用いられています。ニューラルネットワークは、画像、音声、自然言語処理などの分野での高度な認識能力を持ち、人工知能やロボット工学、自動運転などの分野での応用が期待されています。
今回は教材の復習も兼ねて実施しました。まずはデフォルト値での実施です。
import keras
from keras.models import Sequential
from keras.layers import Dense
# モデルの定義
model = Sequential()
model.add(Dense(8, input_dim=X_train.shape[1], activation='relu'))
model.add(Dense(1, activation='sigmoid'))
# モデルのコンパイル(デフォルトのパラメーターを使用)
model.compile(loss='binary_crossentropy', optimizer='rmsprop', metrics=['accuracy'])
# モデルの学習(デフォルトのパラメーターを使用)
history = model.fit(X_train, y_train, epochs=10, batch_size=32, validation_data=(X_test, y_test))
# モデルの評価
score = model.evaluate(X_test, y_test, verbose=0)
print('Test loss:', score[0])
print('Test accuracy:', score[1])
正答率71.4%というかなり低い数値が出ました。ニューラル・ネットワークはチューニング要素が多くデフォルト値が不適切だったと思われます。
次にパラーメータサーチです。
import tensorflow as tf
from sklearn.model_selection import GridSearchCV
# ニューラルネットワークを定義する
def create_model(activation='relu', optimizer='adam'):
model = tf.keras.Sequential([
tf.keras.layers.Dense(64, activation=activation),
tf.keras.layers.Dense(32, activation=activation),
tf.keras.layers.Dense(1, activation='sigmoid')
])
model.compile(optimizer=optimizer, loss='binary_crossentropy', metrics=['accuracy'])
return model
# ハイパーパラメータの候補を設定する
params = {
'activation': ['relu', 'tanh'],
'optimizer': ['adam', 'sgd'],
'batch_size': [32, 64],
'epochs': [10, 20]
}
# GridSearchCVで最適なパラメータを探索
nn_grid = GridSearchCV(estimator=tf.keras.wrappers.scikit_learn.KerasClassifier(build_fn=create_model), param_grid=params, cv=5)
nn_grid.fit(X_train, y_train)
# 最適なパラメータを表示
print("Best parameters:", nn_grid.best_params_)今回は上記の範囲でグリッドサーチを実施しました。得られた結果を入力したとこ正答率は以下となりました。

かなり改善が見られました。
もう少しハイスコアが期待できると思い、手動で調整したところ以下のコードで最適解が得られました。
import keras
from keras.models import Sequential
from keras.layers import Dense
from tensorflow.keras import optimizers
import tensorflow as tf
# モデルの定義
model = Sequential()
model.add(Dense(264, input_dim=X_train.shape[1], activation='relu'))
model.add(Dense(128, input_dim=X_train.shape[1], activation='relu'))
model.add(Dense(32, input_dim=X_train.shape[1], activation='relu'))
model.add(Dense(1, activation='sigmoid'))
opt = tf.keras.optimizers.Adam(learning_rate=0.01)
# モデルのコンパイル
model.compile(loss='binary_crossentropy', optimizer='adam', metrics=['accuracy'])
# モデルの学習
history = model.fit(X_train, y_train, epochs=15, batch_size=32, validation_data=(X_test, y_test))
# モデルの評価
score = model.evaluate(X_test, y_test, verbose=0)
print('Test accuracy:', score[1])
数十回実施しましたが他のモデルに及びませんでした。データが不向きだったのか、チューニング調整が不十分だったようです。
モデルの評価
5つのモデルの検証が終わりました。モデルの評価をしてみましょう。
正答率ランキング
今回の検証では以下のような結果となりました。
K-NN : 99.61%
XGBoost : 99.37%
LightGBM : 99.35%
ランダムフォレスト: 99.30%
ニューラル・ネットワーク:98.27%
考察・反省
正答率ランキング結果から、K-NNが最も高い正答率を示していることが分かります。一方で、ランダムフォレスト、LightGBM、XGBoostも高い正答率を示していますが、K-NNよりも若干低い結果となっています。これらのモデルは、決定木をベースとしたアンサンブル学習の手法であり、K-NNとは異なるアプローチでデータを解析しています。
今回のデータセットに対しては比較的単純な手法であるK-NNが最も適しており、より複雑なデータに対応できるニューラル・ネットワークがうまく機能しなかった可能性があります。次回はより複雑なデータセットで検証をしていきたいと思います。
また、パラメータの設定やモデルのアルゴリズムに関して知識が十分でないことも痛感しました。今後は統計学や数学の知識を積極的に取り入れ、引き続き学習を強化していきたいと思います。
参考URL
今回の検証にあたり以下のサイトを参考にさせていただきました。
今回のコードは以下に格納してあります。
この記事が気に入ったらサポートをしてみませんか?
