【W5】化合物クラスタリング_06_選抜結果
【W5の目的】
・化合物をグループ化する方法と、多様性のある化合物セットを選ぶ方法
を学びます。Python版TeachOpenCADDのT5が対応しますが、こちらはより発展的です。
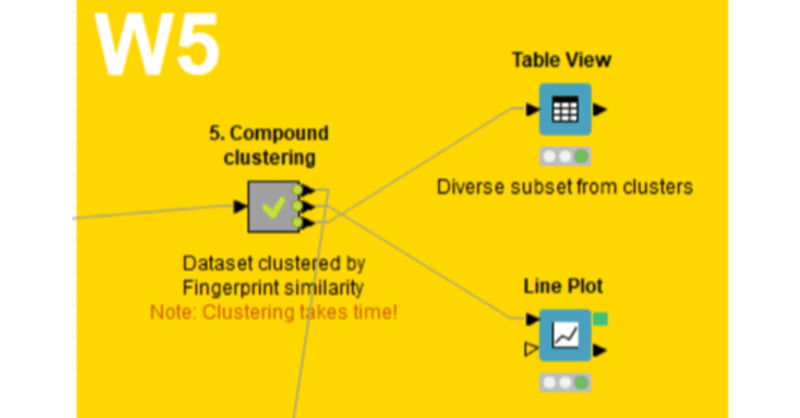
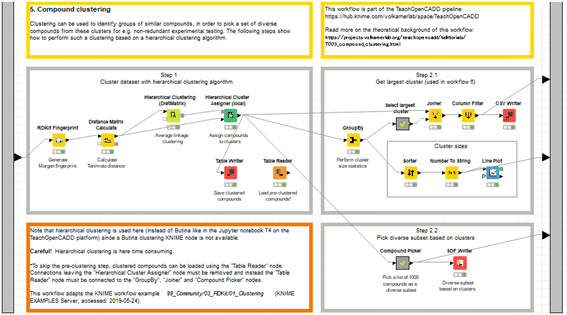
W5の本体であるメタノード「5. Compound clustering」で階層型クラスタリングによるグループ化と多様性のある化合物セットを選ぶ方法を体験しました。
W5の最終回となる今回は、2通りの出力形式で選抜結果を見てみます。
【Table View】
ノードの操作法などの説明はW1-3でしてきたので割愛します。
日本語化されたディスクリプションを引用します。
データをHTMLテーブルビューで表示します。
このビューには、いくつかのインタラクティブな機能があり、行を選択することもできます。
ノードはカスタムCSSスタイルをサポートしています。
CSSルールを1つの文字列にまとめて、ノードの設定ダイアログでフロー変数「customCSS」として設定するだけです。
CSSに抵抗感がない方ならばカスタマイズはいろいろできるでしょうが、TeachOpenCADDではデフォルト設定でのみ利用しています。
結果:
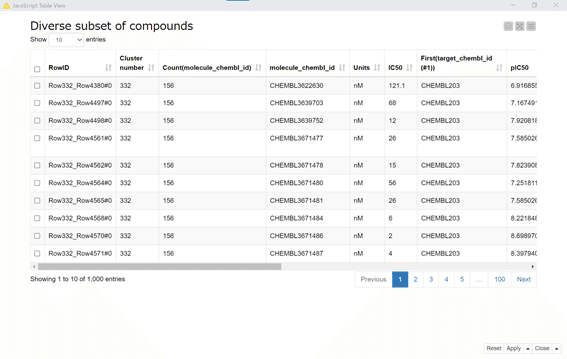
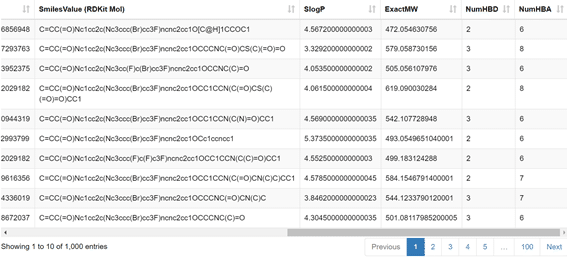
1000化合物を1ページに10化合物ずつ表示しています。
構造情報はSMILESの文字列表示になっています。
折角なので構造式も表示して見たいだろうと思います。KNIMEでMarvin Viewを使って閲覧する手法は既に紹介しました。
Step2.2で書き出したSDファイル(SDF)はご自身の利用する任意のViewerで化合物リストを見たいときに有用です。
【デモデータのSDFを入手する】
W5のStep2.2のSDF Writer
の設定をみてファイル名を確認してください。
相対パス表示なので初心者には探しにくいです。
こういう時はファイル名で検索すると比較的楽です。
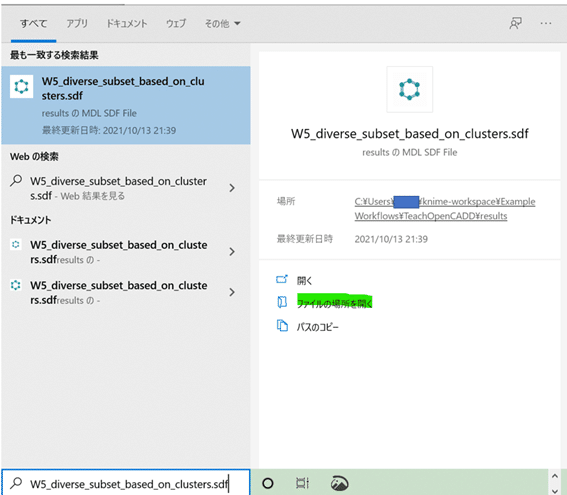
「ファイルの場所を開く」を選んでみましょう。
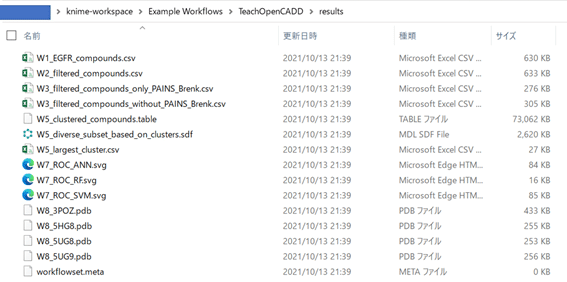
TeachOpenCADDでは各種データファイルをresultフォルダにまとめて格納する仕様になっています。目的の「W5_diverse_subset_based_on_clusters.sdf」もありますね。お好きなプログラムでご利用ください。
今回はあえてデータの中身を見ていこうと思います。
【SDFのしくみについて】
SDFについては下記のサイトの説明が丁寧です。
<参考リンク>船津研究室「ケモインフォマティクスのオンライン入門書」
コンピュータで画像を保存する場合、jpeg,png,gifなどの特定の形式でファイルにデータを保存すると思います。
それと同じように、化合物の構造を保存する場合にも、特定の形式でファイルに保存することができます。
(ここでは、化合物の構造とは3次元の構造を想定していますが、2次元の場合もあります。)
画像データに様々な形式があるように、化合物の構造データの場合にもいくつかの形式があります。
MOL形式とSDF形式
まず初めに、MOL形式やSDF(Structure Data File)形式がどういうものかということについて軽く説明します。
これらはいずれも分子内の原子の座標を記録したものであり、行列表記法とも呼ばれます。 また、この2つといくつかの派生した形式を総称してCTfileという名前がつけられています。
MOL形式とSDF形式の最も大きな違いは、1ファイルにつき1化合物か複数化合物かということです。 実は、SDF形式の中で実際に構造を表す部分はMOL形式がそのまま使われています。 SDF形式ではそれに加えて物性値など、構造以外の情報も保存することができます。
デモデータを使って具体的に見てみます。
先述の「W5_diverse_subset_based_on_clusters.sdf」をあえてテキストエディタ(メモ帳とか、サクラエディタとか)で開いてみます。
RDKit 2D
30 33 0 0 0 0 0 0 0 0999 V2000
-7.5000 -5.1962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.0000 -5.1962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2500 -3.8971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.0000 -2.5981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7500 -3.8971 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
-3.0000 -2.5981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5000 -2.5981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7500 -1.2990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7500 -1.2990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5000 -2.5981 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
3.0000 -2.5981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7500 -3.8971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2500 -3.8971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.0000 -2.5981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.5000 -2.5981 0.0000 Br 0 0 0 0 0 0 0 0 0 0 0 0
5.2500 -1.2990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7500 -1.2990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0000 -0.0000 0.0000 F 0 0 0 0 0 0 0 0 0 0 0 0
1.5000 0.0000 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
0.7500 1.2990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7500 1.2990 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
-1.5000 0.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0000 0.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7500 -1.2990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2500 -1.2990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.0000 0.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-7.4918 0.1568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-7.8037 1.6240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.5046 2.3740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.3899 1.3703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0
2 3 1 0
3 4 2 0
3 5 1 0
5 6 1 0
6 7 2 0
7 8 1 0
8 9 2 0
9 10 1 0
10 11 1 0
11 12 2 0
12 13 1 0
13 14 2 0
14 15 1 0
14 16 1 0
16 17 2 0
17 18 1 0
9 19 1 0
19 20 2 0
20 21 1 0
21 22 2 0
22 23 1 0
23 24 2 0
24 25 1 0
26 25 1 1
26 27 1 0
27 28 1 0
28 29 1 0
29 30 1 0
24 6 1 0
30 26 1 0
22 8 1 0
17 11 1 0
M END
> <Cluster number>
332
> <Count(molecule_chembl_id)>
156
> <molecule_chembl_id>
CHEMBL3622630
> <First(target_chembl_id (#1))>
CHEMBL203
$$$$「$$$$」が区切りで、上記は最初の1化合物のデータです。
テキスト形式で見るとこんな風にデータが入っているとわかります。
次の行からは1000化合物分のデータが同様に格納されています。
各部分を少し見てみましょう。
RDKit 2D
30 33 0 0 0 0 0 0 0 0999 V2000は今回のSDFのデータ形式の情報です。先述の船津研の記事から引用します。

1行目、3行目は書いた通りの意味で、特に無ければ空行となります。
2行目は、データを作成したプログラム名、作成日時、座標の次元(2D or 3D)などが記入されます。
(ここまでの情報の中でRDKitで読み込まれるのは分子名と次元だけです。)
4行目は、原子数、結合本数などを表し、ここの値がこの後のAtomブロック、Bondブロックの行数になります。
その下の
-7.5000 -5.1962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0から始まり
M ENDまでの数値の行列やリストがMOL形式での化合物の構造データです。
Atomブロックは、各行が各原子の情報を表し、何の原子がどこにあるかを表します。
同様に、Bondブロックは各行が各結合を表し、どの原子とどの原子が何次で結合しているかを表します。 ここでの番号は、Atomブロックの何番目の原子かという意味です。
おー見た目もまさにツーブロック。失礼しました。
以降はPropertiesブロック
Propertiesブロックは、構造に関する補足の情報を表します。
> <Cluster number>
332など、各データ名と内容がリストアップされます。
このお作法が定型となっています。詳しくは記事を熟読されることをお勧めします。
SDFについては以上です。次回はW6へ進めます。
おまけ:
【Marvinライセンス】
Marvin Viewで結果を見せようとしたら、
出ました。
Oops!トライアル期間が過ぎていました。
楽しい時間をありがとう。
記事を読んでいただきありがとうございます。 先人の智慧をお借りしつつ、みなさんに役立つ情報が届けられたらと願っています。 もしサポートいただけるなら、そのお金はKNIMEの無料勉強会の開催資金に充てようと思います。
