【W4】リガンドベーススクリーニング_04_Step2_Tanimoto係数
【本パート(W4)の目的】
化合物をエンコード(記述子、フィンガープリント)し、比較(類似性評価)する様々なアプローチを取り扱います。さらに、バーチャルスクリーニングを実施します。
上記はPython版のT4の説明ですが、W4の目的も同じです。
そのための教材として
既知のEGFR阻害剤ゲフィチニブ(Gefitinib)をクエリとして使用し、EGFRに対して試験済みの化合物データセットの中から類似した化合物を検索します。
Step1で化合物をエンコードしたので、類似性評価へ進めます。
【Tanimoto係数を指標とした類似度評価】
Magattacaさんの記事から引用します。
記述子/フィンガープリントの計算ができれば、それらを比較することで、二つの分子の間の類似度を評価することができます。化合物類似度は様々な類似度係数で定量化することができますが、よく使われる2つの指標はタニモト係数とDice係数です(Tanimoto and Dice index)
J. Med. Chem. (2014), 57, 3186-3204)。https://pubs.acs.org/doi/abs/10.1021/jm401411z

化合物の特徴量抽出も多数の手法があり、類似度係数も様々あります。迷っちゃいますね。第一選択としてはMorganフィンガープリントで比較し、タニモト係数でスコア化が多いと思うので、今回はひとまずこの1条件だけ体験してみることにします。
【KNIMEで類似性評価してみよう】
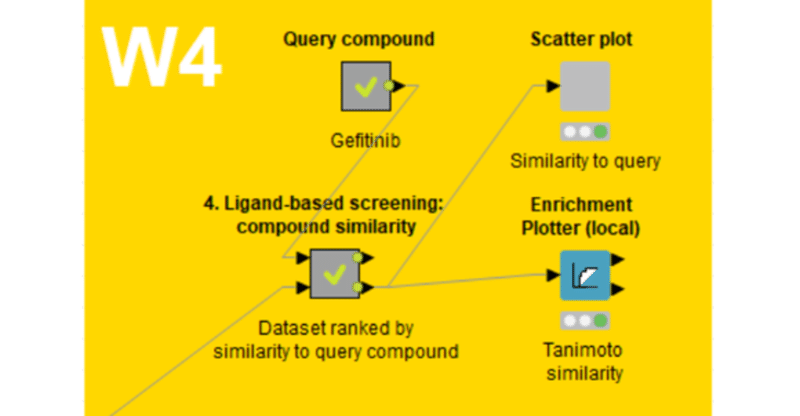
今回は
「4. Ligand-based screening: compound similarity」メタノード内のStep2
の最上列のフローのみ扱います。
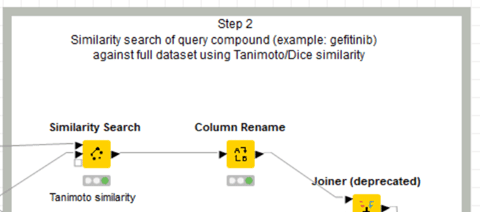
Magattacaさんの記事から引用します。
類似性検索はある特定のデータベースの全ての化合物と一つの化合物との間の類似度を計算することで実行することができます。データベースの化合物の類似度係数によるランク付けにより、最も類似度の高い分子が得られます。
ランク付けをして、しきい値を決めて絞り込めばバーチャルスクリーニングです。とりあえず類似度を計算してみましょう。
【Similarity Search】Tanimoto係数を利用
日本語化したノードディスクリプションより引用します。
このノードは、クエリテーブル(ポート0)の各行を受け取り、参照テーブル(ポート1)を検索して、指定された類似性/距離の基準に一致する複数の行を探します。
複数の結果が要求された場合、クエリ結果の行は後続のマッチごとに複製されます。
参照テーブル(ポート1)へは今回ゲフィチニブ1行だけを入力していますが、複数化合物を入力して類似度評価もできるようです。例えばですが、5つの参照化合物を使ってクエリテーブル(ポート0)の各化合物に対して類似度を算出し、一番近い参照化合物との類似度データを出力することなどができたりします。合成展開時に複数のキー化合物があるときに使ってみたい機能です。
今回はゲフィチニブを参照化合物として、いろいろな類似度評価を体験することが目的なので、あまり深追いしません。
設定:
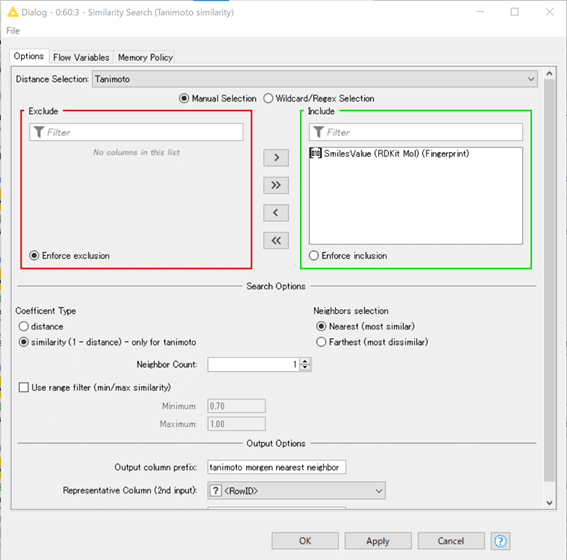
今回のノードへの入力はMorganフィンガープリントです。
Distance SelectionはTanimotoにしています。
すなわち、類似度をMorganフィンガープリントで比較し、タニモト係数でスコア化する設定です。
結果:
4510化合物に対してゲフィチニブとの類似度が算出されています。
SimilarityでDecending(降順)に並べ替えをしてみます。
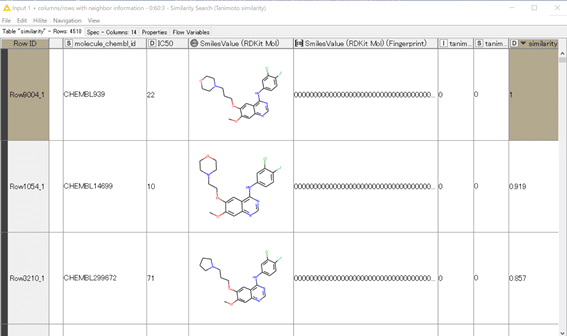
ゲフィチニブ(ChEMBL939)は同一化合物なので当然類似度が1で、アルキル鎖の長さやモルホリン環部分が異なる化合物が類似度高めに算出されています。納得できる結果になっているのではないでしょうか?
IC50値の違いもゲフィチニブ(22nM)と比較して2~3倍の範囲ですね。
構造的に類似の化合物は類似の特性、そして類似の生理活性を示すという考え方は、類似性質原則(similar property principle、SPP)や構造活性相関(structure activity relationship、SAR)に表れています。この文脈において、バーチャルスクリーニングは、結合親和性のわかっている化合物セットがあれば、そのような化合物をさらに探すことができる、というアイデアに基づいています。
今回のデモデータでは
注:ChEMBLにはゲフィチニブ(よく研究された化合物なので)の完全な構造活性相関分析がふくまれていて、したがって私たちが取得したデータセットにゲフィチニブ様化合物が多く含まれていることは驚くべきことではありません。
とのことです。類縁体がちゃんと上位に来ているのはバーチャルスクリーニングの体験としてはまずまず良い結果だったと思います。実際のバーチャルスクリーニングだと意外な上位化合物に驚くこともあるので。
【Column Rename】
設定:
結果:

あとで、各種類似度評価した結果をJoinerで集計するので、どの条件で算出したSimilarityかを明記しました。
次回以降もworkflowとしての作りは同様ですが、類似度の評価関数を変えて体験します。
おまけ:玄人と話す時は気を付けましょうと言うお話です。
今回も@maskot1977さんの記事を紹介させていただきます。
化合物の類似性を語るにあたって、有名な指標に「タニモト係数」があります。ところが、「タニモト係数」とだけしか言わない人がけっこういて、「それだけでは説明不十分ですよね?」って悶々とする機会がけっこうあるわけです。
玄人の方と語るときには算出条件をなるべく詳細にお伝えしたいですね。
記事を読んでいただきありがとうございます。 先人の智慧をお借りしつつ、みなさんに役立つ情報が届けられたらと願っています。 もしサポートいただけるなら、そのお金はKNIMEの無料勉強会の開催資金に充てようと思います。
