【W6】最大共通部分構造_02_Step1前編
【W6の目的】
化合物セットが共通してもつ最大の部分構造を計算する方法を学びます。
workflow概要:
【MCS解析】
最大共通部分構造(maximum common structure、MCS)は2つあるいはそれ以上の対象化合物に含まれる最大の部分構造として定義されています。
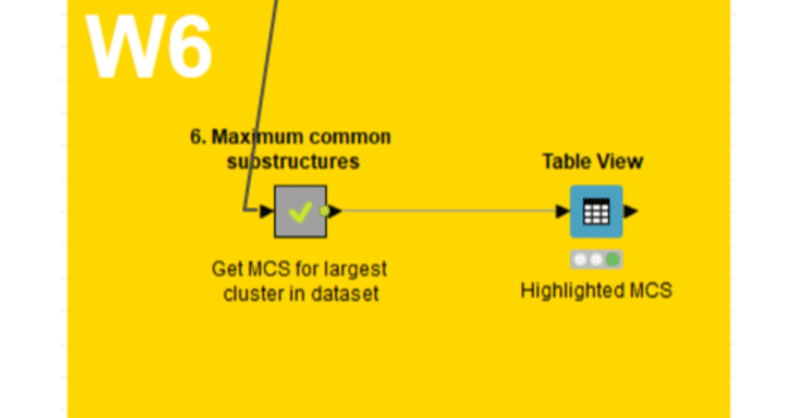
6. Maximum common substructuresメタノード
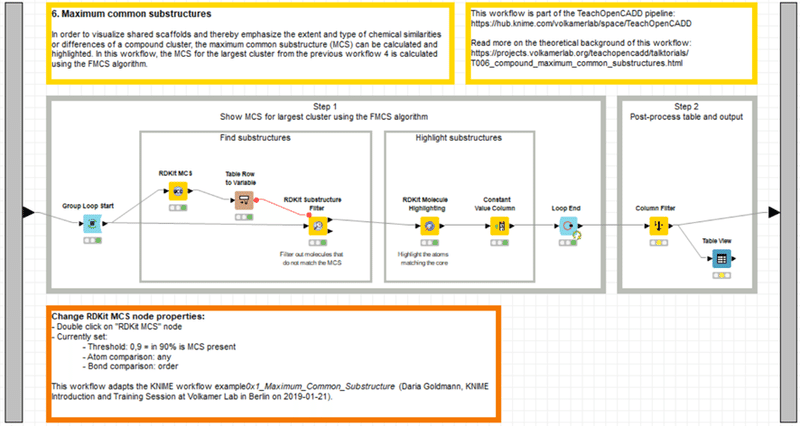
のStep1の前半を今回見ていきます。
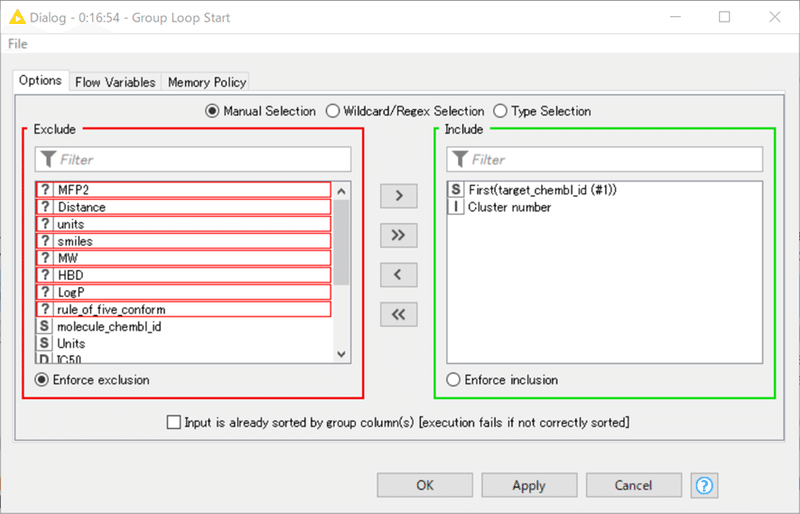
【Group Loop Start】
入力データですが、デモデータはW5のStep2.1で抽出した、最大クラスターの156化合物です。
このworkflowではGroup Loop Startを使って、クラスター別にループを回す設定となっています。基本的にW5での利用例と同様です。
上記のように、今回は最大クラスター1つだけを扱うので実はループ処理は必要ないのですが、おそらく設計はデモデータ全てすなわち4511化合物をまとめて解析するときなども想定してより汎用性が高く作られているのではないでしょうか。
後日紹介予定ですが、W6ではMCSの計算条件をふっていろいろと試すことでより理解が進みます。今回のデモデータの設定は、あえて単一のクラスターで計算時間を節約して何種類もの解析条件を試すことができるように工夫されているのではないかと思います。前置きが長くなってしまいました。設定を見ておきましょう。
設定:
処理結果:
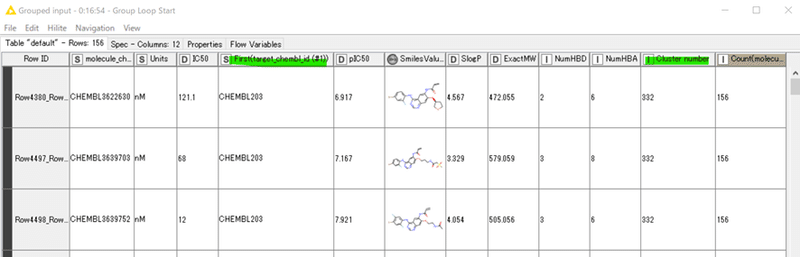
入力データが
First(target_chembl_id(#1)) = CHEMBL_203
Cluster number = 332
の化合物しかないので、1回目のループで全156化合物が処理されます。
つまり今回のデモデータでは入力と出力で行数は同じです。
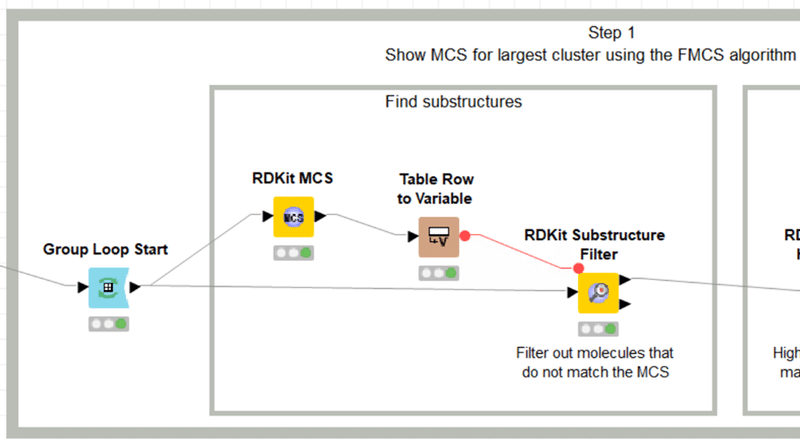
【RDKit MCS】
W6の最重要ノードです。RDKitのFindMCS(FMCS)を実施できます。
日本語化されたディスクリプションから引用しながら設定を解説します。
このノードは、RDKitのMCSコードを使用して化合物セットの最大共通部分構造(MCS)を見つけるために使用されます。
設定:
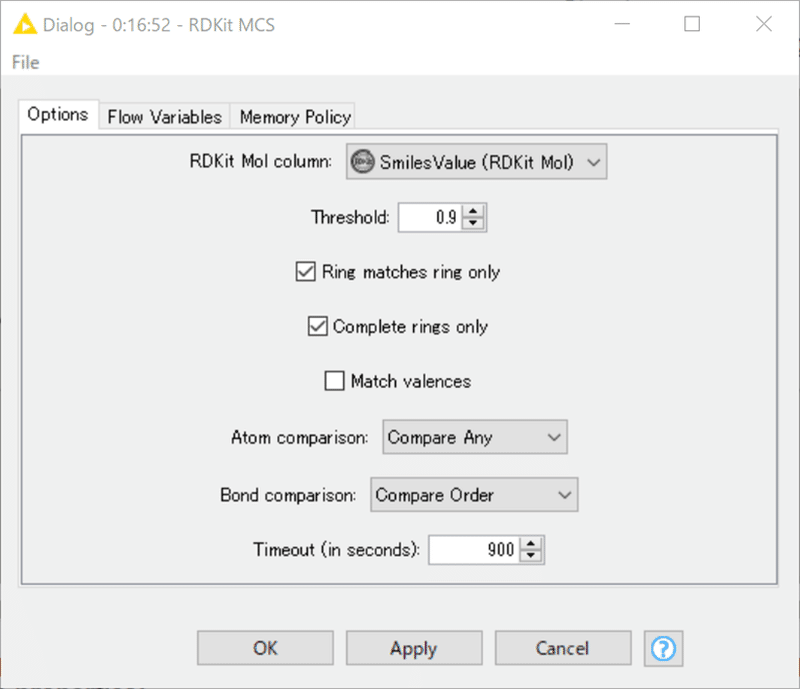
RDKit molecule column
入力として使用する RDKit Molecule カラムをここで選択します。
Threshold
MCSがカバーしなければならない分子の割合。
デフォルトは1.0です。
0.0 < threshold ≤ 1.0です。
Ring matches ring only
リングボンドが他のリングボンドにのみマッチするかどうかを決定します。
デフォルトではチェックされていません。
Complete rings only
完全なリングのみをMCSに含めるかどうかを決定する。
デフォルトではチェックされていません。
Match valences
比較の際に原子の価数を使用するかどうかを決定します。
デフォルトはチェックされていません。
Atom comparison
ここでは、原子の比較方法を指定します。
Compare Any, Compare Elements, Compare Isotopes.
また、Bond comparisonは比較時に結合様式の一致判定の条件を指定します。
細かいHit判定条件は下記の記事で丁寧に説明されていますのでご覧ください。KNIMEのノードやPythonコードとともに丁寧に情報が開示されるだけでもありがたいのに、有志の方が解説を加えて下さっており、RDKitコミュニティは本当に素晴らしいなとあらためて思います。
さらに
このような計算は長時間の処理になる可能性があります。
そのため、オプションの一部としてタイムアウトを指定する必要があります。
実はデモデータだと初期設定だと警告が出ています。
WARN RDKit MCS 0:16:52 The configured timeout is greater than 5 minutes. Please note that
the MCS calculation continues in the background and occupies the processor
until it is done or that timeout is hit, even after cancelling the node.そこで今回のデモデータ156化合物の場合は例えばですが
初期設定の300秒
![]()
から先述の図のように900秒へ変更しておくと警告が出なくなります。
結果:
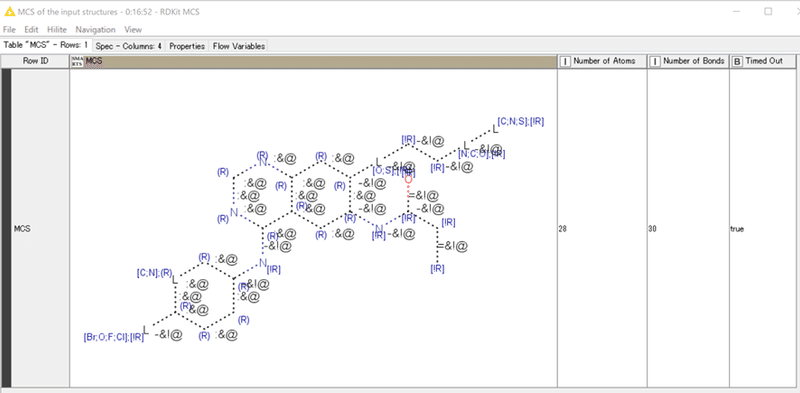
デモデータ156化合物のうち、0.9すなわち9割の化合物に共通する部分構造をSMARTS形式で示しています。今回は詳しくは見ないで次へ進めます。
【Table Row To Variable】
前にも紹介した記事ですが再掲します。
変数とは何かから詳しくまっきーさんが解説して下さっています。
Table Row to Variableは良く使います。KNIMEは変数を処理するNodeよりもテーブルの値を処理するNodeが多いですからね。このNodeはTableの1行目の値を変数に変換するNodeです。
設定:
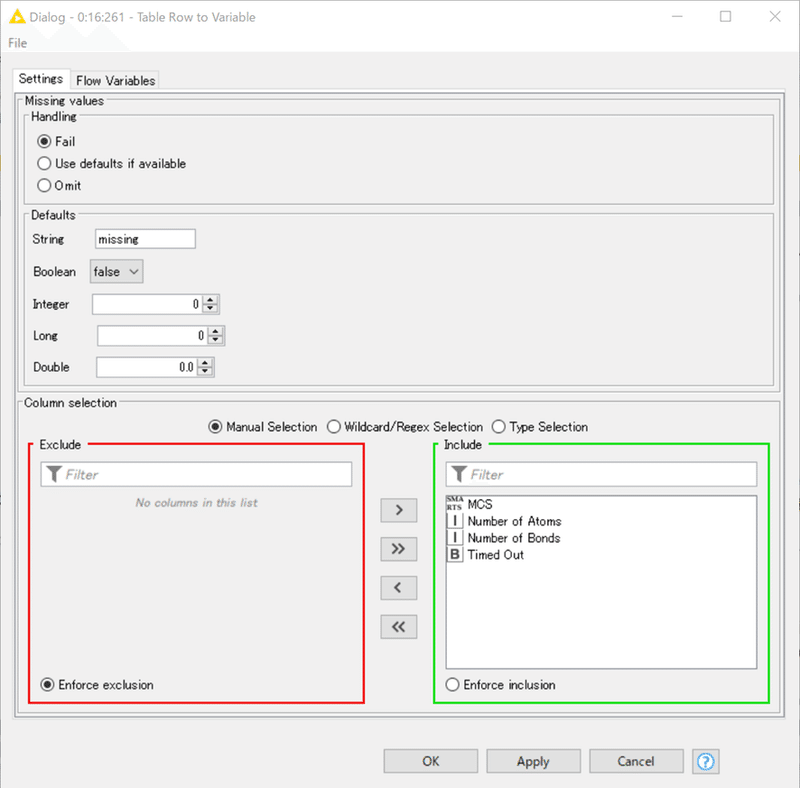
実際にあとで使う変数はMCSだけですが、ここでは特に変数への変換対象カラムの絞り込みをしていないです。
結果:
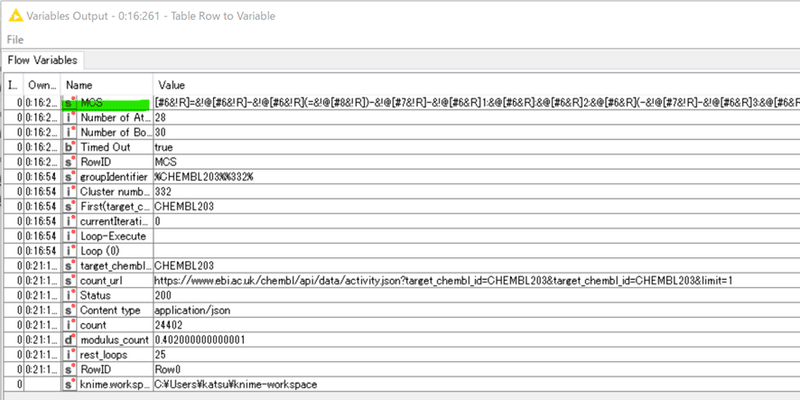
設定より多くの変数が出力されていて驚くかもしれないですが、W1-5で取得した各種変数もあります。
変数”MCS”を次のノードで利用します。
【RDKit Substructure Filter】
ノード自体はW3で利用しました。
今回は上記のMCS全ての化合物のうち9割に一致する共通部分構造ですので、MCSをQueryとして部分構造検索すれば、残りの1割が除去されます。
設定:
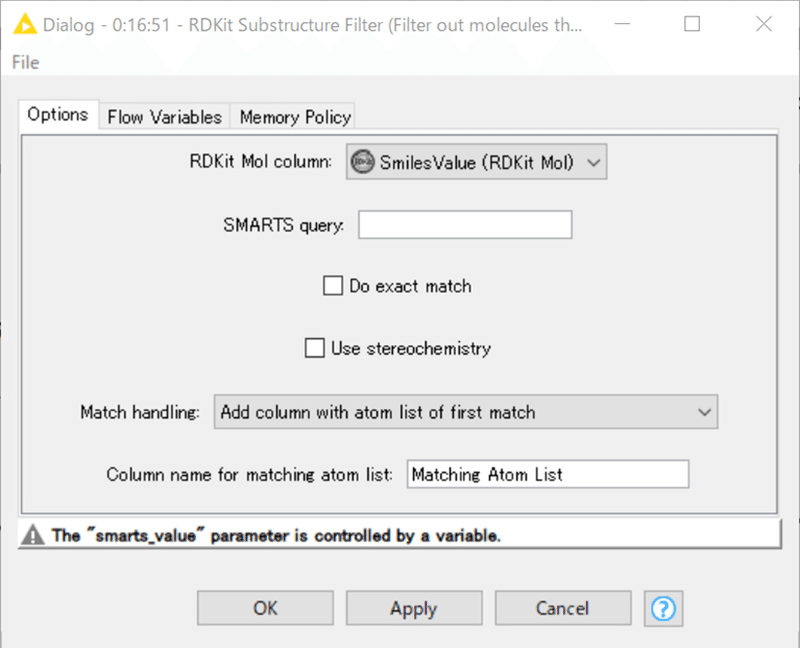
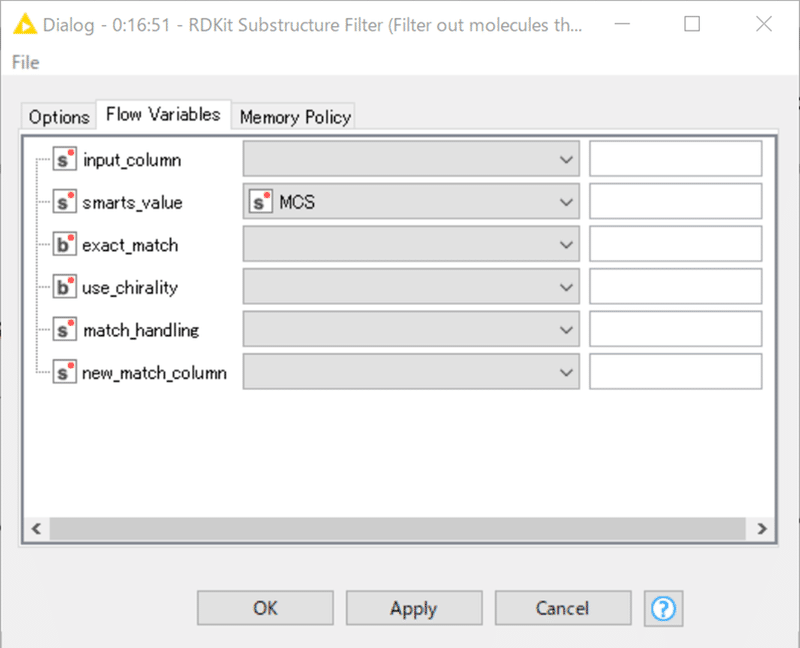
QueryとなるSMARTSは上述の変数”MCS”で定義します。
結果:
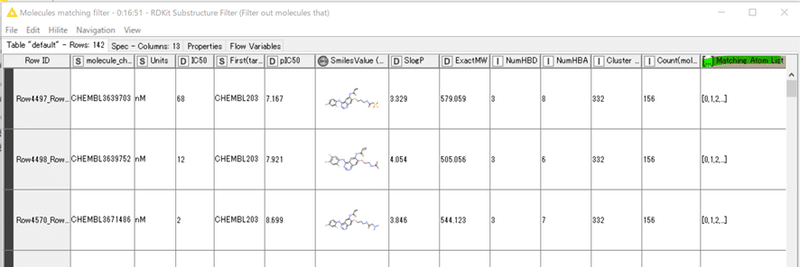
入力の156化合物から9割に当たる142化合物が選抜されています。
また、Matching Atom Listカラムが増えています。
KNIMEをつかうと、ノーコードでMCS解析が実施できました。
次はMCSの可視化へ進めます。
記事を読んでいただきありがとうございます。 先人の智慧をお借りしつつ、みなさんに役立つ情報が届けられたらと願っています。 もしサポートいただけるなら、そのお金はKNIMEの無料勉強会の開催資金に充てようと思います。
