【W5】化合物クラスタリング_05_Step2.2
【W5の目的】
化合物をグループ化する方法と、多様性のある化合物セットを選ぶ方法
を学びます。
Python版TeachOpenCADDのT5が対応しますが、こちらはより発展的です。
【多様性のある化合物セットの選び方の例】
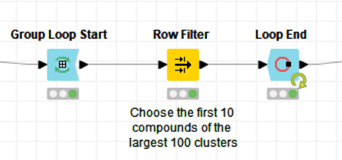
W5の本体であるメタノード「5. Compound clustering」
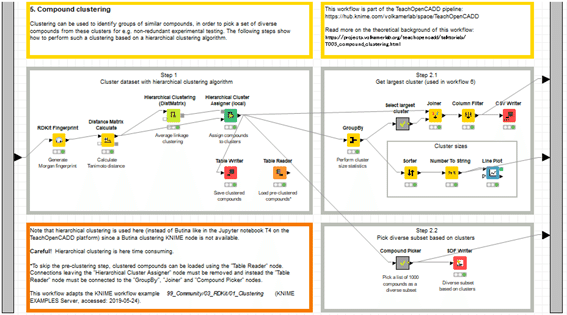
のStep1で階層型クラスタリングを体験しました。Step2.1ではクラスターサイズを算出してデータ処理しました。今回はStep2.2を扱います。多様性のある化合物セットを選ぶ方法を体験します。
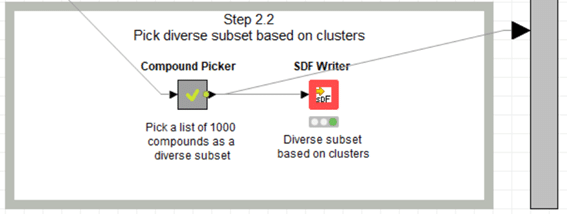
【Compound Pickerメタノード】
機能は化合物クラスタリングの結果を利用して多様性に考慮して1000化合物に絞ります。より具体的には、デモデータで言えば4511化合物のクラスタリング結果をデータ処理して、大きなクラスターから順に100個のクラスターからそれぞれ化合物を10ずつ選びます。
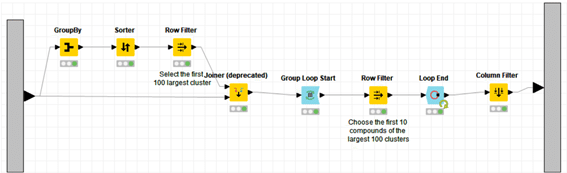
メタノードの中身のワークフローは上図の通りです。順番に見ていきましょう。
【クラスターサイズ算出】GroupByとSorter
Step2.1で全く同じ設定で使っていますので記事引用に留めます。
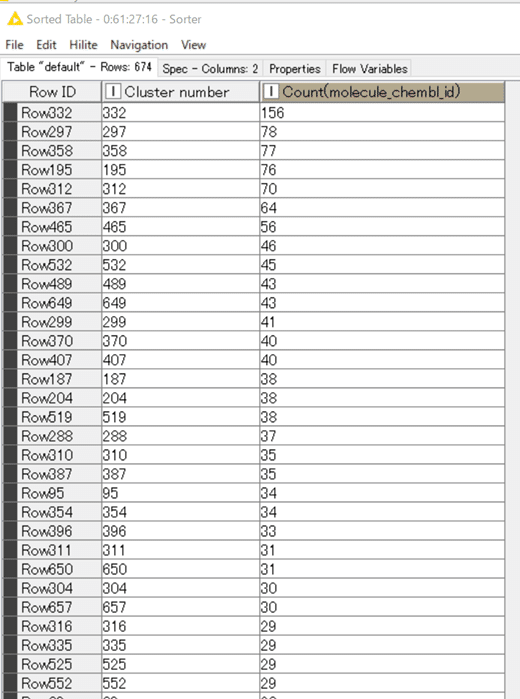
結果:Sorterまでの実行結果
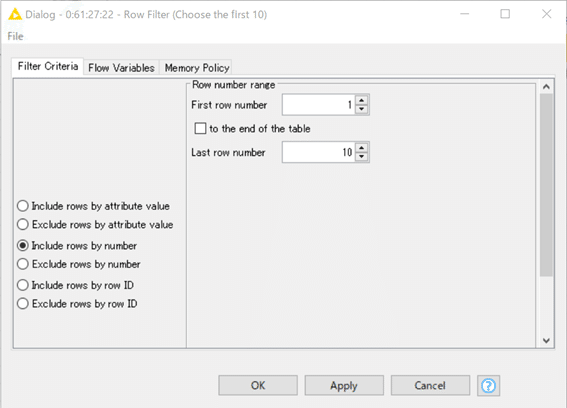
【Row Filter】クラスターを選抜
前回同様に日本語化されたディスクリプションより再掲します。
ノードでは、特定の基準に従って行をフィルタリングできます。特定の範囲(行番号による)、特定の行IDを持つ行、および選択可能な列(属性)内の特定の値を持つ行を含めることも除外することもできます。
今回は上位の100行だけに絞ります。
設定:
結果:
敢えて下位の方を表示しました。
100番目に大きいクラスターはID番号321で、化合物数は12です。
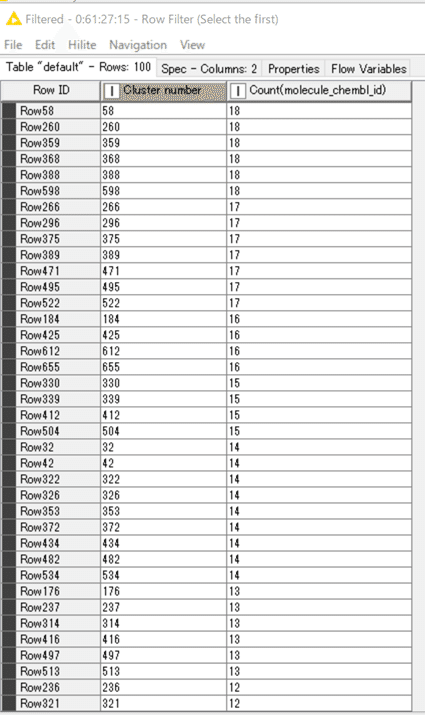
デモデータでは上位100クラスターから10こずつの化合物抽出で、1000化合物が選べますね。デモデータ以外の実際の場合、このクラスター群の分布なども見ながら選び方を工夫することでしょう
【Joiner】
Joinerも基本的に前回と同じ用途と設定で利用していますので設定は割愛します。
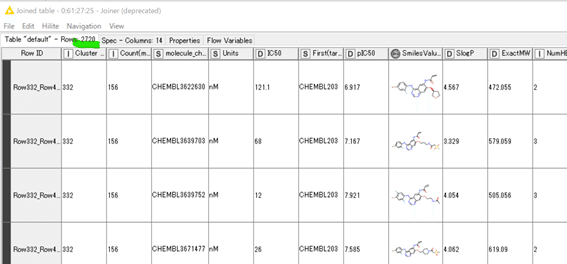
結果:
2720化合物のデータが取れました。
【Groupでのループ処理】
日本語化されたGroup Loop Startのディスクリプションがとてもわかりやすいと思います。
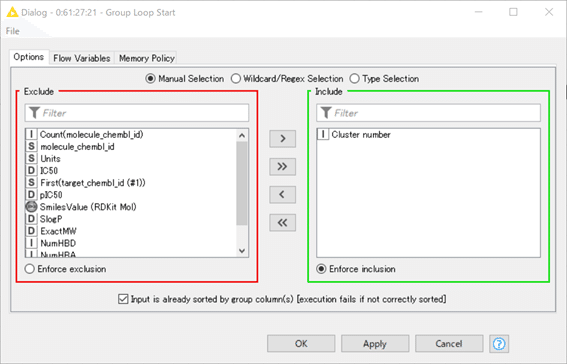
グループループを開始し、各反復で別のグループの行を処理します。
グループ化の対象となる列を指定する必要があります。
デフォルトでは、ループを開始する前に、指定された列に基づいて入力データテーブルがソートされます。
グループ化する列に基づいて入力データテーブルがすでに適切にソートされている場合は、ソートをオフにすることができます。
今回のデモデータはまさにすでに適切にソートされたリストを処理します。
グループ化の対象となる列とはID番号であるCluster Numberですね。
設定:
Cluster Numberごとに2720化合物を分けて、次の処理を行います。
【Row Filter】10化合物ずつ選抜

設定:
今回はトータルで100回ループが回ります。各回でどんなデータが出てくるかなどループ処理を詳しく追いたい場合はLoop Endノードを右クリックして
Step Loop Executionを何回かクリックして各ノードの出力をご確認ください。
こういう確認がしやすいのはKNIMEの長所かなと思います。
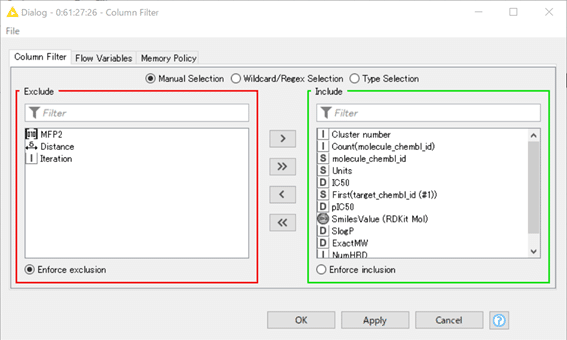
【Column Filter】
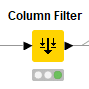
こちらも基本的にStep2.1の同ノードと利用目的は似ています。
設定:
SDFファイルへの書き出しに備え、不要なフィンガープリントと距離ベクトル、そしてループの回数データを除いています。
結果は省略させていただきます。
メタノードの中の説明はここまでです。
【SDF Writer】
W1-4まで振り返って一度もSDファイル(SDF形式)に出力してないですね。ケモインフォマティクスを扱っているとSDF形式はとてもよく使うと思います。
日本語化したディスクリプションより
SDF Writerノードでは、分子をMDLの構造データファイル(SDF)に保存します。
…はい。正確なのですがもう少し説明が欲しいので下記を紹介しておきます。
<参考リンク>船津研究室「ケモインフォマティクスのオンライン入門書」
より詳しくは次回にSDファイルの中身を見ることにして、今回はひとまずSDファイルを書き出すところまで実施します。
設定:
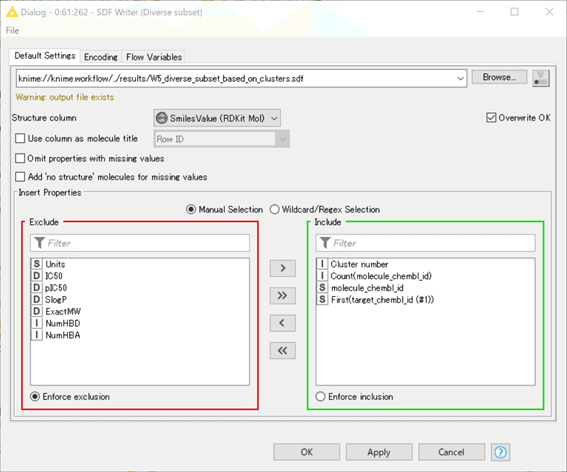
またディスクリプション情報を一部引用します。
Structure column
分子構造として扱われる入力テーブルの列。
(デフォルト:サポートされている分子構造タイプを持つ最初の列)
Use column as molecule title
出力テーブルで分子名として使用される入力テーブルの列。
(デフォルト:入力テーブルに応じて "Name "または "Drug Name"。
そのようなカラムがない場合は "Row ID")
Omit properties with missing values
選択した場合、出力ファイルには、入力テーブルで値が欠落しているプロパティは含まれません。
(デフォルト:true)
加えて、日本語では説明がなかったもう一つのメニュー
Add 'no structure' molecules for missing values
If selected, rows with missing molecule entries are written as molecules with no structure into the file. Otherwise missing molecules are simply ignored and not written into the file.
今回はチェックを入れてはいないもののデモデータの場合はそもそも化学構造データの入っていない行はW1ですでに除外されているので影響がないです。
文字列データのエンコード方式指定も読み取るプログラム側の都合で変更すべき時もあります。今回はデフォルト。
結果は指定の場所にSDファイルが書き出されています。
次回で内容を確認していきましょう。
おまけ:
多様性のある化合物セットを選ぶ方法をより広く学ぶならこちらがおすすめです。
単一のライブラリーで広範な生理活性・性質をカバーしたい場合には,できるだけ「似ていない」化合物を集めることで達成できると考えられます.
問題は類似性の議論でも述べたように,何をもって「似ている」とするかは場合によって異なるので,万能な方法がないということになります.
いかに効率よく目的のケミカルスペースを網羅できるかという問題の最適解って簡単ではないです。
更に余談となりますが最近気になった記事はこちらです。
市販化合物800万種に対するin silicoスクリーニングを、研究開始から1ヵ月未満の短期間で完了しました。
(中略)←略していいのか疑問ですが
268化合物を購入し、酵素アッセイとシロイヌナズナ生育阻害試験にて有望なヒット化合物の選抜を行いました。さらに両社が協力して、薬効向上などを目的に化合物のデザインと合成(合成展開)を行うことで、薬剤の酵素阻害効果の指標である50%阻害濃度(IC50)が100 nMを切る化合物の創出に成功し、シロイヌナズナの生育阻害効果も認められました。
今回のアグロデザインとPFNの共同研究では、両社のリソースを最大限に活かした短期間での除草剤候補化合物の創出に挑戦いたしました。その結果、大規模 in silicoスクリーニング完了まで1ヶ月未満、スクリーニングのヒット化合物選抜まで4ヶ月、初回の合成展開完了まで6ヶ月という短期間で各工程を実施し、同時に酵素アッセイ・植物生育試験で薬効を確認することに成功しました。
クラスタリング技術を活用したライブラリの濃縮とかしてるのか、まさか今やもう要らないのか。大規模 in silicoスクリーニングの詳細に興味津々です。
記事を読んでいただきありがとうございます。 先人の智慧をお借りしつつ、みなさんに役立つ情報が届けられたらと願っています。 もしサポートいただけるなら、そのお金はKNIMEの無料勉強会の開催資金に充てようと思います。
