ヒト免疫細胞へのバイオフィルム形成はVibrio choleraeの多細胞捕食戦略である
記事|186巻12号 p2690-2704.e20, 2023年06月08日号
全号ダウンロード
ヒト免疫細胞へのバイオフィルム形成はVibrio choleraeの多細胞捕食戦略である
https://www.cell.com/cell/fulltext/S0092-8674(23)00527-5
ルシア・ビダコビッチ
ソフヤ・ミハレワ 11
ハンナ・イェッケル 11
アレクサンドル・ペルサット
ロイ・アブラハム
クヌート・ドレッシャー 12
すべての著者を表示する
脚注を表示する
オープンアクセスDOI:https://doi.org/10.1016/j.cell.2023.05.008
PlumX メトリクス
ハイライト
ビブリオコレラは免疫細胞にバイオフィルムを形成し、様々な種類の免疫細胞を包み込む
免疫細胞上のバイオフィルムマトリックスは、他の表面上のマトリックスとは異なる
バイオフィルムの形成により、免疫細胞を殺すための毒素ヘモリシンの送達が促進される
免疫細胞上のバイオフィルム形成は、攻撃的な多細胞細菌の特徴である
まとめ
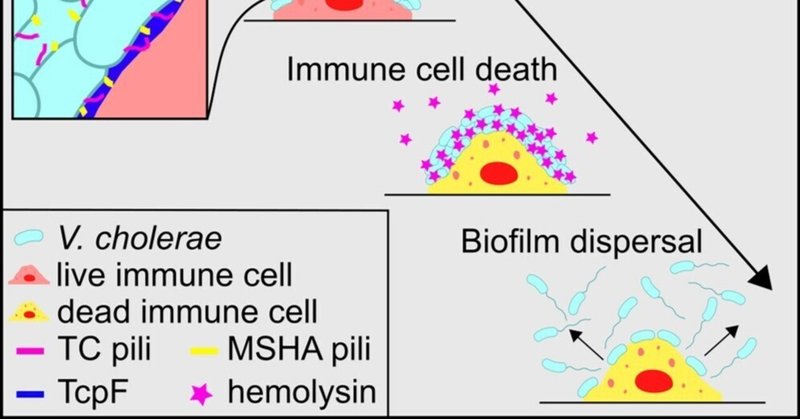
バイオフィルム形成は、抗生物質、バクテリオファージ、ヒト免疫系の白血球などの環境脅威に対する細菌の防御機構として一般に認識されている。今回、我々はヒトの病原体であるビブリオコレラエのバイオフィルム形成が、防御特性だけでなく、異なる免疫細胞をまとめて捕食する攻撃的な特性であることを明らかにした。我々は、V. choleraeが、主にマンノース感受性ヘマグルチニンピリ、毒素制御ピリ、分泌型コロニー形成因子TcpFからなる細胞外マトリックスを用いて真核細胞表面にバイオフィルムを形成し、他の表面上のバイオフィルムのマトリックス組成と異なることを発見した。このバイオフィルムは免疫細胞を包み込み、分泌型ヘモリシンの局所濃度を高くして免疫細胞を死滅させた後、c-di-GMP依存的にバイオフィルムが散逸する。これらの結果から、細菌がバイオフィルム形成を多細胞戦略として利用し、ヒトの免疫細胞を狩る側、細菌を狩られる側という典型的な関係を逆転させていることが明らかになりました。

図解抄録
大きな画像を見る
高解像度画像をダウンロードする
キーワード
宿主-病原体相互作用
コレラ感染症
免疫
IV型ピリ
エンテロイド
オルガノイド
はじめに
環境中の細菌は、バクテリオファージ、他の細菌種、真核生物など、様々な捕食者に頻繁に遭遇する。細菌と捕食者の相互作用は微生物集団を形成し、捕食は細菌の死亡の主な原因として認識されています。
1
,
2
,
3
細菌と捕食者の間の絶え間ない軍拡競争の結果、細菌は殺されるのを避けるために高度な防御機構を進化させてきた。古典的なバクテリオファージによる防御システムや免疫回避システムなどの単細胞戦略に加えて、細菌はバイオフィルムの形成を通じて捕食から身を守るために集団的に行動することができます。
4
,
5
,
6
,
7
,
8
バイオフィルムは、細胞外のバイオフィルムマトリックスがバリアとなり、バクテリオファージや捕食性細菌によるコミュニティへの侵入を防ぐため、細菌の保護避難所として機能することができる。
9
,
10
,
11
また、バイオフィルムは、細菌の認識を妨げたり、炎症反応を抑えることで、細菌がヒト宿主の免疫系から逃れることを可能にします。
12
,
13
,
14
,
15
,
16
また、単細胞と比較してバイオフィルム共同体のサイズが大きくなるため、白血球による貪食に対してバイオフィルムのかすがい抵抗性が生じる。
17
さらに、バイオフィルムに生息する細胞は、抗生物質に対して高い耐性を持つ、
18
,
19
これは、ヒトの感染症におけるバイオフィルムの保護的な性質に新たな一面を与えている。長期感染時にバイオフィルムマトリックス過剰産生変異体が頻繁に出現することや、細菌がバイオフィルムを形成することで免疫細胞の存在に応答できることから明らかなように、ヒト感染時のバイオフィルム形成は細菌にとって重要な適応である。
20
,
21
,
22
,
23
感染症におけるバイオフィルムの保護的側面が文書化されている以上に、バイオフィルムと免疫系との相互作用の範囲とメカニズムは不明である。
バイオフィルムと免疫系の相互作用を調べるために、ヒト白血球と、バイオフィルム研究のモデル生物であり、下痢性疾患コレラの原因菌であるビブリオコレラ(V. cholerae)を用いました。
24
,
25
,
26
ヒトに感染すると、V. choleraeは小腸に定着し、上皮が破壊され、炎症反応が確立される。
27
,
28
,
29
,
30
免疫反応が誘導されると、様々な種類の免疫細胞が感染部位に浸潤し、これらの免疫細胞がコレラ菌と直接相互作用することがある。
27
,
28
,
29
,
30
,
31
,
32
,
33
好中球によるクリアランスを回避するために、V. choleraeはいくつかの毒素を産生することが示されており、これらは共に小腸での細菌の生存に寄与している。
34
コレラ菌が細胞レベルでどのように免疫細胞と相互作用しているのか、またバイオフィルムがこの相互作用に関与しているのかどうかは不明である。
コレラと免疫細胞の相互作用を調べることで、我々は予想外のプロセスを発見した: V.コレラはヒトの免疫細胞表面にバイオフィルムを形成し、免疫細胞を死滅させ、その後バイオフィルムが飛散する。我々は、この相互作用過程の基礎となるマクロファージのメカニズムを明らかにし、バイオフィルムが白血球表面付近に特定の毒素を高濃度に定着させることにより、免疫細胞の殺傷を促進することを示した。したがって、バイオフィルム形成は、免疫細胞からの集団的防御機構であるだけでなく、免疫細胞を集団的に捕食する機構でもあることがわかった。
結果
V. choleraeは異なるヒト免疫細胞上にバイオフィルムを形成する。
自然免疫系および適応免疫系の細胞とコレラ菌との相互作用を調べるため、ヒト血液から好中球、CD4+ T細胞、ナチュラルキラー(NK)細胞、B細胞およびCD14+単球を分離し、CD14+単球をマクロファージに分化させた。これらのヒト免疫細胞に野生型(WT)V.コレラを曝露したところ、細菌細胞が免疫細胞表面に素早く付着することが確認された。その後、細菌は時間の経過とともに蓄積し、厚い多細胞層を形成して免疫細胞表面全体をコロニー化した(図1A)。細菌層に包まれた数時間後、免疫細胞は死滅し、その後、細菌が飛散した。免疫細胞への細菌の蓄積と飛散のダイナミクスは、顕微鏡を用いた、視野内の各免疫細胞の周囲の環状の体積あたりの付着細菌の体積の測定によって反映される(図1B)。我々は、V. choleraeが、初代CD14+単球から分化したマクロファージ(図1Aおよび1B)だけでなく、単球系細胞株THP-1から分化したマクロファージ(図1C)ともこの相互作用過程を示すことを見いだした。興味深いことに、V. コレラは、末梢血由来細胞(図S1A)とTHP-1細胞株(図1D)のいずれにおいても、単球と比較してマクロファージに非常に強い付着力を示し、この相互作用プロセスが特異的であることが示された。マクロファージと単球のプロテオミクス比較により、マクロファージ表面における多数の潜在的な細菌結合パートナーが明らかになり(表S1)、以下のバイオフィルムマトリックス組成のセクションで説明するプルダウン実験によりさらに調査した。V.コレラが様々な免疫細胞に対して同様の相互作用ダイナミクスを示すことから、THP-1由来マクロファージを代表例として、この相互作用プロセスのメカニズムと機能の包括的な解析を行い、初代マクロファージを用いて主要な結果を確認した。また、マクロファージ、腸管単層、V. コレラ菌による共培養感染モデルを用いて、V. コレラ菌とマクロファージとの相互作用を検討しました。
図1V. choleraeがヒト白血球上にバイオフィルムを形成し、その後、免疫細胞死とバイオフィルムの飛散が起こる様子
キャプションの全文を表示する
大きな画像を見る
高解像度画像のダウンロード
図S1コレラ菌によるヒト血液から分離したCD14+単球から分化したマクロファージのコロニー形成と死、および初代CD14+単球への菌の付着(図1、2、3、5を参照
キャプションの全文を表示する
大きな画像を見る
高解像度画像をダウンロードする
マクロファージ上の厚い細菌層が、主に浮遊性細菌の連続付着によるものか、マクロファージ表面での細菌細胞分裂によるものかを明らかにするため、マクロファージ表面の各新しい細菌細胞の起源を、1細胞分解能のライブセル顕微鏡で追跡しました。その結果、浮遊性細菌による細菌の付着率が高い初期期間の後、マクロファージ上の多細胞層構造への蓄積過程を支配するのは、細胞分裂であることがわかった(図1E)。マクロファージへの集積における細菌増殖の重要性は、異なる感染多重度(MOI)での相互作用プロセスの測定によってさらに裏付けられた。一定のマクロファージ数で細菌細胞数を減らして共培養プロセスを開始しても、相互作用の結果は質的に変わらなかったが、マクロファージへの細菌の集積が遅れる結果となった(図S2A)。マクロファージの播種数を変えて細菌数を一定にしても、相互作用の結果は有意に変化しなかった(図S2B)。マクロファージ表面に付着した3次元の細菌構造は、主に細胞の凝集ではなく細菌の細胞分裂によって形成され、複数の細菌細胞層からなることから、細胞を結合する細胞外マトリックス(以下でさらに特徴付ける)の存在を示唆することから、マクロファージの表面でV. choleraeが形成するこれらの構造はバイオフィルムであると考えられる。
図S2V. choleraeバイオフィルム形成動態とマクロファージ死に対する初期細菌数とマクロファージ数の影響、V. cholerae増殖に対するマクロファージ溶解液の影響、単球死に対するV. choleraeの影響、共培養中のV. choleraeとマクロファージの互いの応答、図1および図5を参照
キャプションの全文を表示する
大きな画像を見る
高解像度画像をダウンロードする
私たちが免疫細胞で観察したバイオフィルムの形成過程(図1)は、生物学的表面におけるバイオフィルムのライフサイクルと類似しており、付着、3次元構造への成長、そして分散を含んでいます。私たちは、マクロファージ上のバイオフィルムの機能を明らかにする前に、まず、この3つの発生段階の鍵となるメカニズムを理解し、他の表面上のバイオフィルムの発生と異なるかどうかを確認することを目的としました。
マクロファージへの付着は、鞭毛、運動性、そして2種類のタイプIVピリによって可能になる
V. choleraeがヒトマクロファージと密接な相互作用を確立する仕組みを理解するため、RNAシーケンス(RNA-seq)を用いて、共培養中の細菌のトランスクリプトームを時間分解で測定しました。データ解析の結果、4つの生物学的複製物すべてにおいて、V. choleraeの転写変化が進行しており、1,577個の遺伝子が相互作用過程の過程で有意に発現上昇していた(図S2FおよびS2G)。その中で、鞭毛や、コンピテンスピルス、マンノース感受性ヘマグルチニン(MSHA)ピルス、毒素制御ピルス(TC)である異なるタイプIVピリの生産に関わる遺伝子を特定した(図2A)。さらに、V. choleraeの生物学的および非生物学的表面への付着を促進する他のいくつかのタンパク質をコードする遺伝子も存在する。
35
,
36
は、発現プロファイルに差異があることを示す。今回のトランスクリプトームデータに基づき、これらの因子がV. choleraeのマクロファージ表面への付着に関与していると仮定した。
図2V. コレラのマクロファージ表面への付着は、IV型ピリによって可能となる。
キャプションの全文を表示する
大きな画像を見る
高解像度画像をダウンロードする
しかし、これらの各因子の欠失変異体では、共培養の最初の1時間以内にマクロファージへの付着に必須なのは、鞭毛とMSHA piliのみであることがわかった(図2B)。上皮細胞への付着に重要であることが知られているアドヘシンMam7、GbpA、OmpU、FrhA、
35
は、マクロファージとの結合には必要ない(図2B)。ネイティブプロモーターの制御下でmshAまたはflaAを異所性発現させると、細菌の付着が回復した。また、フェナミルの添加により旗モーターを停止させると、付着が抑制されたが、その効果は旗欠失変異体ほど強くなかった。このことから、マクロファージへの付着には、旗の構造と旗の回転によって可能になる細胞運動の両方が寄与していると考えられる。THP-1由来マクロファージで得られたこれらの知見は、初代CD14+由来マクロファージでも当てはまることを確認した(図S1B)。驚くべきことに、長時間の共培養により、ΔmshA細胞は、主要なピリンTcpAと分泌された拡散性タンパク質TcpFを有するTCピルスの存在によって、数時間後にマクロファージにも付着できることが明らかになった(図2C)。これは、tcpオペロンの発現ダイナミクス(図2A)と一致する。これに対応して、共培養中にTC piliとTcpFを早く発現させると、マクロファージへの付着が早くなる(図S3A)。したがって、V. choleraeのマクロファージ表面への付着には、鞭毛とMSHA pili、または鞭毛とTC piliとTcpFのいずれかが必要であることがわかった。
図S3ΔmshA細胞の付着とバイオフィルム形成に対するTC piliとTcpFの影響、マクロファージ上に形成された成熟V. cholerae WTバイオフィルム内のRbmA、Bap1、RbmCの局在、vps遺伝子の転写、図2および3を参照。
キャプションを表示する
大きな画像を見る
高解像度画像をダウンロードする
マクロファージ上のバイオフィルムのマトリックス組成は、他の表面上のマトリックスと異なる
バイオフィルムが複数の細胞層を持つ3D構造に成長するには、細胞同士の結合が必要であり、その際、一般的に細胞外マトリックスが関与する。生物表面では、コレラ菌バイオフィルムのマトリックス成分として、Bap1、RbmA、RbmCというタンパク質とビブリオポリサッカライド(VPS)が確認されている。
37
免疫蛍光法と転写レポーターを用いて、これらのマトリックス成分がマクロファージ上のバイオフィルムにも存在することを確認したが、そのレベルは低かった(図S3C-S3F)。マトリックスタンパク質は、バイオフィルムの外縁部に位置する少数の細菌細胞のみを包み、vps転写レポーターの低い蛍光シグナルはバイオフィルム全体に存在した。しかし、マクロファージ上では、この変異体が生物学的表面でのバイオフィルム形成が不可能であるにもかかわらず、これらの構成要素をすべて欠く細菌が、WTバイオフィルムと同様のバイオフィルムを形成できることが確認された(図3A)。
37
図3マクロファージ上に形成されたバイオフィルムの細胞外マトリックスは、MSHA pili、TC pili、TcpFからなり、これらは異なるバイオフィルム機能を提供する
キャプションの全文を表示する
大きな画像を見る
高解像度画像をダウンロードする
マクロファージ上でのバイオフィルムの増殖には、これらのよく知られたマトリックス成分に頼るのではなく、2種類のタイプIVピリのうち少なくとも1つの産生が必要であることを見いだしました: MSHAピリまたはTCピリである(図3AおよびS1C)。さらに、TCピリを介したバイオフィルムの成長は、TcpFの存在に依存している(図3AおよびS1C)。上記のように、これら2つの繊毛構造は、コレラ菌のマクロファージへの付着に必要である(図2)。しかし、繊毛は細胞外構造でもあり、細胞外マトリックス成分として機能する可能性がある。MSHA piliとTC piliが細胞外マトリックスの一部であるかどうかを調べるために、免疫蛍光法でMSHA pili、TC pili、TcpFを直接可視化して定量化したところ、それらがマクロファージ上のバイオフィルムに確かに存在することがわかった(図3B〜3D)。MSHA piliはバイオフィルム全体に一様に局在していたが(図3B)、TC piliと分泌タンパク質TcpFはマクロファージ表面付近に多く見られた(図3Cおよび図3D)。したがって、マクロファージ上のV. choleraeバイオフィルム・マトリックスは、他の表面上のバイオフィルム・マトリックスとは異なる構成要素に依存していると考えられる。
TC piliとTcpFの相互作用を調べるために、TC piliを過剰発現する細菌株に外因的に供給した精製TcpFをΔmshA ΔtcpFのバックグラウンドで実験を行った。tcpオペロンの過剰発現は、転写活性化因子ToxTを過剰発現させることで実現した。これらの実験により、マクロファージ上での細菌バイオフィルム形成には、TC piliの存在だけでは不十分であることが明らかになった(図S3B)。しかし、精製TcpFを外因的に添加すると、マクロファージ上でのこの細菌株のバイオフィルム形成は、TcpFの濃度依存的に増加した。これらの結果から、V. choleraeはTC piliを使ってタンパク質TcpFに結合し、TcpFは真核生物の表面と直接相互作用することが示唆された(図3D)。これは、図2C、3A、S3A、S3Bに示したように、TC piliとTcpFが一緒に作用するという我々の観察を説明できる。
マクロファージ表面におけるバイオフィルム基質成分の結合パートナーを明らかにするために、マクロファージと単球のプロテオミクスによる存在量の差分解析(表S1)を行い、さらにMshAまたはTcpFを餌、マクロファージ溶解液を餌としてプルダウン実験を行い、液体クロマトグラフィー質量分析(LC-MS)分析を行いました。MSHAピリの場合、マクロファージ表面で22の潜在的結合パートナー(単球では見られない)を同定し、そのうち12は糖タンパク質であった。TcpFの場合、マクロファージ表面で36の潜在的タンパク質結合パートナーを見つけた(表S2およびS3)。
MSHAピリとTCピリがマクロファージ上のバイオフィルムマトリックスの一部であることが判明したことから、これらのピリはバイオフィルムに異なる特性を付与している可能性がある。マクロファージ上のバイオフィルムの機械的安定性を解析した結果、TC piliの存在により、V. choleraeバイオフィルムは機械的ストレスによりよく耐えられることがわかった(図3E)。また、マクロファージ上のバイオフィルム構造を解析した結果、TC piliはマクロファージ上のV. choleraeバイオフィルム内の高い細胞間配列の確立に寄与していることが明らかになった(図3F)。マクロファージへの付着(図2C、S3A、S3B)およびマクロファージ上でのバイオフィルム形成(図2C、3A)には、MSHA piliまたはTC piliのいずれか1種類のピラスの生産ですでに十分であることから、今回のデータは、両方のタイプ IV piliが細菌-細菌相互作用および細菌-免疫細胞相互作用に関わっていることを示しています。
マクロファージ上でのバイオフィルム拡散は、TC型絨毛とc-di-GMPの減少に依存する
バイオフィルムの分散は、マクロファージ上でのバイオフィルム形成の最終段階であり、マクロファージがバイオフィルム・マトリックスから離脱することが必要である。マクロファージからバイオフィルムが離脱する際の重要なメカニズムを理解するために、我々は離脱時のマトリックス成分の違いについて調べた。その結果、TC piliの存在がバイオフィルムの安定性と構造に影響を与えるだけでなく(図3Eと3F)、TC piliがバイオフィルムの飛散範囲を決定することも確認した。TCピリを欠損したバイオフィルム(ΔtcpA)は親株よりも強い分散性を示し、tcpオペロンを過剰発現させたバイオフィルムは分散性を低下させた(図4A)。MSHAピリを欠くバイオフィルムは親株と同様の分散能力を示した(図4A)。これらのデータから、TCピリ産生の制御がバイオフィルムの分散に必須であることが示唆された。
図4バイオフィルムの分散性は、細胞内のc-di-GMPレベルとTC piliの存在によって決定される
キャプションを表示する
大きな画像を見る
高解像度画像をダウンロードする
この仮説と一致して、V. choleraeとマクロファージの相互作用の間のトランスクリプトームデータは、tcpオペロンが分散中に実際にダウンレギュレーションされることを示している(図4B)。ヒトや動物モデルにおけるV. cholerae感染の後期にも、tcpはダウンレギュレーションされている、
38
,
39
これは、上皮細胞からの分散だけでなく
39
のみならず、免疫細胞からも分散できる可能性がある。mshオペロンの転写レベルは分散に影響されない(図4B)。これは、MSHAの欠如がバイオフィルムの分散に影響しないという知見と一致する(図4A)。
生物表面で増殖したバイオフィルムでは、第2メッセンジャーシグナル分子c-di-GMPの細胞内レベルの低下が、いくつかの種でバイオフィルム形成と分散との間の移行を制御することが知られている。
40
そこで我々は、c-di-GMPレベルの低下が、マクロファージ表面からのV. コレラ菌バイオフィルムの飛散を引き起こす可能性があると推測した。そこで、不安定蛍光タンパク質レポーターを用いたc-di-GMPレベルのレポーターを用いて、バイオフィルムの成長と分散におけるc-di-GMPレベルを測定することにより、この考えを検証した。この測定により、バイオフィルムの分散はc-di-GMPレベルの低下と一致することが明らかになった(図4C)。c-di-GMPレベルの段階的な低下がバイオフィルムの分散を引き起こすかどうかを調べるために、マクロファージ上でバイオフィルムを自然に分散する直前まで成長させ、2種類のc-di-GMP分解ホスホジエステラーゼ(RocSとCdgJ)の発現を誘導しました。その結果、2つのホスホジエステラーゼの過剰発現後、バイオフィルムが直接分散したのに対し、空ベクターコントロールのバイオフィルムは成長を続け、後から分散することがわかった(図4D)。図4の結果から、マクロファージ表面では、バイオフィルムの分散にはTCピリの制御が必要であり、分散は細胞内のc-di-GMPレベルによって制御されることが示された。
マクロファージ周辺のバイオフィルムは、毒素の運搬を増加させることでマクロファージによる殺傷を促進する
マクロファージへの細菌の付着、バイオフィルムの形成、およびバイオフィルムの分散に関する主要なプロセスを上記のように特定した後、我々はV. choleraeがマクロファージ上のバイオフィルム形成から得る機能的利益を調べることに着手した。我々はまず、厚いバイオフィルムとそのピルスベースのマトリックスが、バイオフィルムを介したサイトカインの拡散を制限することによって、炎症シグナルを阻害するのではないかという仮説を立てた。しかし、分泌されたサイトカインレベルを測定したところ、マクロファージ上のV. コレラ菌バイオフィルムは、これらのシグナルを環境中に放出することを妨げないことがわかった(図S2H)。
あるいは、マクロファージ周囲の厚いバイオフィルムがマクロファージの殺傷に寄与しているのではないかという仮説を立てた(図1Aおよび図1C)。この仮説を裏付けるように、マクロファージ上のバイオフィルムの厚さは、マクロファージの死滅と相関していることがわかりました(図5A)。そこで、V. choleraeがどのようにマクロファージを死滅させるのか、そしてこのプロセスにバイオフィルムがどのように関与しているのかを調査しました。
図5細菌バイオフィルムによるマクロファージの封じ込めはマクロファージの細胞死を増加させる
キャプションの全文を表示する
大きな画像を見る
高解像度画像をダウンロードする
V. choleraeは多くの仮説および既知の毒素をコードしており、そのうちのいくつかはマクロファージとの共培養中に転写の差分を示す(図5B)。マクロファージ死滅への寄与を調べるため、潜在的な殺傷因子をそれぞれ欠失させた変異体を作製した。マクロファージに7時間曝露した結果、毒素HlyAを欠損したV. choleraeは、WT菌と比較してマクロファージ死滅が有意に少ないことがわかった。他の毒素の欠失は、WT菌と比較して有意な差は生じなかった(図5C)。ヘモリシンHlyAは、単量体で分泌されるが、宿主細胞膜でオリゴマー化し、細胞溶解を誘導する孔形成性毒素である。
41
,
42
,
43
,
44
HlyAを過剰発現させると、表現型が元に戻り、マクロファージ死が増加した(図5C)。また、ヒト血液から分離した単球由来のマクロファージに対しても、HlyAが細胞死を誘導することが確認された(図S1D)。ΔhlyA背景株でさらに毒素コード遺伝子を欠失させても、ΔhlyA細胞と比較してマクロファージ死滅率に大きな変化はなかった(図S4)。また、MOIの増加によりマクロファージ死滅が増加することが確認された(図S2C)。これらの結果から、V. choleraeはHlyAをマクロファージを殺すための主要な毒素として使用していることが立証された。
図S4マクロファージ死滅の主な原因である毒素HlyA(図5を参照
キャプションを表示する
大きな画像を見る
高解像度画像をダウンロードする
マクロファージ周囲のバイオフィルムがHlyA依存性のマクロファージ死滅に寄与しているかどうかを調べるため、マクロファージにバイオフィルムを形成できない細菌(ΔmshA ΔtcpA)を曝露し、顕微鏡検査とヨウ化プロピジウム染色を用いてマクロファージ死の発生をモニターしました。バイオフィルム形成不全の細菌(ΔmshA ΔtcpA)は、WT細菌よりも有意に少ないマクロファージ死を誘導した(図5D)。マクロファージと比較して細菌付着の減少が観察された単球については(図1D)、細胞死の減少も観察された(図S2E)。ヘモリシンの追加欠失(ΔmshA ΔtcpA ΔhlyA)は、マクロファージ死のさらなる減少を引き起こし(図5D)、ヘモリシンとバイオフィルム構造の両方がマクロファージの殺傷に寄与することが示されました。また、ヒト血液から分離した単球由来のマクロファージを用いて、HlyAによるマクロファージ死滅にバイオフィルムが重要であることを確認した(図S1E)。
バイオフィルムの形成とヘモリシンの生成は、いずれもマクロファージの生存率に影響を与えるため、コレラ菌とマクロファージの相互作用におけるこれら2つの要因が、細菌との遭遇に対するマクロファージの反応にどのように影響するかを理解しようと考えた。マクロファージをWT株、バイオフィルム欠損株、ヘモリシン欠損株のいずれかに曝露した後、RNA-seqを行ったところ、これらのV. コレラ株はそれぞれ、未処理のマクロファージと比較して、マクロファージの転写産物に同様の変化を引き起こすことがわかった(図S2I)。したがって、バイオフィルムの成長とヘモリシン産生は、両特徴がマクロファージの死に大きく寄与しているにもかかわらず、V. コレラ菌との遭遇時に観察されるマクロファージの転写反応に寄与していないことがわかる。
バイオフィルムがHlyA依存性のマクロファージ死滅にどのように寄与しているかを理解するために、免疫蛍光法を用いて分泌されたHlyAの局在を決定した。マクロファージ上にV. コレラのバイオフィルムが存在すると、HlyAはマクロファージ表面とその近傍に集積する(図5E)。一方、マクロファージがバイオフィルム欠損菌に曝されると、分泌されたHlyAの量は著しく減少した(図5E)。したがって、マクロファージ上のバイオフィルム形成は、マクロファージ表面付近に分泌された拡散性毒素HlyAを高濃度に定着させることにより、V. choleraeのマクロファージを殺す能力を高める。
ヘモリシンは宿主細胞膜に孔を形成し、マクロファージの細胞内容物を無秩序に排出するため、V. コレラがマクロファージの細胞成分を利用して増殖できるかどうかを調べました。マクロファージから生成した溶解液の存在下で細菌の増殖をモニタリングしたところ、V. choleraeは溶解液の濃度依存的に増殖の促進を示した(図S2D)。したがって、マクロファージの殺傷はV. choleraeの捕食特性である。
V. choleraeは、ヒト腸管単層膜を突破した後、マクロファージ上にバイオフィルムを形成する。
ヒト小腸におけるV. choleraeの病原性は、腸管上皮のコロニー形成に依存する。
45
このコロニー形成は、感染部位への免疫細胞の浸潤と上皮バリアの破壊をもたらし、細菌と免疫細胞との相互作用を可能にする。
27
,
28
,
29
,
30
,
31
,
32
,
33
,
46
V. コレラが上皮に定着・突破した後、免疫細胞上にバイオフィルムを形成できるかどうかを調べるため、ヒト小腸オルガノイド(腸管)由来の上皮単層とマクロファージによる共培養モデルを確立した。この共培養モデルでは、透過性インサート上で分化させた腸管単層の基底側にマクロファージを培養した(図6A)。その後、腸管単層の先端側に細菌を加えることで感染を開始した(図6A)。細菌、上皮バリア、マクロファージの相互作用は、タイムラプス共焦点顕微鏡でモニターした。感染中、V. cholerae WTによる腸管単層のコロニー形成(図6A)と上皮バリアの完全性の崩壊が進行していることが観察された。上皮バリアが損なわれた後、細菌は基底側に到達し、図1に記載したプロセスと同様に、マクロファージ上にバイオフィルムを形成しました(図6A)。THP-1由来マクロファージの代わりに、ヒト血液から分離した単球由来のマクロファージを用いても、同様の感染の進行が見られた(図S5A)。異なるヒトドナー由来の腸管を使用すると、マクロファージ上での相互作用のダイナミクスとバイオフィルムの形成が類似していた(図6AおよびS6A)。
図6V. cholerae細胞がヒト腸管上皮バリアを破り、その後マクロファージの下にバイオフィルムを形成する様子
キャプションの全文を表示する
大きな画像を見る
高解像度画像をダウンロードする
図S5異なるMOI、異なる細菌変異体、および異なるマクロファージに対する腸管マクロファージ共培養モデルのV. choleraeによる感染(図6を参照
キャプションの全文を表示する
大きな画像を見る
高解像度画像をダウンロードする
コレラ菌感染ウサギモデルにおける小腸の細菌負荷の以前の測定では、内腔上皮表面積0.3cm2あたり105~109個の細菌細胞が存在することが示された。
47
これらの測定結果にヒントを得て、共培養モデルにおいて、腸管上皮表面0.33 cm2あたり3.2×105~6.4×106個の範囲の細菌負荷を試験しました(図S5B)。上皮単層を異なる細胞数のV. cholerae WTにさらすと、初期MOIを減少させると腸管単層の破壊が遅れることが示されたが、それでもすべての初期MOIが最終的に上皮バリアを破壊し、その下のマクロファージにバイオフィルム形成をもたらした(図S5B)。
腸管単層膜を破壊した後にマクロファージ上に形成されたバイオフィルムは、免疫蛍光法で明らかになったように、MSHA pili、TC pili、および分泌タンパク質TcpFからなるマトリックスを有していた(図6B)。また、V. choleraeの変異株をこの共培養モデルで解析したところ、MSHAピリ(ΔmshA)、TCピリ(ΔtcpA)、またはその両方を欠く株(ΔmshA ΔtcpA)は上皮細胞に付着して上皮のバリアを破ることがわかった、 しかし、マクロファージ上でのバイオフィルム形成には、2種類のIV型繊毛のうち1つが必要であった(図6C-6E、S5F、S6)。これは、図2および図3に示した所見と一致する。鞭毛を欠く細菌(ΔflaA)は腸管単層の基底側に到達せず、その結果、その下のマクロファージにバイオフィルムを形成しなかった(図6FおよびS6I)。コレラ毒素(ΔctxAB)、ヘモリシン(ΔhlyA)、あるいは生物表面上のバイオフィルムに採用されている正規のマトリックス成分(Δbap1 ΔrbmA ΔrbmC ΔvpsL)を欠く細胞は、それでも腸管単層を破壊でき、マクロファージ上にバイオフィルムを形成した(図S5C〜S5E、S6D〜S6F)。いずれの細菌変異体でも観察された相互作用過程は、ヒト腸管ドナーに依存しなかった(図S5およびS6)。
図S6異なるV. cholerae株とTHP-1マクロファージおよびドナー#2からのヒト腸管単分子膜との共培養結果、図6を参照。
キャプションの全文を表示する
大きな画像を見る
高解像度画像をダウンロードする
腸管由来のヒト腸管モデルの感染症から、コレラ菌は腸管上皮に定着し、上皮バリアを破壊した後、ピルスベースのマトリックスでマクロファージ上にバイオフィルムを形成できることが示されました。マクロファージ上でのバイオフィルム形成はMSHA piliとTC piliに強く依存するが、これらのpiliがない場合でも細菌は腸管上皮に付着し破壊することができる。
考察
本研究により、V. choleraeは様々なヒト免疫細胞の表面でバイオフィルムを形成し、免疫細胞を包み込むことが明らかになった。我々は、腸管感染モデルにおいて、上皮バリアを突破した後に免疫細胞へのバイオフィルム形成が起こることを見出した。さらに、好中球、NK細胞、CD4+ T細胞、B細胞、マクロファージでこの挙動が観察されたが、単球では観察されなかったことから、免疫細胞上のバイオフィルム形成は広範囲に及ぶプロセスであり、標的がないプロセスではない。私たちの分析により、これらのバイオフィルムの2つの基本的な側面が明らかになりました。すなわち、免疫細胞上のバイオフィルム発生の異なる段階を制御するメカニズム、そして免疫細胞上のバイオフィルムの機能です。
マクロファージ上でのバイオフィルムの発達は、細菌の付着、マトリックスの生成、3次元的な成長、そしてバイオフィルムの分散を含み、これは生物学的表面でのバイオフィルムの発達と定性的に類似している。しかし、我々はマクロファージ上でのバイオフィルム形成に関わるメカニズムが、他の表面上でのそれとは異なることを明らかにした。
ガラス、寒天、キチン上のV. choleraeのバイオフィルムマトリックスは、主に多糖類VPSとタンパク質RbmA、RbmC、Bap1から構成されています。
37
腸管上皮細胞上では、V. choleraeはTC piliによって細胞同士が結合したマイクロアグリゲートを形成することができる。
48
懸濁液中では、V. choleraeはTCピラスとDNA取り込みピラスを用いて多細胞の塊に自動凝集することができる、
49
,
50
これは、細菌の増殖を必要としないプロセスである。一方、マクロファージ上で増殖したバイオフィルムは、MSHAピリ、TPピリ、TcpFからなるマトリックスを形成し、正規のマトリックス成分であるVPS、RbmA、RbmC、Bap1は必要ないことが示された。我々は、V. choleraeの急性感染症において、IV型絨毛がバイオフィルムのマトリックス成分として有用であると推測している。なぜなら、これらの絨毛は素早く組み立てられたり引っ込められたりして、バイオフィルムを素早く再編成し、分散させることができる可能性があるからだ。異なる環境下でバイオフィルムに異なるマトリックス成分を利用することは、細菌の適応として興味深い。
バイオフィルムのマトリックス組成の違いだけでなく、マクロファージでは他の表面と比較して、表面付着のメカニズムも異なっている。ガラスやキチン表面への付着には、鞭毛やMSHAピリが関与する。
51
,
52
上皮細胞への付着には、TC piliとTcpF、そしてアドヘシンGbpA、FrhA、OmpU、Mam7が必要である。
48
,
53
,
54
,
55
,
56
,
57
,
58
一方、V. choleraeのマクロファージへの付着には、アドヘシンGbpA、FrhA、OmpU、Mam7は無関係であることがわかった。その代わりに、マクロファージへの付着には、鞭毛、細菌の運動性、およびMSHA piliまたはTC piliの2つのタイプIV piliのうち少なくとも1つと分泌型コロニー形成因子TcpFが必要となる。両ピリを持たない菌株(ΔmshA ΔtcpA)はマクロファージに付着できないが、上皮細胞には付着することができる。
マクロファージ上のバイオフィルムの機能は、他の表面上のバイオフィルムの機能に関する一般的な解釈とは異なっているのも事実である。バイオフィルムの形成は、ストレスに対する耐性を高め、細菌集団への捕食者の接近を抑制することで、構成する細胞を保護することが多い。
8
,
10
,
19
,
20
,
37
,
59
一方、マクロファージ上にバイオフィルムを形成すると、マクロファージの周囲に分泌毒素HlyAが局所的に高濃度に定着し、マクロファージの死滅が促進されることを明らかにした。興味深いことに、V. コレラは、マウスモデルにおいて、毒素依存的に好中球による駆除を回避することが示されている、
34
このことから、毒素の効率的な送達には、これらの免疫細胞上でのバイオフィルム形成が重要である可能性が示唆された。緑膿菌(P. aeruginosa)については、プランクトン細胞やバイオフィルム内の細胞から分泌されるラムノリピッドが免疫細胞を殺すことが、特に免疫細胞が成熟した緑膿菌バイオフィルムに向かって積極的に移動するときに観察されたことが以前にある。
60
,
61
,
62
一方、本研究では、V. choleraeが免疫細胞上やその周囲にバイオフィルムを形成することで免疫細胞との直接的な相互作用を確立し、最終的に免疫細胞を死滅させることが示された。したがって、免疫細胞上およびその周囲でのV. choleraeのバイオフィルム形成と、バイオフィルムに依存した免疫細胞への毒素送達の強化は、これまで報告されてきた細菌と免疫細胞との相互作用過程とは定性的には異なる。多くの細菌性病原体が感染時にバイオフィルムを形成するが、免疫細胞上でのバイオフィルム形成が他の病原体の間でどの程度普及しているのか、また免疫細胞上のバイオフィルムが毒素の運搬や捕食の促進以外にも新たな機能を持つのかについては、まだ調査する必要がある。
ヒトにおけるV. コレラ菌の感染は、炎症反応を引き起こし、その結果、その後の感染に対する免疫が長期にわたって持続する。
29
,
63
さらに、コレラ患者の血液中には、抗MSHA抗体が検出される、
64
これは、MSHA piliがV. choleraeの病原に関与していることが知られていなかったため、これまで謎であった。実際、V. choleraeは、腸管コロニー形成の初期段階において、MSHA piliに結合する粘膜免疫因子である分泌型IgAによる認識を避けるために、MSHA piliの生産を抑制する必要があり、それによって細菌の粘液層への侵入とその後の上皮細胞への結合を防止する。
65
,
66
V. choleraeと上皮細胞との相互作用研究およびV. cholerae感染の様々な動物モデルにより、TC piliとTcpFが宿主のコロニー形成時に一緒に作用する因子として同定されたが、MSHA piliの欠如はコロニー形成能に影響を与えない。
48
,
53
,
54
,
67
MSHAピリは、免疫細胞への付着や免疫細胞上のバイオフィルムマトリックスの一部として重要な役割を果たすことが明らかになり、V. cholerae感染時に抗MSHA抗体が存在することが説明できるようになった。さらに、今回の発見は、TC piliとTcpFが免疫細胞との相互作用に関与していることから、感染症におけるTC piliの重要性を示す新たな一面を提供した。
結論として、V. choleraeは、細菌が集団で行動して感染部位の免疫細胞を殺し、周囲の食細胞数を減少させるメカニズムを開発した。その結果、V. choleraeは、他の細菌に見られるような、免疫細胞から逃れるための避難場所としてバイオフィルム形成を利用することはない。その代わりに、V. choleraeはバイオフィルム形成を免疫細胞を捕食するための攻撃的な多細胞戦略として用いている。
研究の限界
今回の結果は、ヒトにおけるV. cholerae感染の既知のプロセスと一致しており、ヒト血液から分離した初代好中球、NK細胞、CD4+ T細胞、B細胞、および初代マクロファージ上でV. choleraeバイオフィルム形成を観察し、現実的なMOIで腸管単層突破後にマクロファージ上にバイオフィルムを形成することを観察したが、ヒトにおける感染時に免疫細胞上でのバイオフィルム形成が起こることを直接検証することは不可能であった。
V.コレラはヒトの病原体であり、通常他の動物に感染することはありませんが、ヒトの感染症の特徴の多くを反映した動物モデルが開発されています。マウスやウサギをベースに、動物に薬剤を投与して感染させます。
68
,
69
コレラ菌に暴露される前に、マウスやウサギを用いた動物モデルが開発されている。マウスやウサギの免疫系はヒトとは異なるが、
70
コレラ菌感染の後期を動物モデルで研究することは有益であるが、そのような研究は、ヒトのコレラ菌感染における免疫細胞上のバイオフィルムの役割を直接検証することにはならない。
STAR★メソッド
主要リソース表
REAGENTまたはRESOURCEIDENTIFIER抗体ヤギ抗ウサギIgG、Alexa Fluor 647ThermoFisherCat#A-21244; RRID: AB_2535812ヤギ抗マウスIgG、Alexa Fluor 647ThermoFisherCat#A-21235; RRID: AB_2535804Penta His Alexa Fluor ConjugateQiagenCat#35370HA Tag Monoclonal antibody, Alexa Fluor 647ThermoFisherCat#26183-A647; RRID: AB_2610626Cholera Hemolysin Monoclonal AntibodyThermoFisherCat#MA1-83520; RRID: AB_936752Rabbit anti-MshAFloyd et al.
71
N/ARabbit anti-TcpASun et al.
72
N/AB細菌およびウイルス株表S4本論文N/AC化学物質、ペプチドおよび組換えタンパク質LB培地(Luria/Miller)Carl RothCat# X968.3LB agar(Luria/Miller)Cat# X969.3Gentamicin sulfateCarl RothCat#0233.4Gentamicin sulfateITW ReagentsCat#A1492Kanamycin sulfateCarl RothCat#T832. 3Polymyxin B sulfateAppliChemCat#A0890,0001Streptomycin sulfateCarl RothCat#HP66.3Gibco DMEMThermoFisherCat#11880028GlutaMAX SupplementThermoFisherCat#35050038Gibco Fetal bovine serumThermoFisherCat#10500064X-GalCarl RothCat#2315.4 3IPGGoldbioCat#I2481C5L(+)-ArabinoseCarl RothCat#5118.2Dimethyl sulfoxide(DMSO)Carl RothCat#7820. 4LymphoprepSTEMCELL TechnologiesCat#07801CD14 Micro-Beads humanMilteny BiotecCat#130-050-201CD4 Micro-Beads humanMilteny BiotecCat#130-045-101CD19 Micro-Beads humanMilteny BiotecCat#130-050-. 301CD56 Micro-Beads humanMilteny BiotecCat#130-050-401TheraPEAK X-VIVO™-15 Serum-free Hematopoietic Cell MediumLonzaCat#BEBP02-061QRecombinant Human GM-CSFPeprotechCat#300-03Phorbol 12-myristate 13-. acetate(PMA)Sigma-AldrichCat#P1585-1MGCorning MatrigelCorningCat#356231ジブコAdvanced DMEM/F-12ThermoFisherCat#12634010 ジブコHEPESThermoFisherCat#15630106ジブコB-27 SupplementThermoFisherCat#12587010 ジブコN-. 2 SupplementThermoFisherCat#17502001N-acetyl-L-cysteineSigma-AldrichCat#A9165NicotinamideSigma-AldrichCat#N0636Human EGFSigmal-AldrichCat#E9644Recombinant Human RSPO1-FcSUN Bioscience、 EPFLN/ARecombinant Human Noggin-FcSUN Bioscience、 EPFLN/ASB202190Sigma-AldrichCat#S7067A 83-01Sigma-AldrichCat#SML0788Prostaglandin E2Lucerna-ChemCat#HY-101952Human [Leu15]-gastrin ISigma-AldrichCat#G9145Wnt-... surrogate-Fc Fusion Recombinant ProteinThermoFisherCat#PHG0401Amphotericin BThermoFisherCat#15290018ThiazovivinSigma-AldrichCat#SML1045CHIR99021Sigma- AldrichCat#361571TrypLE Express EnzymeThermoFisherCat#12605010SmBM Basal MediumLonzaCat#CC-3181SingleQuots supplementsLonzaCat#CC-4149Rat-tail collagen type ISigma-. AldrichCat#C3867CellTracker Deep Red DyeThermoFisherCat#C34565CellTracker Red CMTPX DyeThermoFisherCat#C34552Gibco HBSSThermoFisherCat#14175053Propidium iodideCarl RothCat#CN74. 3CellMask Plasma Membrane StainThermoFisherCat#10046Sylgard 184 Silicone Elastomer KitDOWCat# 1673921FormaldehydeSigma-AldrichCat#F8775-25MLBovine serum albuminSigma-. AldrichCat#A3059-100GRNAlaterQiagenCat#76106TURBO DNaseThermoFisherCat#AM2238トリクロロ酢酸Sigma-AldrichCat#T6399Critical commercial assayIL- 1 beta human uncoated ELISA KitThermoFisherCat#88-7261-22IL-8 human uncoated ELISA KitThermoFisherCat#88-8086-22TNF alpha human uncoated ELISA KitThermoFisherCat#88-7346-22mirVana miRNA Isolation KitThermoFisherCat#AM1560デポデータ生・解析トランスクリプトームデータGene Expression OmnibusGEO: GSE184077, GSE184078質量分析データProteomeXchange ConsortiumPRIDE: PXD039577, PXD039579実験モデル: 細胞株THP-1ATCCATCC TIB-202ヒト腸管上皮細胞LonzaCat#CC-2931; Lot 0000751359ヒト腸管筋線維芽細胞LonzaCat#CC-2902; Lot 00006621323dGRO Duodenum Intestinal OrganoidsMerckCat#SCC324オリゴヌクレオチド表S6本論文N/ARecombinant DNAT表S5本論文N/ASoftware and algorithmsCustom image analysis code本論文https: 本論文では、//doi. org/10.5281/zenodo.7900472 Nikon NIS-Elements ARNikonVersion 4.51.01MATLABMathWorksVersion R2017bPrismGraphPad SoftwareVersion 9.3.1Micro-ManagerEdelstein et al.
73
バージョン2.0STARDobin et al.
74
Version 2.5.3aSpectronautBiognosysVersion 15.7.220308.50606MSstatsChoi et al.
75
バージョン4.2.0
新しいタブで表を開く
リソースの有無
リードの連絡先
詳細情報、リソース、試薬のリクエストは、リードコンタクトであるKnut Drescher (knut.drescher@unibas.ch)までお願いします。
材料の入手方法
菌株、細胞株、プラスミドのリクエストは、リードコンタクトにお願いします。
実験モデルおよび被験者の詳細
菌の増殖条件
本研究で使用したすべてのV. cholerae株は、野生型V. cholerae O1 biovar El Tor株C6706(KDV201)から派生したものである。通常、細菌は適切な抗生物質を添加したLB培地(Luria/Miller)で、37℃、250rpmの振盪で培養した。
V.コレラと免疫細胞との相互作用を調べる実験のために、細菌はまず、ゲンタマイシン(最終濃度30μg mL-1)を添加したLB培地で指数関数成長期まで培養した。その後、細胞を1:500に逆希釈し、10%(v/v)のウシ胎児血清(FCS; Gibco cat. no. 10500064)とゲンタマイシンを添加したGlutaMax (Gibco cat. no. 35050038)を加えたDulbecco変法イーグル培地 (DMEM; Gibco cat. 11880028) で静止成長相まで再増強した。
細胞培養と免疫細胞の分化
白血球は、ドイツ、ギーセンの輸血医学・血液療法センターから健康なドナー(使用前に非特定)から入手した血液バフィーコートから単離した。すべてのドナーは、科学的研究のために血液サンプルを使用することに同意した。白血球は、Lymphoprep密度勾配培地(STEMCELL Technologiesのカタログ番号07801)を用いて単離し、さらに製造者のプロトコールに従ってMilteny MACS MicroBeads(CD14、CD4、CD19、CD56)を用いて単球、CD4+ T細胞、B細胞およびNK細胞に分離した。単離した単球、CD4+ T細胞、B細胞、NK細胞は、V.コレラ-免疫細胞相互作用研究の項で述べたように、V.コレラ-免疫細胞相互作用研究に直ちに使用した。
マクロファージを得るために、8×104個の新鮮な単球(前項のように単離された)を、5%(v/v)FCSを補充した200μL X-Vivo 15培地(Lonza cat.no. BEBP02-061Q) に懸濁した。その後、96ウェルガラスボトムプレート(Greiner Bio-One cat.no.655892)の個々のウェルに播種し、15ng mL-1ヒト組み換えGM-CSF(Preprotech cat.no.300-03) で37℃、加湿5%CO2インキュベーター内で7日間処理してからコレラ菌との相互作用試験を実施(次のセクションに記述)。4日間の分化の後、15 ng mL-1ヒト組換えGM-CSFとともに、さらに50μLの新鮮な培地をウェルに添加した。マクロファージが腸管単層を含む相互作用試験に使用される場合、8×104個の新鮮な単球が15ng mL-1ヒト組換えGM-CSFを含む50μLの培地に懸濁し、12ウェルガラス底プレート(セルビスの猫番号P12-1.5H-N)の個々のウェル中央に播種した。翌日、950μLの培地を追加した。分化の4日後、さらに200μLの新鮮な培地と15ng mL-1のヒト組換えGM-CSFをウェルに添加した。
好中球を得るために、十分な説明を受け、本研究へのサンプル提供に同意した健康なヒトドナーから血液サンプルを採取した。提供された血液は、Philipps-Universität Marburgの倫理規定に従って取り扱われた(許可証:AZ 143/20)。好中球は、Ohらによって発表されたプロトコルに従って全血から分離された。
77
により全血から分離し、コレラ菌-免疫細胞相互作用研究の項で述べたように、コレラ菌との相互作用研究に直ちに使用した。
上記の初代細胞に加えて、ヒト単球細胞株THP-1(ATCC TIB-202)をこの研究に使用した。THP-1細胞は、10%(v/v)FCSおよびゲンタマイシン(最終濃度30μg mL-1)を補充したGlutaMax入りDMEM培地で、T75またはT175フラスコ中、加湿した37℃、5%CO2インキュベーターで培養した。THP-1単球をマクロファージに分化させるために、200μLの新鮮な培地に懸濁した2.5×104個の細胞を96ウェルガラスボトムプレートの個々のウェルに播き、20nMのホルボール12-ミリスチン酸13-酢酸(PMA;Sigma cat.no. P1585-1MG )で37℃、5%CO2で72h処理した。12ウェル組織培養プラスチックプレートでの細胞分化を必要とする実験では、1ウェルあたり2.5×105個の単球を使用し、各ウェルに1mLの培地を充填した。組織培養ディッシュ(直径60 mm)で単球を分化させる場合は、5 mLの培地に1.5×106個の単球を播種した。腸管単層を含む相互作用試験にマクロファージが必要な場合は、2.5×104個の単球をPMAを含む培地50μLに懸濁し、12ウェルガラスボトムプレートの個々のウェルの中心に播いた。1日後、各ウェルの総量を1 mLに増やし、単球をさらに2日間放置して分化させた。分化後、マクロファージは、方法のセクション「V. cholerae-immune cell interaction studies」に記載されているように、V. cholerae interaction studiesに使用された。
ヒト腸管上皮の培養
ヒト腸管上皮細胞(ドナー#1、Lonza cat.no. CC-2931、Lot 0000751359、胎児小腸から分離した初代細胞)をマトリゲル(Corning cat.no.356231)に埋め込み、ヒトオルガノイド拡大培地(hOEM)を用いて、37℃、5%CO2で加湿インキュベーター内で腸管に成長させた。hOEMはBasal培地(Advanced DMEM/F-12 [ThermoFisher cat. 番号12634010]、10mM HEPES [ThermoFisher cat.番号15630106]、1% GlutaMAX [ThermoFisher cat.番号35050061])を添加し、2% B-27 Supplement (ThermoFisher cat. 12587010), 1% N-2 Supplement (ThermoFisher cat. 17502001), 1mM N-acetyl-L-cysteine (Sigma-Aldrich cat. No. A9165)、10mMニコチンアミド(Sigma-Aldrich cat. N0636)、50ng mL-1 Human EGF(Sigma-Aldrich、E9644)、500ng mL-1 recombinant Human RSPO1-Fc(SUN Bioscience、EPFL)、100ng mL-1 recombinant Human Noggin-Fc(SUN Bioscience、EPFL)、10μM SB202190(Sigma-Aldrich cat.no.S7067 )、500nM A 83-01(Sigma-Aldrich cat.n.m. SML0788)、10 nM Prostaglandin E2(Lucerna-Chem cat.no.HY-101952)、10 nM human [Leu15]-gastrin I(Sigma-Aldrich cat.no.G9145), 0.5 nM Wnt-surrogate-Fc(ThermoFisher cat. 番号PHG0401)、30μg mL-1 ゲンタマイシン(ITW Reagents cat.番号A1492)および0.25μg mL-1 アンフォテリシンB(ThermoFisher cat. 番号15290018)である。成長した腸管は、前述したように継代した。
78
,
79
簡単に言うと、マトリゲルドームを氷冷Basal培地に溶解し、回収して5分間ペレット化した(200g、4℃)。腸管ペレットを1mLの氷冷Basal培地にアクティブピペッティングで再懸濁し、9mLの氷冷Basal培地を追加した。2回目の遠心分離ステップの後、腸管を解凍したばかりのマトリゲルに再懸濁した。腸球菌懸濁液25μLを含む液滴を24ウェルプレート(Corning cat.No.3526)のウェルに加え、2μMチアゾビビン(Sigma-Aldrich cat.No.SML1045)2.5μM CHIR99021(Sigma-Aldrich cat.No. 361571)を補充した500μL hOEMを加える前に37℃で15分間マドリゲルドームを重合した。エンテロイドは、密度に応じて1:2または1:3の比率で4-5日ごとに継代された。エンテロイドは、継代3から10までの間の実験に使用された。
図S6に示す実験では、ドナー#2からのオルガノイド(3dGRO Duodenum Intestinal Organoids, Merck/Sigma-Aldrich cat.no. SCC324、33歳女性ドナーから単離されたもの)を用いた。これらのドナー#2腸管は、製造者のプロトコールに従って培養され、上記のように継代された。
分化したヒト腸管モノレイヤーの培養
ドナー#1からの腸管単層は、製造業者のプロトコールに従って作成した。簡単に言うと、腸管を遠心分離(5分、200g、4℃)で集め、1mLのTrypLE(ThermoFisherのカタログ番号12605010)を使って単細胞に分解した。単細胞を40μmのセルストレーナー(pluriSelect cat.no.PS-43-10040-40)に通し、遠心分離(5 min, 200 g, 4℃)し、SingleQuote Supplements(Lonza cat.no.CC-4149) を添加した SmBM-2(Lonza cat.no. CC-3181)に再懸濁しました。細胞数を1.15x106細胞mL-1に調整し、100μLの細胞懸濁液を、ラットテールコラーゲンI型(30μg mL-1; Sigma-Aldrich cat. No. C3867)でプレコートした3μmの透明PET膜インサート(ファルコン cat. No. 353096)上に添加した。インサートをヒト筋線維芽細胞(Lonza cat.no. CC-2902, Lot 0000662132)の上に配置した。ヒト筋線維芽細胞は、製造者のプロトコールに従って培養し、1mLのSmBM-2(Lonza cat. no. CC-3181)を充填した24ウェル細胞培養プレート(Corning cat. no. 3526)のウェルに、前日、シングルクォート サプリ(Lonza cat. No. CC-4149)を補充して1x105細胞を播いた。細胞の分化と上皮単層の生成を達成するために、ヒト小腸上皮細胞を筋線維芽細胞と37℃、5%CO2の加湿インキュベーターで6日間共培養した。培地は播種の翌日に交換し、その後は1日おきに交換した。分化6日後のコンフルエントな上皮単層は、約9.1x104個の細胞から構成されていた(n = 4独立播種によるPET膜上の平均値)。
ドナー#2腸管単層は、3dGRO Duodenum Intestinal Organoids (Merck/Sigma-Aldrich cat. no. SCC324) から、上記のパラグラフでドナー#1について述べたものと類似のプロトコルを使用して生成した。単細胞懸濁液は、上記のLonzaオルガノイドについて記載したものと同様のプロトコルで作成した。細胞をセルストレーナーに通し、最後の遠心分離ステップを行った後、細胞を、2μMチアゾビビン(Sigma-Aldrichの猫番号SML1045)および2.5μM CHIR99021(Sigma-Aldrich の猫番号361571)を補充したhOEMに再懸濁した。細胞数を1x106 cells mL-1に調整し、100μLの細胞懸濁液を、ラットテールコラーゲンI型(30μg mL-1; Sigma-Aldrich cat. No. C3867)でプレコートした3μmの透明PET膜インサート(ファルコン cat. No. 353096)上に添加した。このインサートを、2μMのチアゾビビン(Sigma-Aldrichの猫番号SML1045)および2.5μMのCHIR99021(Sigma-Aldrichの猫番号361571)を補充した500μLのhOEMで満たされた24ウェル細胞培養プレート(Corning cat.no 3526)の1ウェルに入れた。均一な単層を形成するために、37℃、5%CO2の加湿インキュベーターで7日間インキュベートした。培地は、細胞を膜に播種した1日後に交換し、その後、2μMのチアゾビビン(Sigma-Aldrichの猫番号SML1045)のみを補充した新鮮なhOEMを用いて、7日目まで1日おきに交換した。7日目からは、ヒトオルガノイド分化培地hODM(2%B-27TMサプリメント[ThermoFisher cat.no.12587010],1%N-2 サプリメント[ThermoFisher cat.no.17502001],1mM N-acetyl-L-cysteine[Sigma-Aldrich cat.no.12587010] を添加したBasal medium)で培養された。A9165]、50 ng mL-1 Human EGF、100 ng mL-1 recombinant Human Noggin-Fc、500 nM A-83-01、10 nM human [Leu15]-gastrin I [Sigma-Aldrich cat. no. G9145]、30 μg mL-1 gentamicin [ITW Reagents cat. no. A1492]) で単層が完全に分化するまでさらに5日間に亘って行った。
79
12日間の分化の後、コンフルエントになった上皮単層は、約31x104個の細胞から構成されていた(n = 3独立した播種によるPET膜上の平均値)。このプロトコルに対応するデータを図S6に示す。
方法の詳細
細菌株の構築
本研究で使用した細菌株、プラスミド、オリゴヌクレオチドを表S4、S5、およびS6に示す。遺伝子欠失を生成するために、プラスミドpNUT144と同様にV. choleraeからの目的の遺伝子の1 kbpフランキング領域を増幅し、ポリメラーゼ連鎖反応(PCR)産物をGibsonアセンブリを介して組み立てた。
80
得られたプラスミドをKDV201株にコンジュゲートし、既述のプロトコルにしたがって菌体を選択した。
81
最終的な選択ステップの後、細菌はPCRによって遺伝子欠失をスクリーニングされた。
野生型tcpFをヒスチジン(His)タグ付きtcpFと交換することにより、C末端6xヒスチジン(His)タグ付きTcpFを発現するV. choleraeを作成した。このためにプラスミドpNUT2130を構築し、V. choleraeにコンジュゲートし、PCRおよびDNA配列決定により、遺伝子置換の成功を確認した。同様に、C末端に6xヒスチジン(His)タグを持つRbmAおよびRbmC、またはC末端にヘマグルチニン(HA)タグを持つBap1を発現するV. cholerae株を作成し、それぞれプラスミドpNUT918、pNUT2061およびpNUT2049を使用して結合させた。
目的の遺伝子(mshA、flaA)をそのネイティブプロモーターとともにV. choleraeのlacZ部位に挿入することにより、相補性株を得た。このために、目的の遺伝子、そのプロモーター、プラスミドpNUT276を増幅し、PCR産物をギブソンアセンブリーでプラスミドに組み込んだ。
80
このプラスミドをV. choleraeにコンジュゲートし、上記のように細胞を選択した。lacZ部位へのDNA挿入は、5-ブロモ-4-クロロ-3-インドリル-β-D-ガラクトピラノシド(X-Gal、最終濃度40μg mL-1)を用いた細菌コロニーの青/白スクリーニングで確認した。
vps の発現を調べるための蛍光転写レポーターを作成するために、プラスミド pNUT2111 を用いて sfgfp を vpsF と vpsG の間のネイティブ vps 遺伝子座に挿入した。
V.コレラにおけるToxTの過剰発現のために、アラビノース誘導性プロモーターPBADとtoxTを一緒に染色体上のlacZ部位に統合した。これを実現するために、pNUT276をベースに、toxTとPBADを保有するプラスミドを構築した。このプラスミドをV. choleraeにコンジュゲートし、DNA断片の挿入について細菌を選択した。
V. choleraeにおけるヘモリシンHlyAの過剰発現は、過剰発現プラスミドpNUT2425を使用して達成した。このプラスミドの構築には、V. cholerae hlyAとプラスミドpNUT1246のバックボーンを増幅し、ギブソンアセンブリを介して結合した。
80
このプラスミドをV. choleraeに結合させ、過剰発現株を作製した。
V. コレラで RocS と CdgJ を過剰発現させるために、過剰発現プラスミド pNUT3068 を構築した。このために、V. choleraeのrocSとcdgJ、およびプラスミドpNUT1246のバックボーンを増幅し、ギブソンアセンブリを介して結合した。
80
得られたプラスミドをV.コレラに結合させ、過剰発現株を作製した。
一般に、pKAS32由来の自殺ベクターpNUT144に基づくプラスミドクローンは、まず大腸菌S17-1 λpir株で構築した。その後、このプラスミドをV. choleraeにコンジュゲートした。pSC101複製起点を持つプラスミドは、大腸菌Top10株で構築し、その後、三親交配によりコンジュゲーションヘルパープラスミドpRK600を保有する追加の大腸菌株の助けを借りて、V. choleraeに移植されました。
V. コレラ細胞の蛍光イメージングのための sfGFP の構成的生産を可能にするために、スーパーフォルダー緑色蛍光タンパク質遺伝子(sfgfp)を含むプラスミド pNUT542 を用いた。
82
を含むプラスミド pNUT542 を、ヘモリシン過剰発現株を除くすべての V. コレラ菌に結合させた。これらの株では、Ptac-sfgfpをプラスミドpNUT480を用いてV. choleraeの染色体上のlacZ部位に組み込んだ。
V. コレラ菌と免疫細胞の相互作用試験
単離したばかりの単球、好中球、CD4+T細胞、B細胞、NK細胞との相互作用研究のために、免疫細胞をCellTracker Deep Red Dye(ThermoFisher cat. C34565、最終濃度0.75μM、Hanks Balanced Salt Solution(HBSS)に懸濁)またはCellTracker Red CMTPX Dye(ThermoFisherのカタログ番号C34552、最終濃度3μM、HBSSに懸濁)により37℃、加湿5%CO2インキュベーター内で15分間、染色した。染色した免疫細胞をペレット化(200g、5分)して染色液を除去し、10%(v/v)FCSおよびゲンタマイシン(最終濃度30μg mL-1)を補充したGlutaMax入りDMEMに再懸濁した。細胞数を4.572×105細胞mL-1に調整し、細胞懸濁液175μL(8×104免疫細胞に相当)を96ウェルガラス底プレートの1ウェルに添加した。その直後、免疫細胞をV.コレラに曝露した。このために、増殖した細菌(細菌の増殖条件は上述の通り)を一度洗浄し、新鮮な細胞培養培地を用いて4.1×108細胞mL-1に希釈した。その後、25μLの細菌懸濁液を免疫細胞に加え、播種時のMOIを128とし、細胞間相互作用を蛍光共焦点顕微鏡でモニターした。合計で、ウェルは200μLの液体で満たされた。
マクロファージを用いた実験では、96ウェルガラスボトムプレートのウェルから3日後(THP-1細胞株)または7日後(初代細胞)に分化培地を全量除去し、ガラス面に付着したマクロファージを100μLの染色液(前述のHBSSに懸濁したCellTracker)で37℃、加湿5%CO2インキュベーター中で15分間染色した。その後、染色液を除去し、マクロファージをHBSSで1回洗浄した後、V. choleraeに曝露した。細菌懸濁液は、THP-1単球由来マクロファージを調査する場合に細菌を1.6×107細胞mL-1に希釈した以外は、前項と同様に調製した。初代細胞を使用した場合は、5.12×107細胞mL-1を含むように細菌懸濁液を調製した。必要に応じて、細菌懸濁液を生成する前に、細胞培養液にヨウ化プロピジウム(最終濃度8μM)を補充し、これにより、V. cholerae-マクロファージ相互作用試験中に死んだマクロファージを検出することができた。最後に、染色・洗浄したマクロファージに細菌懸濁液を200μL添加し、直ちに蛍光共焦点イメージングを開始した。
V.コレラ-マクロファージ相互作用試験を12ウェルプラスチックボトムプレートで実施した場合、マクロファージを蛍光染色するために500μLの染色液を使用した以外は前項と同様にTHP-1単球由来マクロファージと細菌を準備した。さらに、細菌懸濁液は1.73×107細胞mL-1を含み、この懸濁液1mLを染色・洗浄した1.35×105マクロファージに添加し、初期MOIを128とした。
腸管単層を含むV.コレラ-免疫細胞相互作用実験のために、1mLの染色液を使用した以外は、上記のようにマクロファージを調製した。染色後、500μLの新鮮なSmBM-2(SingleQuots Supplementsおよび30μg mL-1ゲンタマイシンを補充)またはhODM(30μg mL-1ゲンタマイシンを補充)を染色したマクロファージに添加した。腸管単層(「分化したヒト腸管単層の培養」の項で説明したように調製した)を、CellMask Deep Red Plasma membrane stain(ThermoFisher cat.no. C10046、最終濃度2.5μg mL-1、SingleQuots Supplementsを補充したhODMまたはSmBMに懸濁)を37℃、5%、CO2で加湿したインキュベーター内で30分間、染色した。染色した腸管単層を新鮮な培地で1回洗浄した後、SmBM-2(SingleQuots Supplementsおよび30μg mL-1ゲンタマイシンを添加)またはhODM(30μg mL-1ゲンタマイシンを添加)50μLを頂部側に添加した。その後、腸管上皮単層膜を有するメンブレンインサートをマクロファージの上に置き、ポリ(ジメチルシロキサン)(PDMS)ホルダーを用いて位置を固定した。増殖した細菌をBasal培地を用いて1回洗浄し、新鮮なSmBM-2培地(SingleQuots Supplementsおよび30μg mL-1ゲンタマイシンを補充)またはhODM(30μg mL-1ゲンタマイシンを補充)で6.4x107細胞mL-1に希釈した。その後、50μLの細菌懸濁液を分化した腸管単層の先端側に加え、蛍光共焦点顕微鏡を用いて、V. choleraeと上皮細胞およびマクロファージとの相互作用をモニターした。腸管上皮単層体を含むほとんどの実験では、MOIを変化させた実験を除き、50μLの細菌播種懸濁液は3.2x106個のVコレラ細胞を含んでいた(図S5B)。腸上皮単層の下に、THP-1細胞に基づく実験では2.5x104個のマクロファージを、初代細胞に基づく実験では8x104個のマクロファージを培養した。
MSHA pili、TC pili、および分泌タンパク質TcpF、RbmA、RbmC、Bap1、HlyAの免疫蛍光法による視覚化
マクロファージ(96ウェルガラス底プレートで分化)の表面に形成されたバイオフィルム中のMSHA piliおよびTC piliを可視化するために、THP-1単球由来マクロファージを37℃、5% CO2で4時間V. choleraeに曝露した。その後、ウェルから培養液全体を除去し、サンプルを固定するために、100μLの4%パラホルムアルデヒド溶液(シグマ社製 cat. F8775-25ML;1xリン酸緩衝生理食塩水、PBS、pH7で希釈)により固定した。このステップ、およびそれに続くすべてのステップは、室温で行った。インキュベーション時間10分後、固定液を除去し、200μLの2%ウシ血清アルブミン(BSA;Sigma cat.no. A3059-100G;1x PBS, pH7に溶解)をウェルに添加した。1時間インキュベートした後、BSAを除去し、100μLのMshAまたはTcpAに対する一次抗体[ウサギ抗MshA、
71
またはウサギ抗TcpA
72
]を2μg mL-1の最終濃度でウェルに添加した(1x PBS、pH 7で希釈)。2時間後に抗体を除去し、200μLの1x PBS pH 7を用いてウェルを3回洗浄し、各洗浄ステップの間に10分間インキュベーションした。その後、Alexa Fluor 647に結合した二次抗ウサギ抗体(ThermoFisher cat.no. A-21244)100μLを2μg mL-1の最終濃度で加えた(1x PBS, pH7で希釈)。2時間後、ウェルを上記のように3回洗浄し、100μLの4%パラホルムアルデヒド溶液を用いて10分間、サンプルを2回目に固定した。最後に、蛍光イメージングを行うまで、サンプルを100μLの1xPBS(pH7)中で4℃にて保存した。
ヒト小腸上皮単層との共培養実験中に、MSHA piliおよびTC piliがマクロファージ上のバイオフィルムで可視化された場合、「V. cholerae-immune cell interaction studies」のセクションで説明したように共培養実験を設定し、マクロファージ上の細菌バイオフィルム形成を共焦点蛍光顕微鏡で監視した。マクロファージ表面に厚いバイオフィルムが観察された時点で、腸内単層膜を保有するインサートを含む全培養液を除去した。その後、上記と同様の固定と抗体標識のプロトコルを適用し、1点のみ変更した: 各ステップでは、ウェルの全面を覆うために500μLの液体を使用した。
分泌タンパク質TcpF、RbmA、RbmC、Bap1およびHlyAは、生きたサンプルの免疫蛍光染色によって可視化した。TcpF、RbmAおよびRbmCの場合、マクロファージにC末端6xヒスチジン(His)-Tagを保有する目的のタンパク質を発現するV. choleraeを曝露し、細胞培養液にAlexa Fluor 647(Qiagenのカタログ番号35370、最終濃度0.4 μg mL-1)と結合した抗6x-His-Tag抗体 を補充し、細胞ライブイメージングは、この抗体の添加が行われました。Bap1を調べる場合、マクロファージにC末端ヘマグルチニン(HA)-Tagを持つBap1を発現するV.コレラを曝露し、培地にAlexa Fluor 647(ThermoFisherの猫番号26183-A647)に結合した抗HA-Tag抗体を終濃度0.4μgmL-1で添加した。HlyAの可視化のために、マクロファージは、hlyAを過剰発現しているV. cholerae細胞に曝露された。細胞培養液には、HlyAを過剰発現させるために0.1 mM IPTGを添加するとともに、Alexa Fluor 647に結合した二次抗マウス抗体(ThermoFisher cat.no. A21235; 最終濃度4 μg mL-1)と共に4 μg mL-1のマウス抗HlyA抗体 (ThermoFisher cat. no. MA1-83520) も添加されました。
異なるマクロファージ溶解液濃度の存在下での細菌増殖の測定
V.コレラの増殖に対するマクロファージ溶解液の効果を調べるために、3x106個の単球を、上記のように組織培養皿でマクロファージに分化させた。その後、分化培地を除去し、セルスクレーパーを用いてマクロファージを表面から掻き出した。マクロファージを回収し、10%(v/v)FCSおよびゲンタマイシン(最終濃度30μg mL-1)を補充したGlutaMax入り新鮮DMEM1mLに再懸濁した。マクロファージ溶解液を生成するために、免疫細胞を4℃で10分間超音波処理することによって溶解し(超音波処理の設定:3秒オン、90秒オフ、50%振幅、50%一定)、12.000rpmで1分間遠心分離することによって細胞破片を除去した。マクロファージ溶解液は、細菌増殖試験に直ちに使用した。V.コレラ菌の細胞は、まずゲンタマイシン(最終濃度30μg mL-1)を添加したLB培地で増殖させ、その後、上記の「細菌の増殖条件」のセクションに記載したように、10%(v/v)FCSおよびゲンタマイシン(最終濃度30μg mL-1)を補充したGlutaMax添加DMEMに移した。成長した細菌を新鮮なDMEM(GlutaMax、10%(v/v)FCSおよびゲンタマイシンを補充)で一度洗浄し、続いて3.2x106個の細菌細胞を96ウェルプレートの各ウェルに添加した。各ウェルは、GlutaMax、10%(v/v)FCSおよびゲンタマイシンを補充したDMEMをベースに、異なる濃度のマクロファージ溶解液も含む200μLの液体を含んだ。細菌増殖は、自動プレートリーダーで37℃にてモニターした。
共焦点蛍光顕微鏡法
コレラとヒト免疫細胞との相互作用をモニターするために、横河CSUスピニングディスクユニットをNikon Ti-Eclipse 倒立顕微鏡に取り付け、40xオイルNA1.3対物レンズ(Nikon)とAndor iXon-Ultra EMCCDカメラで蛍光共焦点画像を撮った。腸管単分子膜を含む実験は、20x空気NA0.75対物レンズで撮像した。また、蛍光タンパク質や色素の励起には、3つのレーザー(488nm、552nm、637nm)を使用した。画像は、EMCCDカメラのEMゲインを用いて、低い励起光強度、30msの露光時間で、30分ごとに取得した。フォーカスドリフトは、ハードウェアオートフォーカスシステム(PFS、ニコン)を用いて補正した。ハードウェアの制御は、Micro-Manager
73
またはNIS Elements (Nikon)で制御した。免疫蛍光染色によって可視化された分泌タンパク質およびIV型ピリは、100倍油NA1.45対物レンズ(Nikon)を用いて画像化された。バイオフィルムの構造解析とマクロファージ表面での細菌増殖の追跡には、100倍のオイルNA1.45対物レンズ(Nikon)を使用し、追加の2倍レンズをCSUとNikon Ti-E side portの間に配置した。顕微鏡には、ライブセルイメージング中の温度とCO2レベルを制御するためのインキュベーションチャンバーが装備されていた。
酵素結合免疫吸着法(ELISA)
V. choleraeとマクロファージの相互作用の際に分泌されるサイトカイン(IL-1β、IL-8、TNF-α)の定量にサンドイッチELISAを使用した。培養上清は、共培養開始から4時間後に採取した。これは、細菌バイオフィルムの分散が起こると予測される約20分前に相当する。この相互作用の時点では、ごく少数のマクロファージのみが死滅している(図S2C、青線)。ELISAキットはThermoFisher社から購入し(猫番号88-7261、88-8086、88-7346-22)、実験は製造者の説明書に従って実施された。
細菌RNAシーケンスのためのサンプル調製
RNA-シークエンシングのために、V. cholerae-マクロファージ相互作用アッセイを12ウェルプラスチックボトムプレートで実施した。THP-1由来マクロファージを加湿した5%CO2インキュベーター内で37℃、規定時間、V. コレラに曝露した後、サンプルをRNAlater(Qiagen、猫番号76106)で固定した。培養上清を回収した。ウェルに残った付着した細胞を掻き取り、別々に回収した。4℃で2000g、10分間の遠心分離後、細胞ペレットを液体窒素でスナップ冷凍し、mirVana Kit (Ambion, cat. no. AM1560) を用いて製造者の指示に従ってRNA抽出を行うまで-80℃で保存した。RNAの品質は、電気泳動(Agilent 2100 Bioanalyzer)を用いて確認した。シーケンスライブラリーの調製とイルミナシーケンスは、Vertis Biotechnologie AG, Freising-Weihenstephan, Germanyによって実施された。
細菌RNAシーケンス後の細菌リード処理およびマッピング統計量
Fastqリードは、参照ゲノムV. cholerae O1 biovar El Tor str.にアライメントした。N16961(NCBI参照配列NC_002505.1およびNC_002506.1)に、STARを使用してアライメントした。
74
(バージョン2.5.3a)を使用した。リードカウントはSTARを使用して計算した。データはライブラリサイズのファクターで正規化した。ファクターは、各サンプルのリード総数を全サンプルのリード総数の中央値で割ることで算出しました。データはlog2スケールに変換された。最小発現量閾値は5に設定した。Fold変化は、すべてのナイーブサンプルの平均値から各サンプルについて計算された。主成分分析(PCA)を全遺伝子について実施した。アップレギュレートされた遺伝子を特定するために、1.5の閾値が設定された。
マクロファージRNAシーケンスのためのサンプル調製
RNA配列決定のために、V.コレラ-マクロファージ相互作用アッセイを12ウェルプラスチックボトムプレートで実施した。THP-1由来マクロファージを加湿された5%CO2インキュベーター内で37℃、規定時間、V. コレラに曝露した後、サンプルをRNAlater(Qiagen、猫番号76106)で固定した。培養上清を除去した。ウェルに残った付着した細胞を掻き取り、回収した。4℃で2000g、10分間の遠心分離後、細胞ペレットを液体窒素でスナップ冷凍し、mirVana Kit (Ambion, cat. no. AM1560) を用いて製造者の指示に従ってRNA抽出を行うまで-80℃で保存した。DNase処理後、エタノール沈殿でmRNAを精製した後、電気泳動(Agilent 1450 TapeStation)でRNAの品質をチェックした。
マクロファージライブラリーの調製とシークエンス
RNA-seqライブラリーは、前述したように、MARS-seqプロトコルを使用して調製した。
83
簡単に言うと、細胞からのmRNAは、サンプルバーコードと同時にcDNAに変換された。サンプルをプールし、第2DNA鎖の合成とT7 in vitro転写を行った。増幅されたRNAは断片化され、その後、部分的なP5イルミナ配列がライゲーションされ、cDNAに変換された。P5の全配列とバーコード付きP7をPCRで追加した。ライブラリーを調製し、イルミナ装置でサンプルあたり0.8~2.6百万リードの深さまで配列決定した。
マクロファージリード処理およびマッピング統計
シーケンシングリードをフィルター処理し、デマルチプレックスし、MARS-seq解析パイプラインを使用して、ヒトゲノム(hg19)にマッピングした(前述)。
83
データは、ライブラリサイズ係数で正規化した。ファクターは、各サンプルのリード総数を全サンプルのリード総数の中央値で割ることで算出した。データはlog2スケールに変換された。最小発現量閾値は3.5に設定された。リードカバレッジが低いため、1つのサンプルがデータセットから削除された。
マクロファージ遺伝子発現差分解析
統計解析は、一元配置分散分析(one-way ANOVA)を用いて行った。多重検定を考慮するため、偽発見率(FDR)を使用した。調整済みp < 0.1の遺伝子は、有意に差次的に発現しているとみなされた。感染サンプルとナイーブサンプルの間の発現変化に基づいて、遺伝子をアップレギュレートとして分類した。
TcpF、MshAおよびsfGFPの精製
タンパク質TcpF、MshAおよびGFPは、Protino Ni-IDA Resin (Macherey-Nagel, cat. No. 745210.120) を用いたアフィニティクロマトグラフィーにより精製した。大腸菌BL21(DE3)Starを、C末端6x-His-Tagを有するtcpF、mshAまたはsfgfpを含むpETプラスミドでトランスフェクトした。タンパク質発現のために、0.05%ラクトース存在下、30℃で一晩かけて細胞を培養した。超音波処理による細胞溶解後、製造元のプロトコルにしたがってタンパク質を精製した。タンパク質の精製が成功したことは、SDS-PAGEによって確認された。
84
液体クロマトグラフィー質量分析(LC-MS)分析のためのサンプル調製
マクロファージ表面におけるMshAとTcpFの結合パートナーを同定するため、あるいはマクロファージと単球間のタンパク質存在量の差を調べるため、3x106 THP-1単球を、上記のように直径60 mmの培養皿を用いてマクロファージに分化した。マクロファージは、5mLのPBS(pH7.5)で3回洗浄した。その後、氷冷した溶解バッファーを合計6mL添加した(MshAおよびTcpF実験用: 50 mM Tris、150 mM NaCl、5%グリセロール、0.5% NP40、pH 7.5;タンパク質存在量の差の実験用: 1%デオキシコール酸ナトリウム、10 mM TCEP、100 mM Tris、pH = 8.5)。その後、マクロファージを表面から削り取り、回収した。マクロファージの溶解は、氷上で30分間、5分ごとに激しくピペッティングし、その後、ティッシュグラインダーを使用することで達成した。THP-1単球(3x106.cells)は、各実験で遠心分離(200g、5分)により収集し、5mLのPBSで3回洗浄した。最後の洗浄ステップの後、単球を上記のように溶解バッファーで溶解させた。マクロファージおよび単球からの溶解物は、さらに使用するまで-80℃で保存した。
プルダウン実験には、Protino Ni-IDA 150カラムを使用し(Macherey-Nagel、猫番号745150.10)、すべてのステップを4℃で実施した。製造元のプロトコールに従ってカラムを平衡化した後、精製した6x-Hisタグ付きベイト蛋白(MshA、TcpFまたはGFP)1mgをカラムにロードした。30分間のインキュベーションの後、カラムは重力によって排出された。その後、マクロファージまたは単球のライセートをカラムにロードした。細胞溶解液が通過した後、40mMのイミダゾールを含む500μLのLEWバッファーでカラムを5回洗浄した。最後に、結合したタンパク質を250mMイミダゾールを含む240μLのLEWバッファーで3回溶出し、100%TCA(Sigma、猫番号T6399)で沈殿させた。ペレットをアセトンで洗浄し、タンパク質を再懸濁バッファー(5% SDS、10mM TCEP、100mM TEAB)に溶解させた。
ディファレンシャルアバンダンス測定のために、単球とマクロファージの溶解液をBioRuptorシステム(Diagenode、10サイクル、30秒ON、30秒OFF)を用いて超音波処理した。
すべてのサンプルのタンパク質濃度は、トリプトファン蛍光解析
85
は、少量のサンプル分注を用いて測定した。全タンパク質50μgを含むサンプルアリコットを95℃で10分間還元し、15mMクロロアセトアミドで37℃、30分間アルキル化した。ペプチドは、iSTカートリッジ(Pre-Omics、ミュンヘン)を用いて、製造者のプロトコルに従って脱塩された。サンプルは真空下で乾燥させ、LC-MS分析にさらに使用するまで-20℃で保存した。
液体クロマトグラフィー-質量分析(LC-MS)分析
乾燥したペプチドサンプルを0.1%ギ酸水溶液に溶解し、0.15μgのペプチドを前記のようにLC-MS分析した。
86
マクロファージと単球のタンパク質量の差分解析のために、ペプチドのクロマトグラフィー分離は、1.9μmのC18樹脂(Reprosil-AQ Pur、Maisch博士)を自社で充填した加熱RP-HPLCカラム(75μm×30cm)を備えたUltimate 3000 nano-LC (Thermo Fisher Scientific) で実施されました。ペプチドは、LC-MS/MSランごとに、98%溶媒A(0.15%ギ酸、2%アセトニトリル)および2%溶媒B(98%アセトニトリル、2%水、0.15%ギ酸)から5分かけて12%B、その後45分で35%Bまで上昇、10分で50%Bまで上昇するステップ状の線形グラジェントを用い、流速300nL min-1で分析しました。質量分析分析は、ナノエレクトロスプレーイオン源(いずれもThermo Fisher Scientific社製)を搭載したOrbitrap Eclipse質量分析計で、データ非依存取得(DIA)を用いて実施した。MS1では、最大注入時間45msでNormalized AGC Target設定を300%に設定し、120,000 FWHMの分解能でスキャンを記録した。DIAスキャンは、Normalized AGC Target設定400%で、蓄積時間22ms、分解能15,000 FWHM(200m/z時)に設定されて取得されました。386から1016m/zの質量範囲をカバーする9m/zの大きさの合計70個のDIAウィンドウが、1m/zのウィンドウの重なりでMSサイクルごとに収集されました。HCDの衝突エネルギーは28%(正規化)に設定され、すべてのデータはセントロイドモードで取得された。
MshA、TcpFおよびGFPとのアフィニティプルダウン解析のために、ペプチドは、EASY-nLC 1000(両方ともThermo Fisher Scientific)を装着したQ Exactive Plus質量分析計を使用してLC-MS/MS解析に供された。ペプチドは、EASY-Sprayカラム(PepMap RSLC, C18, 2 μm, 100 A, 75 μm × 25 cm, Thermo Scientific)を用いて、200 nL min-1の流速で分離しました。ペプチド分離には、5分間で5%のバッファBから10%のバッファBまで、次いで30分間で35%のバッファBまで、次いで5分間で50%までバッファBを増加させる段階的直線グラジェントを使用した。バッファAは水中0.1%ギ酸からなり、バッファBは水中80%アセトニトリル、0.1%ギ酸からなる。MS1では、3x106個のイオンが最大100msの時間にわたってOrbitrapに蓄積され、70,000 FWHMの分解能でスキャンされた(200 m/zで)。MS2スキャンは、1x105イオンをターゲットに設定し、最大蓄積時間50ms、分解能17,500 FWHM (at 200 m/z)で取得しました。単荷電イオン、電荷状態≧6のイオン、および電荷状態が未指定のイオンは、MS2イベントのトリガーから除外した。正規化コリジョンエネルギーは27%に設定し、質量分離ウィンドウは1.4m/zに設定し、各スペクトルに対して1つのマイクロスキャンを取得した。
質量分析(MS)データ解析
マクロファージと単球のタンパク質量の差分解析のために、取得した生ファイルは、デフォルト設定を使用して、SpectroNautソフトウェア(Biognosys、バージョン15.7.220308.50606)によって、ヒトUniProtタンパク質データベース(バージョン2月2022)および一般的に観察される汚染物質(合計20753配列)に対してdirectDIAによって検索されました。検索条件は、完全なトリプシン特異性が必要(リジンまたはアルギニン残基の後にプロリンが続かない限り切断)、2回の切断ミスが許される、カルバミドメチル化(C)は固定修飾、酸化(M)とN末端のアセチル化は可変修飾とした。誤認識率は1%に設定された。検索結果はSpectroNautからエクスポートし、MSstats(v.4.2.0)を用いてタンパク質存在量を統計解析した。
75
マクロファージ表面に関連するタンパク質を、すべての差分存在量タンパク質(fold change >2, p value < 0.05)から特定するために、DAVIDを用いて以下のGene Ontology用語:細胞表面、細胞外領域、細胞外エクソソーム、細胞外空間、細胞膜外側、アピカル細胞膜を用いて細胞内位置についてデータをフィルタリングした。
87
,
88
また、UniProt Annotation "secreted "を持つタンパク質を検索した。
MshA、TcpF、GFPを用いたアフィニティプルダウン解析では、取得したRawファイルをMSFraggerを用いたFragPipeで検索した。
89
V. cholerae N16961 (EBI, release date 2022/5/12) のTcpFとMshAの予測UniProtエントリーと一般的に観察されるコンタミを含むヒトUniProtタンパク質データベース (version Feb 2022) に対して、取得したRawファイルをMSFragger 89を使用してFragPipeで検索した(合計20755個の配列がある)。検索にはデフォルト設定を用い、検索条件は、完全なトリプシン特異性が必要(リジンまたはアルギニン残基の後にプロリンが続かない限り切断)、2回の切断ミスが可能、カルバミドメチル化(C)は固定修飾、酸化(M)とN末端のアセチル化は可変修飾として設定した。タンパク質の偽発見率が1%になるように、ターゲット-デコイ検索戦略を使用した。検索結果はScaffoldソフトウェア環境(https://www.proteomesoftware.com)を用いて解析した。プルダウン実験で濃縮された全てのタンパク質からマクロファージ表面に関連するタンパク質を同定するために、以下のGene Ontologyの用語を用いて細胞内位置についてデータをフィルタリングした:細胞表面、細胞外領域、細胞外エクソソーム、細胞外空間、細胞膜外側、アピカル細胞膜。また、UniProt Annotation "secreted "を持つタンパク質を検索した。MshAのアフィニティプルダウン解析の場合、真核生物の結合パートナー候補のリストは、マクロファージと単球を用いた存在量差分析で、2倍以上の変化とp値<0.05を示したタンパク質についてさらにフィルターにかけられた。
画像解析
画像解析は、BiofilmQソフトウェアツール
90
を使用し、マクロファージの検出とバイオフィルムの存在量測定のためのMATLABスクリプトも使用した。画像分割は、最近レビューしたように、従来のフィルタリングと閾値処理を用いて行った、
91
機械学習ベースのセグメンテーションは行わなかった。特定の定量化のための特定の画像解析戦略は、以下に説明される。
マクロファージは、ノイズ除去のために生画像を中央値フィルタリングすることで画像から検出した。続いて、3つのクラスで強くぼやけた画像に大津の方法を適用した閾値処理を行い、2番目のクラスが前景となるように割り当てた。
92
蛍光チャンネル間の重複が少ないため、誤検出を避けるため、画像内の細菌細胞が存在する領域は除外された。閾値処理後、検出されたマクロファージ画像領域の小物除去や穴の除去のための形態学的演算を行い、マクロファージの輪郭を平滑化した。
死滅したマクロファージの検出には、死滅染色チャンネル(死滅染色:ヨウ化プロピジウム)の共焦点蛍光画像を中央値フィルターでノイズを除去し、その後、閾値を設定した。画質に応じて、閾値は3クラスで大津の方法で決定した2つの閾値の平均値に設定するか、画像内の99.5%の最低画素値にフィットする正規分布の平均値に7倍の標準偏差を加えた値で算出した。後者の方法では、死滅したマクロファージがほとんどない画像に対して、より正確なセグメンテーション結果を得ることができた。次に、得られた2値画像にモルフォロジー演算を適用し、非常に小さな2値オブジェクトを除去した。次に、死細胞蛍光チャンネルからの2値画像をマクロファージ検出(上述)と重ね合わせ、死細胞蛍光チャンネルに信号を含むマクロファージの数を数えることによって、結果として死マクロファージの数が決定された。死細胞の比率を求めるには、死細胞の数を全マクロファージの数で割り、マクロファージの典型的な大きさに対する大きさに応じて、触れている(一つの大きな物体として分割された)マクロファージを複数のマクロファージとして数えることで補正した。
マクロファージ周辺の体積あたりの付着細菌細胞の体積を定量化するために、大津閾値を用いた画像分割を行い、その後、ノイズに起因する非常に小さな分割オブジェクトを除去した。共焦点画像zスタックの各2次元画像について、4ピクセル(1.3μmに相当)拡大した後の画像でマクロファージオブジェクトに接続されている細菌が占めるすべての画像領域を、マクロファージに付着していると定義した。マクロファージの周囲にある環状の細菌が占める画像領域をすべて合計し、個別にマクロファージの周囲の環状領域の面積を算出した。免疫細胞に付着した細菌が数個の場合は環状の厚さを3μmとし、実験開始から0.5時間または1時間後に撮影した共焦点画像から付着量を定量化した。バイオフィルム形成を数時間調査し、時系列の全画像を解析した場合は、すべてのフレームで環状部の厚さを6μmに設定した。zスタックの3つのxyスライスについて、マクロファージ周辺の体積あたりの付着細菌の体積と環状体の体積を算出した後、すべてのスライスからの体積の比率を平均して、各マクロファージ周辺の環状体の体積あたりの付着細菌の体積とした。この定量化は、視野内のすべてのマクロファージについて実施した。
図3B-3Dの画像におけるバイオフィルムマトリックスの存在量の定量化のために、細菌細胞とマトリックスは、それらの別々の蛍光チャンネルで閾値を使用してセグメント化した。その後、BiofilmQの "merge channels "機能を用いて、細菌の位置を示す2値画像とマトリックスを示す2値画像を統合し、バイオフィルム全体のバイオボリュームセグメンテーションを得た。これらの2値画像は、バイオフィルムマトリックスの存在量を定量化するための基礎となった。
マクロファージ周辺のヘモリシンの量を定量化するために、検出されたヘモリシン(蛍光抗体による)とマクロファージの画像を大津閾値処理でセグメント化しました。その後、検出されたマクロファージ表面から7μmの距離にある体積の中で、ヘモリシンが占める体積を測定した。
バイオフィルムの構造は、まず、以前に説明したアルゴリズムを用いて3D細胞分割を行うことにより、単一細胞レベルで分析した。
26
単細胞セグメンテーションの結果得られたバイナリ画像をBiofilmQにインポートし、下流の定量化を行った。このアプローチにより、各細胞周辺の局所的な細胞配列(ネマティック秩序パラメータとして定量化)、局所的な細胞間距離(細胞中心から細胞中心までの距離として定量化)、バイオフィルム内の局所的な細胞密度を決定しました。これらの特性はすべて、バイオフィルム内のすべての細胞について、各細胞を中心とした半径45ピクセル(2.9μm)の球体で局所的に定量化した。
模式図
図6、S5、S6の模式図、およびグラフの要約は、BioRender.comで作成しました。
定量化および統計解析
統計解析はPrism(GraphPad Software Inc.)を使用して行った。実験の統計的な詳細は、図の説明文に記載されています。
データおよびコードの入手
トランスクリプトームの生データおよび解析データは、United States National Center for Biotechnology Information Gene Expression Omnibusで入手可能である。質量分析プロテオミクスデータは、PRIDEを介してProteomeXchange Consortiumに寄託されています。
76
パートナーリポジトリに寄託されています。アクセッション番号は、主要リソース表に記載されています。顕微鏡データは、要求に応じてリードコンタクトが共有します。
画像解析に使用されたオリジナルコードは、Zenodo上でキーリソース表に記載されたDOIで公開されています。
本論文で報告されたデータの再解析に必要な追加情報は、要求に応じてリードコンタクトから入手可能である。
謝辞
ウサギの抗TcpA6抗体を惜しみなく提供してくれたKaren Skorupski、白血球の分離に協力してくれたNils Schmererに感謝する。また、Fitnat H. Yildizの助言、Sanika Vaidya、Daniel Rode、Praveen K. Singhの菌株、バーゼル大学BiozentrumのProteomics Core FacilityのAlexander SchmidtとThomas K.C. Bockに感謝します。この研究は、Minna James Heineman Foundationからの助成金(K.D.とR.A.に)、Deutsche Future Foundationからの助成金によって実現した。 )、Deutsche Forschungsgemeinschaft (DR 982/5-1, DR 982/6-1, and SPP 2389)、欧州研究評議会を通じた欧州連合の研究・イノベーションプログラムHorizon 2020(助成協定716734)、Bundesministerium für Bildung und Forschung (TARGET-Biofilms 16GW0245)、 マックス・プランク協会、スイス国立科学財団助成金51NF40_180541による国立研究能力センターAntiResist、スイス国立科学財団連結助成金TMCG-3_213801(K. D.). S.M.は、Peter und Traudl Engelhorn Stiftung zur Förderung der Lebenswissenschaftenからのポスドクフェローシップによって資金を得た。H.J.はStudienstiftung des Deutschen VolkesとJoachim Herz Foundationから資金援助を受けています。A.P.は、Human Frontier Science Program(RGY0077/2020)およびスイス国立科学財団(310030_204190)から資金提供を受けている。R.A.は、イスラエル科学財団(助成番号1289/22)の支援を受けています。B.S.とL.N.S.は、Hessisches Ministerium für Wissenschaft und Kunst (LOEWE Diffusible Signals) とDeutsche Forschungsgemeinschaft (SFB/TR-84 TP C01 and TP C10) から資金援助を受けています。B.S.は、ドイツ教育研究省(BMBF)(ERACoSysMed2 SysMed-COPD-FKZ031L0140およびPermed-COPD FKZ01EK2203)からも資金提供を受けています。
著者の貢献
L.V.とK.D.は、プロジェクトの構想、実験の設計、データの解析を行った。L.V.、S.M.、K.S.、K.R.は、顕微鏡実験を行った。L.V.、K.S.、K.R.は、細菌株を作製した。S.M.とA.P.は、ヒト腸管単層とマクロファージとの共培養モデルの確立を可能にした。H.J.は画像解析を行い、L.V.は画像解析を行った。V.N.、N.B.B.-M.、L.N.S.、R.A.はトランスクリプトーム解析を実施した。V.N.、N.B.B.-M.、R.A.がトランスクリプトームの追加計測を行った。K.S.はプロテオミクス実験を行った。K. Neuhausは顕微鏡のイメージング技術を開発した。K. Nosho、M.A.、L.N.S.は、RNAシーケンスのためのRNA調製に貢献した。A.D.、B.S.、L.S.はヒト試料を提供した。L.V.とK.D.は図を作成し、全著者の意見を取り入れて原稿を執筆した。K.D.はプロジェクトの監督と調整を行った。
利害関係の宣言
すべての著者は、競合する利害関係がないことを宣言している。
インクルージョンと多様性
私たちは、包括的で多様な、そして公平な研究の実施を支持します。
補足情報
.pdfをダウンロードする (.04 MB)
pdfファイルのヘルプ
ドキュメントS1. 参考資料の補足
.xlsx のダウンロード (.25 MB)
xlsxファイルのヘルプ
表S1. 図1に関連するマクロファージと単球のタンパク質存在量の差分解析
ダウンロード .xlsx (.03 MB)
xlsxファイルのヘルプ
表S2. MshAを餌とし、マクロファージ溶解液を餌とするアフィニティプルダウン解析で有意に濃縮されたタンパク質(図3関連
ダウンロード .xlsx (.03 MB)
xlsxファイルに関するヘルプ
表S3. 図3に関連する、TcpFをベイト、マクロファージライセートを餌としたアフィニティプルダウン解析で有意に濃縮されたタンパク質。
.xlsx のダウンロード (.01 MB)
xlsxファイルに関するヘルプ
表S4. STAR Methodsに関連する、本研究で使用した細菌株
ダウンロード .xlsx (.01 MB)
xlsxファイルのヘルプ
表S5. 本研究で使用したプラスミド、STAR Methods関連
ダウンロード .xlsx (.01 MB)
xlsxファイルのヘルプ
表S6. STAR Methodsに関連する本研究で使用したオリゴヌクレオチド
参考文献
Pernthaler J.
水柱における原核生物の捕食とその生態学的意味合い。
Nat. Rev. Microbiol. 2005; 3: 537-546https://doi.org/10.1038/nrmicro1180
記事で見る
スコープス (592)
PubMed
クロスフィルム
グーグル奨学生
ペレス J.
モラレダ=ムニョス A.
マルコス-トレスF.J.
ムニョス-ドラドJ.
細菌による捕食: 75年、そしてカウント。
Environ. Microbiol. 2016; 18: 766-779https://doi.org/10.1111/1462-2920.13171
記事で見る
スコープス(134)
パブコメ
クロスフィルム
グーグル奨学生
ユルゲンス K.
マッツ C.
プランクトン細菌の表現型と遺伝子型の組成を形成する力としての捕食。
Antonie Leeuwenhoek. 2002; 81: 413-434https://doi.org/10.1023/A:1020505204959
記事で見る
スコープス (332)
PubMed
クロスフィルム
グーグル奨学生
ロストル J.T.
マラフィニL.
Ph)ightingファージ: 細菌はどのように寄生虫に抵抗するのか。
セルホストマイクロベ。2019; 25: 184-194https://doi.org/10.1016/j.chom.2019.01.009
記事で見る
スコープス(129)
PubMed
アブストラクト
全文
全文PDF
Google Scholar
サランティスH.
グリンシュタイン S.
病原体生存のためのファゴサイトーシスの転覆。
セルホストマイクロベ。2012; 12: 419-431https://doi.org/10.1016/j.chom.2012.09.001
記事で見る
スコープス (94)
PubMed
要旨
全文
全文PDF
Google Scholar
アルシオラ C.R.
カンポッチャD.
モンタナロL.
インプラント感染症: 接着、バイオフィルム形成、免疫回避。
Nat. Rev. Microbiol. 2018; 16: 397-409https://doi.org/10.1038/s41579-018-0019-y
記事で見る
スコープス(1024)
パブコメ
クロスレフ
グーグル奨学生
マッツ C.
ケレベリ S.
オフ・ザ・フック-バクテリアはいかにして原生動物に食われて生き延びるか。
Trends Microbiol. 2005; 13: 302-307https://doi.org/10.1016/j.tim.2005.05.009
記事で見る
スコープス (453)
PubMed
要旨
全文
全文PDF
Google Scholar
マッツ C.
マクドゥーガルドD.
モレノ A.M.
Yung P.Y.
Yildiz F.H.
Kjelleberg S.
バイオフィルム形成と表現型の変化により、Vibrio choleraeの捕食主導型の持続性が強化された。
Proc. Natl. Acad. Sci. USA. 2005; 102: 16819-16824https://doi.org/10.1073/pnas.0505350102
記事で見る
スコープス (238)
パブコメ
クロスフィルム
グーグル奨学生
ビダコビッチL.
シン・P.K.
ハルトマンR.
ナデルC.D.
Drescher K.
バイオフィルムのダイナミックな構造により、ウイルス防御の個別的・集団的メカニズムがもたらされる。
Nat. Microbiol. 2018; 3: 26-31https://doi.org/10.1038/s41564-017-0050-1
記事で見る
スコープス(150)
パブコメ
クロスフィルム
グーグル奨学生
ウーチャーB.R.
エルサイードM.
アデルマンJ.S.
カドゥーリD.E.(Kadouri D.E.
ナデルC.D.
バクテリアの捕食がバイオフィルムの景観と群集形成を変える。
Curr. Biol. 2021; 31: 2643-2651.e3https://doi.org/10.1016/j.cub.2021.03.036
記事で見る
スコープス(16)
PubMed
要旨
全文
全文PDF
Google Scholar
ディアス-パスカルF.
ハルトマンR.
Lempp M.
ヴィダコヴィッチL.
ソンB.
イェッケル H.
トールマン K.M.
Yildiz F.H.
ダンケル J.
リンク H.
et al.
抗生物質によって誘発されるVibrio choleraeのバイオフィルム構造の破壊は、コミュニティのバリア機能を崩壊させる。
Nat. Microbiol. 2019; 4: 2136-2145https://doi.org/10.1038/s41564-019-0579-2
記事で見る
スコープス(45)
パブコメ
クロスレフ
グーグル奨学生
サーローL.R.
ハンケM.L.
フリッツT.
アングルA.
アルドリッチA.
ウィリアムズ S.H.
エンゲブレッツェンI.L.
ベイルズK.W.
ホースウィル A.R.
キエリアン T.
Staphyloccous auerusのバイオフィルムはmarcophageのpahgocytosisを防ぎ、in vivoで炎症を減衰させる。
J. Immunol. 2011; 186: 6585-6596https://doi.org/10.4049/jimmunol.1002794
記事で見る
スコープス (474)
PubMed
クロスフィルム
グーグル奨学生
クリスティアン・S.A.
ビルケンシュトックT.A.
サウダー U.
マック D.
ゲッツF.
ランドマンR.
バイオフィルムの形成はC3aの放出を誘導し、Staphylococcus epidermidisをIgGおよび補体の沈着から、また好中球依存性の殺傷から保護する。
J. Infect. Dis. 2008; 197: 1028-1035https://doi.org/10.1086/528992
記事で見る
スコープス (138)
パブコメ
クロスフィルム
グーグル奨学生
ドメネク M.
ラモス-セビジャーノE.
ガルシア・E.
モスコーソM.
ユステ J.
バイオフィルム形成が肺炎球菌の補体免疫と貪食作用を回避する。
Infect. Immun. 2013; 81: 2606-2615https://doi.org/10.1128/IAI.00491-13
記事で見る
スコープス (110)
パブコメ
クロスフィルム
グーグル奨学生
ガン・J.S.
バカレッツL.O.
ウォズニアックD.J.
外側にあるものが重要である: グラム陰性バイオフィルムの細胞外高分子物質が宿主免疫の回避に果たす役割と治療介入の標的としての役割。
J. Biol. Chem. 2016; 291: 12538-12546https://doi.org/10.1074/jbc.R115.707547
記事で見る
スコープス (115)
PubMed
概要
全文
全文PDF
Google Scholar
マルケイ・L.R.
イザベラV.M.
ルイス・K.
緑膿菌バイオフィルムの疾患への応用.
Microb. Ecol. 2014; 68: 1-12https://doi.org/10.1007/s00248-013-0297-x
記事で見る
スコープス(291)
パブコメ
クロスフィルム
グーグル奨学生
アルヘデM.
ローレンツM.
フリッツ B.G.
イェンセンP.Ø.
リングH.C.
ベイ L.
ビャーンショルト T.
細菌凝集体の大きさは多形核白血球の貪食効率を決定する。
Med. Microbiol. Immunol. 2020; 209: 669-680https://doi.org/10.1007/s00430-020-00691-1
記事で見る
スコープス (23)
パブコメ
クロスフィルム
グーグル奨学生
ヤン J.
バスラーB.L.
コミュニティとして生き残る: バクテリアバイオフィルムにおける抗生物質耐性と永続性。
セルホストマイクロベ。2019; 26: 15-21https://doi.org/10.1016/j.chom.2019.06.002
記事で見る
スコープス(250)
PubMed
アブストラクト
全文
全文PDF
グーグル奨学生
Høiby N.
ビャーンショルトT.
Givskov M.
モーリンS.
シオフO.
細菌性バイオフィルムの抗生物質耐性。
Int. J. Antimicrob. Agents. 2010; 35: 322-332https://doi.org/10.1016/j.ijantimicag.2009.12.011
記事で見る
スコープス (2183)
PubMed
クロスフィルム
グーグル奨学生
ホール=ストゥードリーL.
コスタートン・J.W.
ストッドリーP.
細菌性バイオフィルム:自然環境から感染症まで。
Nat. Rev. Microbiol. 2004; 2: 95-108https://doi.org/10.1038/nrmicro821
記事で見る
スコープス (4979)
PubMed
クロスフィルム
グーグル奨学生
リモリD.H.
ジョーンズC.J.
ウォズニアックD.J.
バイオフィルムの形成と機能における細菌細胞外多糖類.
Microbiol. Spectr. 2015; 3https://doi.org/10.1128/microbiolspec.MB-0011-2014
記事で見る
スコープス(488)
パブコメ
クロスフィルム
グーグル奨学生
ウォーカー T.S.
トムリンK.L.
ウォーテンG.S.(Worthen G.S.
ポーチ・K.R.
リーバー・J.G.
サーベドラ M.T.
フェスラーM.B.
マルコムK.C.
ヴァシルM.L.
ニックJ.A.
他
ヒト好中球による緑膿菌バイオフィルム形成の促進.
Infect. Immun. 2005; 73: 3693-3701https://doi.org/10.1128/IAI.73.6.3693
記事で見る
スコープス (0)
パブコメ
クロスフィルム
グーグル奨学生
チャンドラ J.
マコーミック T.S.
今村陽子
ムカルジー P.K.
Ghannoum M.A.
カンジダ・アルビカンスとヒト末梢血単核細胞との相互作用により、カンジダ・アルビカンスのバイオフィルム形成が促進され、炎症性サイトカインと抗炎症性サイトカインの発現に差異が生じる。
Infect. Immun. 2007; 75: 2612-2620https://doi.org/10.1128/IAI.01841-06
記事で見る
スコープス (103)
パブコメ
クロスフィルム
グーグル奨学生
ハリスJ.B.
ラロックR.C.(LaRocque R.C.
カドリF.
ライアンE.T.
カルダーウッドS.B.
コレラ
Lancet. 2012; 379: 2466-2476https://doi.org/10.1016/S0140-6736(12)60436-X
記事で見る
スコープス (430)
PubMed
概要
全文
全文PDF
グーグル奨学生
ベルクV.
フォン・J.C.N.
デンプシーG.T.
デベリオグルO.N.
荘X.
リップハルトJ.
Yildiz F.H.
チュー S.
Vibrio choleraeバイオフィルムの分子構造および組立原理。
Science. 2012; 337: 236-239https://doi.org/10.1126/science.1222981
記事で見る
スコープス (282)
PubMed
クロスフィルム
グーグル奨学生
ハルトマンR.
シン・P.K.
ピアースP.
モク・R.
ソン B.
ディアス=パスカル F.
ダンケル・J.
ドレッシャー K.
成長するバイオフィルムにおける3次元の秩序と構造の出現。
Nat. Phys. 2019; 15: 251-256https://doi.org/10.1038/s41567-018-0356-9
記事で見る
スコープス(151)
パブコメ
クロスレフ
グーグル奨学生
マタン M.M.
チャンディG.
マタンV.I.
コレラ患者における上部小腸粘膜の超微細構造的変化.
Gastroenterology. 1995; 109: 422-430https://doi.org/10.1016/0016-5085(95)90329-1
記事で見る
スコープス (62)
PubMed
要旨
全文PDF
グーグル奨学生
Qadri F.
ブイヤンT.R.
Dutta K.K.
ラキブR.
アラム M.S.
アラムN.H.
スヴェンナーホルム A.M.
マタンM.M.
Vibrio cholerae血清群O1およびO139による急性脱水症は、腸管粘膜表面における自然細胞および炎症メディエーターの増加を誘導する。
Gut. 2004; 53: 62-69https://doi.org/10.1136/gut.53.1.62
記事で見る
スコープス (86)
PubMed
クロスフィルム
グーグル奨学生
ワイルA.A.
ベッカーR.L.
ハリスJ.B.
免疫とマイクロバイオームの交差点にあるビブリオコレラエ。
mSphere. 2019; 4: e00597-19https://doi.org/10.1128/mSphere.00597-19
記事で見る
スコープス(26)
パブコメ
クロスフィルム
グーグル奨学生
フラッハC.F.
カドリF.
Bhuiyan T.R.
アラム N.H.
ジェニッシェ E.
レーンロート I.
ホルムグレン J.
急性コレラにおける自然界防御因子の幅広いアップレギュレーション。
Infect. Immun. 2007; 75: 2343-2350https://doi.org/10.1128/IAI.01900-06
記事で見る
スコープス (68)
PubMed
クロスフィルム
グーグル奨学生
ビショップ A.L.
パティマラB.
カミッリA.
新生児マウスコレラモデルにおけるVibrio cholerae誘発性炎症。
Infect. Immun. 2014; 82: 2434-2447https://doi.org/10.1128/IAI.00054-14
記事で見る
スコープス(20)
パブコメ
クロスフィルム
グーグル奨学生
アマドゥ・アマニ S.
ラング M.L.
腸管疾患を引き起こす細菌は、異なる体液性免疫応答を刺激する。
Front. Immunol. 2020; 11: 565648-565714https://doi.org/10.3389/fimmu.2020.565648
記事で見る
スコープス(3)
PubMed
クロスフィルム
グーグル奨学生
セパーA.
ホセインザデA.
ゴーキエビッチG.
リヒテネガーS.
ロイエS.
ライトナーD.R.
レーム M.
グルッチ A.
リードル J.
アーバン C.F.
シルド S.
ビブリオコレラエは、2つの細胞外ヌクレアーゼの活性により好中球の細胞外トラップを回避する。
PLoS Pathog. 2013; 9: e1003614https://doi.org/10.1371/journal.ppat.1003614
記事で見る
スコープス (96)
PubMed
クロスフィルム
グーグル奨学生
クイーン J.
サッチェル K.J.F.
ビブリオコレラエの腸管への封じ込めには、感染症の前炎症期に好中球が必須である。
Infect. Immun. 2012; 80: 2905-2913https://doi.org/10.1128/IAI.00356-12
記事で見る
スコープス (38)
PubMed
クロスフィルム
グーグル奨学生
アルマグロ-モレノS.
プルースK.
テイラーR.K.
Vibrio choleraeの腸内コロニー形成のダイナミクス。
PLoS Pathog. 2015; 11: e1004787https://doi.org/10.1371/journal.ppat.1004787
記事で見る
スコープス(99)
パブコメ
クロスフィルム
グーグル奨学生
キッツG.
ジリオ・K.M.
サモラーノ=サンチェスD.
パークJ.H.
タウンズリーL.
クーリーR.B.
ウーチャーB.R.
クロース K.E.
ナデルC.D.
Yildiz F.H.
他
ビブリオコレラエの大型アドヘシンを制御する保存された制御回路。
mBio. 2019; 10: 1-22https://doi.org/10.1128/mBio.02822-19
記事で見る
スコープス(18)
クロスレフ
グーグル・スカラー
テシュラーJ.K.
ナデルC.D.
ドレッシャーK.
Yildiz F.H.
Vibrio choleraeのバイオフィルム形成と分散を支えるメカニズム.
Annu. Rev. Microbiol. 2022; 76: 503-532https://doi.org/10.1146/annurev-micro-111021-053553
記事で見る
スコープス (9)
PubMed
クロスフィルム
グーグル奨学生
ラロックR.C.
ハリスJ.B.
Dziejman M.
リーX.
カーン A.I.
ファルケA.S.G.
ファルケ・S.M.
ネールG.B.
ライアン E.T.
カドリ F.
et al.
ヒト感染初期および後期における患者検体から直接回収したVibrio choleraeの転写プロファイリング。
Infect. Immun. 2005; 73: 4488-4493https://doi.org/10.1128/IAI.73.8.4488-4493.2005
記事で見る
スコープス (89)
PubMed
クロスフィルム
グーグル奨学生
ニールセン A.T.
ドルガノフN.A.
ラスムセンT.
オットーG.
ミラーM.C.
フェルトS.A.
トーレイユS.
スクールニックG.K.
腸内におけるビブリオコレラの病原性遺伝子発現は、双安定スイッチと解剖学的部位が制御している。
PLoS Pathog. 2010; 6: e1001102https://doi.org/10.1371/journal.ppat.1001102
記事で見る
スコープス (80)
PubMed
クロスフィルム
グーグル奨学生
ジェナルU.
レインダースA.
ロリC.
サイクリックdi-GMP:セカンドメッセンジャー(第二のメッセンジャー)。
Nat. Rev. Microbiol. 2017; 15: 271-284https://doi.org/10.1038/nrmicro.2016.190
記事で見る
スコープス(518)
パブコメ
クロスレフ
グーグル奨学生
ツィッツァーA.
ツィッツァーO.
バクディS.
パーマー M.
Vibrio cholerae cytolysinのオリゴマー化は5量体ポアを形成し、標的膜のコレステロールとスフィンゴ脂質に二重の特異性を持つことがわかった。
J. Biol. Chem. 1999; 274: 1375-1380https://doi.org/10.1074/jbc.274.3.1375
記事で見る
スコープス (97)
PubMed
概要
全文
全文PDF
Google Scholar
ツィッツァー A.
ワッセナーT.M.
ワレフ I.
Bhakdi S.
Vibrio choleraeサイトリジンのヒト腸管細胞に対する強力な膜透過作用と殺細胞作用。
Infect. Immun. 1997; 65: 1293-1298https://doi.org/10.1128/iai.65.4.1293-1298.1997
記事で見る
スコープス (54)
PubMed
クロスフィルム
グーグル奨学生
生貝浩之
赤塚明彦
辻山博啓
中江哲也
島村敏明.
Vibrio cholerae O1 の El Tor ヘモリシンによる膜損傷機構.
Infect. Immun. 1996; 64: 2968-2973https://doi.org/10.1128/iai.64.8.2968-2973.1996
記事で見る
スコープス (60)
PubMed
クロスフィルム
グーグル奨学生
オルソンR.
グオー E.
Vibrio cholerae cytolysin (VCC)プロトキシンの結晶構造および7量体膜貫通孔へのアセンブリ。
J. Mol. Biol. 2005; 350: 997-1016https://doi.org/10.1016/j.jmb.2005.05.045
記事で見る
スコープス (108)
PubMed
クロスフィルム
グーグル奨学生
ミレー Y.A.
アルヴァレスD.
リングゴーS.
フォン・アンドリアン・U.H.
デイビス・B.M.
ウォルドーM.K.
蛍光標識細菌のモニタリングからVibrio choleraeの腸内コロニー形成に関する知見を得た。
PLoS Pathog. 2014; 10: e1004405https://doi.org/10.1371/journal.ppat.1004405
記事で見る
スコープス(125)
パブコメ
クロスフィルム
グーグル奨学生
アミンA.
アリA.
クルナサンS.
チョンT.G.
アルジャシャミーK.A.
ジャアファル H.
ザイヌディン・Z.F.
ラヴィチャンドランM.
Lalitha P.
ウサギ腸管粘膜におけるVibrio cholerae O1 El TorおよびO139 Bengal感染症の病理組織学的特徴の比較。
Histol. Histopathol. 2009; 24: 559-565https://doi.org/10.14670/HH-24.559
記事で見る
PubMed
クロスフィルム
グーグル奨学生
Fu Y.
ホー B.T.
メカラノスJ.J.
Vibrio cholerae感染時の細胞間相互作用の追跡から、腸内マイクロ環境における細菌集団の動態を明らかにした。
Cell Host Microbe. 2018; 23: 274-281.e2https://doi.org/10.1016/j.chom.2017.12.006
記事で見る
スコープス(30)
PubMed
要旨
全文
全文PDF
Google Scholar
クレブス S.J.
テイラーR.K.
幼児マウスモデルにおける毒素制御ピルスを介したVibrio choleraeの保護と付着。
J. Bacteriol. 2011; 193: 5260-5270https://doi.org/10.1128/JB.00378-11
記事で見る
スコープス (70)
PubMed
クロスフィルム
グーグル奨学生
アダムスD.W.
シュトゥッツマンS.
シュトゥットマンC.
ブロケシュ M.
Vibrio choleraeのDNA取り込みピリはキチンのコロニー形成に必要であり、配列特異的な自己相互作用によってキンを認識することができる。
Nat. Microbiol. 2019; 4: 1545-1557https://doi.org/10.1038/s41564-019-0479-5
記事で見る
スコープス(43)
パブコメ
クロスレフ
グーグル奨学生
カーン T.J.
ラファティM.J.
サンドゥC.M.
テイラー R.K.
Vibrio choleraeの腸管コロニー形成とマイクロコロニー形成に必要なピリンドメインの特定。
Mol. Microbiol. 2000; 35: 896-910https://doi.org/10.1046/j.1365-2958.2000.01764.x
記事で見る
スコープス (167)
PubMed
クロスフィルム
グーグル奨学生
キアヴェリD.A.
マーシュJ.W.
テイラーR.K.
コレラ菌のマンノース感受性ヘマグルチニンは動物プランクトンへの付着を促進する。
Appl. Environ. Microbiol. 2001; 67: 3220-3225https://doi.org/10.1128/AEM.67.7.3220-3225.2001
記事で見る
スコープス (168)
パブコメ
クロスフィルム
グーグル奨学生
ワトニックP.I.
フルナーK.J.
コルターR.
Vibrio cholerae El Torによるバイオフィルム形成におけるマンノース感受性ヘマグルチニンの役割。
J. Bacteriol. 1999; 181: 3606-3609https://doi.org/10.1128/jb.181.11.3606-3609.1999
記事で見る
スコープス (230)
パブコメ
クロスフィルム
グーグル奨学生
テリン K.H.
テイラーR.K.
Vibrio cholerae O1 El Tor biotypeおよびO139株のコロニー形成には、マンノース感受性ヘマグルチニンではなく、毒素制御型ピルスが必要である。
Infect. Immun. 1996; 64: 2853-2856https://doi.org/10.1128/iai.64.7.2853-2856.1996
記事で見る
スコープス (252)
PubMed
クロスフィルム
グーグル奨学生
カーン T.J.
テイラーR.K.
TcpFはE1 Torおよび古典的なO1およびO139 Vibrio cholerae血清群によって分泌される可溶性コロニー形成因子および保護抗原である。
Infect. Immun. 2005; 73: 4461-4470https://doi.org/10.1128/IAI.73.8.4461-4470.2005
記事で見る
スコープス (43)
PubMed
クロスフィルム
グーグル奨学生
カーン T.J.
ジュードB.A.
テイラーR.K.
Vibrio choleraeの環境生存とヒト感染を結びつけるコロニー形成因子。
Nature. 2005; 438: 863-866https://doi.org/10.1038/nature04249
記事で見る
スコープス (241)
PubMed
クロスフィルム
グーグル奨学生
クラクラー A.M.
ハム H.
オルトK.
外膜接着因子多価接着分子7は、グラム陰性病原体による感染時に宿主細胞との結合を開始する。
Proc. Natl. Acad. Sci. USA. 2011; 108: 11614-11619https://doi.org/10.1073/pnas.1102360108
記事で見る
スコープス (93)
パブコメ
クロスフィルム
グーグル奨学生
サイード・K.A.
ベイハンS.
コレアN.
クイーンJ.
リュー J.
Peng F.
サッチェル K.J.F.
Yildiz F.
クロース K.E.
Vibrio choleraeの旗状体制御階層は病原性因子の発現を制御する。
J. Bacteriol. 2009; 191: 6555-6570https://doi.org/10.1128/JB.00949-09
記事で見る
スコープス (133)
PubMed
クロスフィルム
グーグル奨学生
スペランディオ V.
ジロンJ.A.
シルベイラW.D.
ケーパーJ.B.
Vibrio choleraeの付着因子であるOmpU外膜蛋白質。
Infect. Immun. 1995; 63: 4433-4438https://doi.org/10.1128/iai.63.11.4433-4438.1995
記事で見る
スコープス (135)
PubMed
クロスフィルム
グーグル奨学生
マッツ C.
バーグフェルドT.
ライスS.A.
Kjelleberg S.
マイクロコロニー、クオラムセンシング、細胞毒性は、原生動物の放牧にさらされた緑膿菌のバイオフィルムの生存を決定する。
Environ. Microbiol. 2004; 6: 218-226https://doi.org/10.1111/j.1462-2920.2004.00556.x
記事で見る
スコープス (153)
パブコメ
クロスフィルム
グーグル奨学生
イェンセン P.Ø.
ビャーンショルトT.
フィップスR.
ラスムセンT.B.
カルムH.
クリストファーセン L.
モーザー C.
ウィリアムズ P.
プレスラー T.
ジブスコフM.
Høiby N.
多形核白血球の急速な壊死は、緑膿菌のクオラムセンシング制御によるラムノリピッドの産生によって引き起こされる(Pseudomonas aeruginosa.
Microbiology. 2007; 153: 1329-1338https://doi.org/10.1099/mic.0.2006/003863-0
記事で見る
スコープス(299)
パブコメ
クロスフィルム
グーグル奨学生
ヴァン・ジェニップM.
クリステンセンL.D.
アルヘデM.
フィップスR.
イェンセンP.Ø.
クリストファーセン L.
パンプ S.J.
モーザー C.
ミケルセンP.J.
コウ A.Y.
他
緑膿菌のrhlA遺伝子の不活性化によりラムノリピッドの産生が阻害され、多形核白血球に対する防御が無効化される。
APMIS. 2009; 117: 537-546https://doi.org/10.1111/j.1600-0463.2009.02466.x
記事で見る
スコープス (142)
PubMed
クロスフィルム
グーグル奨学生
アルヘデ M.
ビャーンショルトT.
イェンセンP.Ø.
フィップスR.K.
モーザーC.
クリストファーセン L.
クリステンセンL.D.
ヴァン・ジェンニップM.
パルセック M.
ホワイビーN.
他
緑膿菌は、多形核白血球の存在を認識し、積極的に反応する。
Microbiology. 2009; 155: 3500-3508https://doi.org/10.1099/mic.0.031443-0
記事で見る
スコープス (175)
パブコメ
クロスフィルム
グーグル奨学生
ラマムルシー T.
ナンディR.K.
Mukhopadhyay A.K.
ダッタ S.
ムトレジャ A.
Okamoto K.
ミヨシ・S.I.
ネールG.B.
Ghosh A.
Vibrio choleraeの病原性における病原性制御と自然宿主応答.
Front. Cell. Infect. Microbiol. 2020; 10: 572096-572122https://doi.org/10.3389/fcimb.2020.572096
記事で見る
スコープス(25)
パブコメ
クロスフィルム
グーグル奨学生
カドリ F.
ジョンソンG.
ベガムY.A.
ウェンナーオズC.
アルベルト M.J.
サラムM.A.
スヴェンナーホルムA.M.
Vibrio cholerae O1およびO139によるコレラ患者におけるマンノース感受性ヘマグルチニンに対する免疫応答。
Clin. Diagn. Lab. Immunol. 1997; 4: 429-434https://doi.org/10.1128/cdli.4.4.429-434.1997
記事で見る
スコープス (40)
PubMed
クロスフィルム
グーグル奨学生
シャオ A.
リウ・Z.
ジョエルソン A.
Zhu J.
ビブリオコレラエの病原性制御因子による宿主免疫の回避機構
Proc. Natl. Acad. Sci. USA. 2006; 103: 14542-14547https://doi.org/10.1073/pnas.0604650103
記事で見る
スコープス (74)
PubMed
クロスフィルム
グーグル奨学生
シャオ A.
トスカーノK.
Zhu J.
Vibrio choleraeのプロコロニーピリ生合成系とアンチコロニーピリ生合成系の間の転写後クロストーク。
Mol. Microbiol. 2008; 67: 849-860https://doi.org/10.1111/j.1365-2958.2007.06091.x
記事で見る
スコープス (20)
PubMed
クロスフィルム
グーグル奨学生
Fu Y.
ウォルドーM.K.
メカラノスJ.J.
ビブリオコレラエの腸内コロニー形成に関するTn-seq解析から、宿主におけるT6SSを介した抗菌活性の役割が明らかになった。
Cell Host Microbe. 2013; 14: 652-663https://doi.org/10.1016/j.chom.2013.11.001
記事で見る
スコープス (180)
PubMed
概要
全文
全文PDF
グーグル奨学生
オリビエV.
クイーンJ.
サッチェル K.J.F.
Vibrio choleraeによる成体マウスの小腸コロニー形成の成功には、ケタミン麻酔とアクセサリー毒素が必要である。
PLoS One. 2009; 4: e7352https://doi.org/10.1371/journal.pone.0007352
記事で見る
スコープス (66)
PubMed
クロスフィルム
グーグル奨学生
アベル S.
ワルドーM.K.
下痢性疾患の乳幼児ウサギモデル。
Curr. Protoc. Microbiol. 2015; 38: 6A.6.1-6A.6.15https://doi.org/10.1002/9780471729259.mc06a06s38
記事で見る
スコープス(9)
パブコメ
クロスフィルム
グーグル奨学生
シュルテ・L.N.
シュバインリンM.
ウェスターマン A.J.
ヤンガ H.
サントスS.C.
アッペンツェラーS.
ウォレス H.
フォーゲル J.
メッツガー M.
サルモネラ菌感染時の宿主と病原体のコンパートメント戦略を明らかにする先進的ヒト腸管共培養モデル。
mBio. 2020; 11: 033488-19https://doi.org/10.1128/mBio.03348-19
記事で見る
スコープス (19)
クロスフィルム
グーグル・スカラー
フロイドK.A.
リーC.K.
Xian W.
ナメタラM.
バレンタインA.
クレイア B.
朱 S.
ヒューズ・H.Q.
Chlebek J.L.
ウー・D.C.
et al.
Vibrio choleraeのIV型MSHAピルスの引き込みと表面付着にc-di-GMPを調節する。
Nat. Commun. 2020; 11: 1549https://doi.org/10.1038/s41467-020-15331-8
記事で見る
スコープス (44)
パブコメ
クロスフィルム
グーグル奨学生
サンD.
ラファティM.J.
ピークJ.A.
テイラー R.K.
Vibrio cholerae toxin coregulated pilin subunit内の細菌コロニー形成を媒介するドメイン。
Gene. 1997; 192: 79-85https://doi.org/10.1016/S0378-1119(97)00007-3
記事で見る
スコープス (32)
PubMed
クロスフィルム
グーグル奨学生
エーデルシュタイン A.D.
土田真也
アモダージN.
ピンカードH.
ベール・R.D.
Stuurman N.
μManagerソフトウェアを用いた顕微鏡制御の高度な方法。
J. Biol. Methods. 2014; 1: 10https://doi.org/10.14440/jbm.2014.36
記事で見る
パブコメ
クロスフィルム
グーグル奨学生
ドビンA.
デイビスC.A.
シュレシンジャーF.
ドレンコウJ.
ザレスキーC.
ジャ S.
バトゥット P.
シャイソンM.
ジンゲラス T.R.
STAR:超高速ユニバーサルRNA-seqアライナー。
Bioinformatics. 2013; 29: 15-21https://doi.org/10.1093/bioinformatics/bts635
記事で見る
スコープス (20725)
パブコメ
クロスフィルム
グーグル奨学生
Choi M.
チャン・C.-Y.
クラウ T.
ブルディD.
キリーンT.
マクリーン B.
Vitek O.
MSstats:定量的質量分析に基づくプロテオミクス実験の統計解析のためのRパッケージ。
Bioinformatics. 2014; 30: 2524-2526https://doi.org/10.1093/bioinformatics/btu305
記事で見る
スコープス (550)
PubMed
クロスフィルム
グーグル奨学生
ペレス・リベロル Y.
バイ・J.
バンドラC.
ガルシア-セイスデドスD.
ヘワパティラナS.
カマッチナタン S.
クンドゥ D.J.
プラカシュ A.
フレリックス=ジッパー A.
アイゼナッカーM.
et al.
2022年のPRIDEデータベースリソース:質量分析に基づくプロテオミクスエビデンスのハブ。
Nucleic Acids Res. 2022; 50: D543-D552https://doi.org/10.1093/nar/gkab1038
記事で見る
スコープス (1007)
PubMed
クロスフィルム
グーグル奨学生
オー H.
シアーノB.
ダイヤモンドS.
好中球の分離プロトコール。
J. Vis. Exp. 2008; : 745https://doi.org/10.3791/745
記事で見る
スコープス (146)
パブコメ
クロスフィルム
グーグル奨学生
プレゲスエロス-マンサノC.
プシュホフJ.
ファンデンブリンクS.
ゲールト V.
ビューマー J.
クレバース H.
成体幹細胞由来のヒト腸管オルガノイドの樹立と培養。
Curr. Protoc. Immunol. 2020; 130: e106https://doi.org/10.1002/cpim.106
記事で見る
スコープス (44)
パブコメ
クロスフィルム
グーグル奨学生
シュターブ J.F.
レム=デュミットJ.M.
ラタニッチR.
パセッティM.F.
ザコス・N.C.
ヒト腸管/コロノイドと自然免疫細胞との共培養システム。
Curr. Protoc. Immunol. 2020; 131: e113https://doi.org/10.1002/cpim.113
記事で見る
スコープス (26)
パブコメ
クロスフィルム
グーグル奨学生
ギブソンD.G.
ヤングL.
チュアン・R.Y.
ベンターJ.C.
ハッチソンC.A.
スミス H.O.
数百キロベースまでのDNA分子を酵素的に組み立てる。
Nat. Methods. 2009; 6: 343-345https://doi.org/10.1038/nmeth.1318
記事で見る
スコープス (5680)
パブコメ
クロスフィルム
グーグル奨学生
スコルプスキ K.
テイラーR.K.
対立遺伝子交換のためのポジティブセレクションベクター。
Gene. 1996; 169: 47-52https://doi.org/10.1016/0378-1119(95)00793-8
記事で見る
スコープス (375)
PubMed
クロスフィルム
グーグル奨学生
ペデラックJ.D.
カバントゥスS.
トラン T.
ターウィリガーT.C.
ワルドG.S.
スーパーフォルダー緑色蛍光タンパク質のエンジニアリングと特性評価。
Nat. Biotechnol. 2006; 24: 79-88https://doi.org/10.1038/nbt1172
記事で見る
スコープス (1481)
PubMed
クロスフィルム
グーグル奨学生
ジャイティンD.A.
ケニグスバーグE.
ケレンショールH.
エレファントN.
ポール・F.
ザレツキーI.
ミルドナーA.
コーエン N.
ユング S.
タネイ A.
アミットI.
超並列シングルセルRNA-seqによるマーカーフリーの組織から細胞タイプへの分解。
Science. 2014; 343: 776-779https://doi.org/10.1126/science.1247651
記事で見る
スコープス (1151)
パブコメ
クロスフィルム
グーグル奨学生
ブルネル・J.L.
グリーンR.
一次元SDS-ポリアクリルアミドゲル電気泳動(1D SDS-PAGE)。
Methods Enzymol. 2014; 541: 151-159https://doi.org/10.1016/B978-0-12-420119-4.00012-4
記事で見る
スコープス (111)
PubMed
クロスフィルム
グーグル奨学生
ウィシュニエフスキーJ.R.
ゴーガズ F.Z.
プロテオミクス解析のための高速・高感度な総タンパク質・ペプチドアッセイ。
Anal. Chem. 2015; 87: 4110-4116https://doi.org/10.1021/ac504689z
記事で見る
スコープス(166)
パブコメ
クロスレフ
グーグル奨学生
アールネー E.
グラッターT.
ビガノC.
シューベルト C.v.
ニグE.A.
シュミット A.
アイソバリックマスタグを用いたタンパク質定量実験における定量精度の評価と改善。
J. Proteome Res. 2016; 15: 2537-2547https://doi.org/10.1021/acs.jproteome.6b00066
記事で見る
スコープス(88)
パブコメ
クロスフィルム
グーグル奨学生
ホアン・D.W.
シャーマンB.T.
Lempicki R.A.
DAVIDバイオインフォマティクスリソースを用いた大規模遺伝子リストの系統的・統合的解析。
Nat. Protoc. 2009; 4: 44-57https://doi.org/10.1038/nprot.2008.211
記事で見る
スコープス (25954)
パブコメ
クロスフィルム
グーグル奨学生
シャーマン B.T.
ハオ M.
Qiu J.
Jiao X.
ベースラーM.W.
レーン H.C.
Imamichi T.
Chang W.
DAVID: a web server for functional enrichment analysis and functional annotation of gene list (2021 update).
Nucleic Acids Res. 2022; 50: W216-W221https://doi.org/10.1093/nar/gkac194
記事で見る
スコープス (488)
PubMed
クロスフィルム
グーグル奨学生
コング A.T.
レプレヴォストF.v.
アフトノモフD.M.
メラチャーヴD.
Nesvizhskii A.I.
MSFragger:質量分析に基づくプロテオミクスにおける超高速かつ包括的なペプチド同定。
Nat. Methods. 2017; 14: 513-520https://doi.org/10.1038/nmeth.4256
記事で見る
スコープス (544)
パブコメ
クロスレフ
グーグル奨学生
ハルトマンR.
イェッケルH.
ジェリ E.
シン・P.K.
ヴァイディヤS.
バイエル M.
ローデ D.K.H.
ヴィダコヴィッチ・L.
ディアス=パスカルF.
フォン・J.C.N.
ら。
BiofilmQによる微生物群集の定量的画像解析。
Nat. Microbiol. 2021; 6: 151-156https://doi.org/10.1038/s41564-020-00817-4
記事で見る
スコープス(99)
パブコメ
クロスフィルム
グーグル奨学生
イェッケル H.
Drescher K.
細菌細胞・群集の画像解析の進歩と可能性。
FEMS Microbiol. Rev. 2020; 45: 1-14https://doi.org/10.1093/femsre/fuaa062
記事で見る
スコープス(30)
クロスフィルム
グーグル奨学生
大津直樹.
グレーレベル・ヒストグラムからの閾値選択法.
IEEE Trans. Syst. Man Cybern. 1979; 9: 62-66https://doi.org/10.1109/tsmc.1979.4310076
記事で見る
スコープス (29995)
クロスフィルム
Google Scholar
記事情報
出版履歴
出版されました: 2023年6月8日
受理された: 2023年5月9日
改訂版受理 2023年1月26日
受理された: 2021年9月15日
識別情報
DOI: https://doi.org/10.1016/j.cell.2023.05.008
著作権について
© 2023 The Author(s). 発行:エルゼビア・インク
ユーザーライセンス
クリエイティブ・コモンズ 表示 - 非商用 - 改変禁止 (CC BY-NC-ND 4.0)|Creative Commons Attribution - NonCommercial - NoDerivs
再利用の方法
サイエンスダイレクト
ScienceDirectでこの論文にアクセスする
図版
グラフの概要
図1V. choleraeがヒト白血球上にバイオフィルムを形成し、その後、免疫細胞死とバイオフィルムの飛散が起こる。
図S1コレラ菌によるヒト血液から分離したCD14+単球から分化したマクロファージのコロニー形成と死、および初代CD14+単球への菌の付着(図1、2、3、5を参照
図 S2コレラ菌バイオフィルム形成動態およびマクロファージ死に対する初期細菌数およびマクロファージ数の影響、コレラ菌増殖に対するマクロファージ溶解液の影響、単球死に対するコレラ菌の影響、共培養中のコレラ菌とマクロファージの互いの反応、図 1 および 5 を参照のこと。
図2マクロファージ表面へのコレラ菌の付着はIV型ピリによって可能になる
図3ΔmshA細胞の付着とバイオフィルム形成に及ぼすTC piliとTcpFの影響、マクロファージ上に形成された成熟V. cholerae WTバイオフィルム内のRbmA、Bap1、RbmC、およびvps遺伝子の転写、図2および図3を参照。
図3マクロファージ上に形成されたバイオフィルムの細胞外マトリックスは、MSHA pili、TC pili、TcpFからなり、それぞれ異なるバイオフィルム機能を発揮する。
図4バイオフィルムの分散は、細胞内のc-di-GMPレベルとTC piliの存在によって決定される
図5細菌バイオフィルムによるマクロファージの被包は、マクロファージの細胞死を増加させる
図S4毒素HlyAがマクロファージ死を引き起こす主な原因である、図5を参照
図6V. cholerae細胞はヒト腸管上皮バリアを破り、その後、その下のマクロファージにバイオフィルムを形成する
図S5異なるMOI、異なる細菌変異体、および異なるマクロファージに対するV. choleraeの腸管マクロファージ共培養モデルへの感染、図6参照
図S6異なるV. cholerae株とTHP-1マクロファージおよびドナー#2からのヒト腸管単層との共培養結果、図6を参照。
関連記事
本サイトのコンテンツは、あらゆる分野の医療従事者や研究者を対象としています。
研究雑誌
細胞
癌細胞
細胞化学生物学
細胞ゲノミクス
細胞宿主と微生物
細胞代謝
セルレポート
セルレポートメディスン
セルレポートメソッド
セルレポート 物理科学
セルレポート サステイナビリティ
細胞幹細胞
細胞システム
化学
化学触媒
カレントバイオロジー
発生細胞
デバイス
ヘリオン
イミュニティ
アイサイエンス
ジュール
物質
医学
分子細胞
ニューロン
一つの地球
パターン
STAR プロトコル
構造
トレンドレビュー誌
生物化学
バイオテクノロジー
癌
細胞生物学
化学
認知科学
エコロジー&エボリューション
内分泌学・代謝学
遺伝学
免疫学
微生物学
分子医学
神経科学
寄生虫学
薬理学
植物科学
パートナージャーナル
AJHG
生物物理学雑誌
生物物理学レポート
HGGアドバンス
モレキュラープラント
分子療法ファミリー
ネクサス
植物通信
幹細胞レポート
イノベーション
コレクション
ベスト・オブ・セルプレス
セルプレスレビュー
セルプレスセレクション
コンソーシアムハブ
Nucleus Collections
スナップショット・アーカイブ
ジャーナルを越えて
Cellキャリアネットワーク
セルメンター
細胞シンポジューム
ラボリンク
ウェビナー
記事を進化させる
コミュニティレビュー
Figure360
スニークピーク
STARメソッド
サイエンス・イン・ソサイエティ
セル画展
セルプレスポッドキャスト
セルプレスビデオ
ぬりえ・コミック
リサーチアーク
コネクト
セルプレスについて
採用情報
お問い合わせ
ヘルプ&サポート
ニュースルーム
出版物のお知らせ
アクセス
購読する
今すぐ読む
図書館に薦める
INFORMATION
広告主様向け
採用担当者様へ
図書館員の方へ
ご利用条件
個人情報保護方針
アクセシビリティ
当社は、サービスの提供・向上およびコンテンツのカスタマイズのためにクッキーを使用しています。クッキーの設定を変更するには、本サイトのクッキー設定にアクセスしてください。
著作権 © 2023 Elsevier Inc.ただし、第三者が提供する一部のコンテンツを除く。
この記事が気に入ったらサポートをしてみませんか?
