SARS-CoV-2のスパイクタンパク質の凝集は、細菌のリポポリサッカライドによって引き起こされる
SARS-CoV-2のスパイクタンパク質の凝集は、細菌のリポポリサッカライドによって引き起こされる
Jitka Petrlova,corresponding著者1 Firdaus Samsudin, 2 Peter J. Bond, 2 , 3 and Artur Schmidtchen 1 , 4 , 5
著者情報 記事の注釈 著作権とライセンス情報 免責事項
関連データ
データの利用可能性に関する声明
移動します。
概要
SARS-CoV-2のスパイク(S)タンパク質は、COVID-19のウイルス侵入に重要である。ここでは、リポポリサッカライド(LPS)が、LPSとSタンパク質の高用量でSタンパク質の凝集を誘発することを示した。我々は、顕微鏡分析、凝集およびゲルシフトアッセイにより、Sタンパク質凝集体の形成を実証した。LPSを高濃度で投与すると、Sタンパク質凝集体の形成が促進されることが、凝集タンパク質に特異的に結合するamytrackerとチオフラビンT色素によって検出された。我々は、エンドトキシンを消去するトロンビン由来のペプチドTCP-25による凝集体の形成を阻害することで、LPSの役割を検証した。また、分子シミュレーションにより、Sタンパク質の凝集しやすい配列がLPSの結合部位の近くにあることが予想され、Sタンパク質とLPSの高次オリゴマーが安定に形成されることが示された。これらの結果は、LPSによるS蛋白質の凝集を証明するものである。
キーワード COVID-19、エンドトキシン、炎症、リポポリサッカライド、タンパク質凝集、スパイクタンパク質
戻る
概要
SARS-CoV-2スパイク蛋白による炎症には、細菌性リポ多糖(LPS)が中心的な役割を担っている。我々はこれまでに、スパイクタンパク質がLPSに結合することを明らかにしてきた。今回、我々は、LPSとスパイクタンパク質の高ミクロモル濃度において、スパイク-LPS複合体が大きな凝集体を形成することを明らかにした。本研究は、サブナノモル濃度の低いスパイクとLPSがToll様受容体4(TLR4)を介した炎症反応を高めるという、これまでの知見を補完するものである。

画像やイラストなどを保持する外部ファイル。
オブジェクト名は、FEB2-9999-0-g001.jpg。
に移動します。
略語について
A3D, アグレスキャン3D
ARDS, 急性呼吸窮迫症候群
BNゲル、ブルーネイティブポリアクリルアミドゲル
CG、粗視化
cryo-EM, 低温電子顕微鏡法
CTD2、C末端ドメイン2
ECD、エクトドメイン
LPS, リポポリサッカライド
MD, 分子動力学
NTD, N-末端ドメイン
RBD、受容体結合ドメイン
RMSD, 二乗平均平方根偏差
Sタンパク質、スパイクタンパク質
TCP-25、トロンビン由来ペプチド
TEM, Transmission electron microscopy(透過型電子顕微鏡
ThT, チオフラビンTアッセイ
TLR4, Toll様受容体4
スパイク(S)タンパク質は、COVID-19の原因となるSARS-CoV-2のようなエンベロープ型陽性鎖RNAウイルスの外表面に突起を形成している[1]。Sは、受容体との結合に関わるS1サブユニットと膜融合を担うS2サブユニットからなる大きなエクトドメイン(ECD)を持つ膜タンパク質である。Sタンパク質が細菌のリポポリサッカライド(LPS)と直接相互作用することを示す研究が増えてきています[2, 3, 4, 5]。LPSはToll様受容体4(TLR4)経路を活性化し、サイトカイン、急性期タンパク質、活性酸素の大量放出を誘発することができます。COVID-19患者の罹患率および死亡率は、典型的には、重度の全身性炎症および急性呼吸窮迫症候群(ARDS)の結果である[6、7]。ARDSは、肺炎、敗血症、重度の火傷、または外傷の際によく見られる全身性の炎症反応である。LPSによって刺激されるTLR4を含むToll様受容体は、ARDSの間に活性化されます。したがって、ARDSと重症COVID-19患者の臨床症状は、疾患中の病態生理において類似しています[8, 9]。
我々は以前の研究で、Sタンパク質がS1およびS2サブユニット上の複数の部位を介してLPSと結合することができ、このような超低レベルのLPSとの相互作用がin vitroおよびin vivoで炎症反応を促進することを示した[2, 5]。S1サブユニットでは、LPSはN末端ドメイン(NTD)と受容体結合ドメイン(RBD)上の暗号ポケットに結合し、このポケットは他の疎水性分子と結合することが低温電子顕微鏡(cryo-EM)により以前に示されていた[10]。S2サブユニットでは、LPSはSタンパク質単量体の間の大きな溝に結合する。興味深いことに、Sタンパク質ベースのCOVID-19ワクチン候補の低温電子顕微鏡構造から、Sタンパク質は3量体の2量体や3量体の3量体といった高次オリゴマーを形成することもでき、Sタンパク質凝集の可能性を示唆しています[11]。実際、生物物理学的・生化学的手法を用いて、LPSに超低濃度ナノモル以上のSタンパク質を添加すると、高分子量の複合体を形成するきっかけとなることを以前に明らかにした[2]。しかしながら、LPSがSタンパク質の凝集に及ぼす影響やその分子機構は未だ不明である。
我々は、高用量のLPSによって引き起こされるSタンパク質の凝集を探索した。その結果、LPSはS蛋白質単独よりも大きなS蛋白質凝集体を形成することが分かりました。さらに、抗毒性ペプチドTCP-25でLPSトリガー効果を阻害することにより、凝集がLPSに依存していることを検証した。計算機による構造解析の結果、LPS結合部位の近傍のループなど、Sタンパク質に凝集しやすい領域があることがわかった。さらに、粗視化分子動力学(MD)シミュレーションにより、高次のSタンパク質オリゴマーがLPSと安定な複合体を形成することが確認された。
次へ
材料と方法
タンパク質とペプチド
SARS-CoV-2 Sタンパク質はACROBiosystems (Newark, DE, USA)によって製造された。SARS-CoV-2 Sタンパク質の配列は、AA Val 16 - Pro 1213 [Accession # QHD43416.1 (R683A, R685A)] を含んでいる。トロンビン由来ペプチドTCP-25 (GKYGFYTHVFRLKKWIQVIDQFGE) (97% purity, acetate salt) はAmbiopharm (Madrid, Spain) によって合成されたものである。
透過型電子顕微鏡観察
Sタンパク質(1μm)を大腸菌由来のLPS(50μm)または緩衝液のみと37℃で30分間インキュベートした後、透過型電子顕微鏡(TEM)(Jeol Jem 1230; Jeol, Tokyo, Japan)を用いて、ネガティブ染色と組み合わせて画像を撮った。マウントした試料について、4つの独立した試料調製から、グリッド(ピッチ62μm)上に10視野を調べた。試料はカーボンコートしたグリッド(Copper mesh, 400)に60秒間吸着させ、7μLの2%酢酸ウラニルで20秒間染色した。凝集体の大きさは,すべての画像を 8bit に変換し,閾値を調整した後,imagej 1.52 k によるグレー値/μm2 ± SEM の平均値として解析した[12].
蛍光顕微鏡観察
LPS(50μm)で30分間チャレンジしたSタンパク質(1μm)のアミロイド形成を37℃で可視化するために、Amytracker 680(Ebba Biotech, Lund, Sweden)による染色を行った。続いて、サンプル(20μL)を、チューブ内の50μLのAmytracker 680(ストック溶液からの1000×希釈)と共に、25℃でさらに30分間インキュベートした。次に、サンプルを(l-リジン)コートしたスライドグラス(Thermo Scientific, Braunschweig, Germany)に移し、洗浄後、蛍光マウント媒体(Molecular Probes; Life Technologies, Eugene, OR, USA)で顕微鏡スライドにマウントした。Zeiss AxioScope (Oberkochen, Germany) A.1 蛍光顕微鏡を使用して、3 つの独立したサンプル調製から 10 視野 (1 × 1 mm) を調べた (対物レンズ。Zeiss EC Plan-Neofluar 40×; camera: Zeiss AxioCam MRm、取得ソフトウェア。Zeiss Zen 2.6 [青版])。凝集体の大きさは、全ての画像を8bitに変換し、閾値を調整した後、imagej 1.52 kにより、グレー値/μm2±SEMの平均値として解析した。
ブルーネイティブポリアクリルアミドゲル電気泳動とウェスタンブロット
10マイクロリットルのSタンパク質(1μm)を、コントロールとして10mm Tris、LPS(50μm)、またはLPS(50μm)およびTCP-25(2および10μm)のいずれかと混合した。サンプルは、ローディングバッファー(4×Loading Buffer Native Gel, cat#BN2003; Life Technologies)と混合する前に37℃で30分間インキュベートし、続いて4-16% Bis-Tris Native Gels (cat#BN1002BOX; Life Technologies) にロードした。サンプルはマーカー(Native Marker Unstained Protein Standard, cat#LC0725; Life Technologies)と並行して150Vで90分間反応させた。ゲルをTrans Turbo Blot system (Bio-Rad) を介して0.2 μm polyvinylidene fluoride (PVDF) membrane (Trans Blot Transfer Pack, cat #1704156, Bio-Rad, Hercules, CA, USA) にトランスファーした。その後、メンブレンを70%エタノールで脱染し、1×PBS-Tween(PBS-T)中の5%ミルクで室温で60分間ブロックした。この膜を、PBS-T中の5%無脂肪乳で1:1000に希釈した濃度のマウスmAb抗His tag(cat# MA1-21315; Thermo Scientific, Rochester, IL, USA)と共に、25℃で1時間インキュベートした。Sタンパク質とその高分子複合体は、5%牛乳で補った1×PBS-Tで1:2000に希釈した二次HRP結合ウサギ抗マウスポリクローナル(cat#P0260;Dako, Santa Clara, CA, USA)を加え、室温で60分間インキュベートした後に検出された。メンブレンを現像基質(Super Signal West Pico PLUS Chemiluminescent Substrate, cat#34580, Thermo Scientific)にインキュベートすると、バンドが観察された。シグナルはChemi-Docシステム(Bio-Rad)を用いて取得した。すべての実験は少なくとも4回行った[13].
チオフラビンTアッセイ
アミロイド形成は、色素のチオフラビンT (ThT)を用いて決定した。ThT (cat# T3516; Sigma, St. Louis, MO, USA) はアミロイド形成性タンパク質/ペプチドのβシート構造に優先的に結合する。凝集および凝集阻止の検討のため、測定前にSタンパク質(1μm)を大腸菌由来のLPS(50μm)またはTCP-25(2および10μm)と共に緩衝液(10 mm Tris, pH 7.4 )中で37℃、30分間インキュベートした。200μリットルの材料を100μm ThTとともに暗所で15分間インキュベートした(ThTストックは4℃で暗所に保存した1mm)。VICTOR3 Multilabel Plate Counter spectrofluorometer (PerkinElmer, Santa Clara, CA, USA) を用いて、励起450 nm、励起および放出スリット幅10 nmでThT蛍光を測定した。各サンプルのシグナルからバックグラウンド(10 mm Tris pH 7.4, LPS, TCP-25)を減算した[14].
S蛋白質凝集の予測
S タンパク質の凝集しやすい領域を特定するために、タンパク質凝集の構造ベース予測ウェブサーバ、Aggrescan3D (A3D) version 2.0 [15]を使用した。Sタンパク質三量体ECDの閉じた状態の低温電子顕微鏡構造(PDB: 6XR8) [16]をA3Dウェブサーバーにアップロードし、デフォルトパラメータを用いて3本すべての鎖を解析した。各残基の凝集スコアは3本の鎖の平均値をとり、低温電子顕微鏡構造にマッピングされた。脂質A分子は、PyMOLの構造アライメントを用いて結合ポケットに整列させた。
Sタンパク質と脂質Aとの凝集体のCG MDシミュレーション
Sタンパク質三量体凝集体の原子構造は、SARS-CoV-2のスパイク二量体三量体 (PDB: 7JJJ) と三量体三量体 (EMDB: 22355) の低温電子顕微鏡構造から得た [11].これらの構造は、膜貫通ドメインとヘプタッドリピート2ドメインを除いたSタンパク質のECDを表している。三量体構造については、個々のスパイクタンパク質三量体の原子構造をucsf chimeraを用いてEM密度マップにフィッティングした。二量体と三量体の原子構造は、タンパク質の二次構造を維持するために弾性ネットワークを適用したmartini 2.2力場を用いてCG表現に変換された[17]。脂質Aのパラメータは、以前の研究[18]から引用した。次に、脂質A分子を、構造アラインメント法 [2, 5]を用いて、以前の研究で説明した結合ポケット内の全鎖上のNTD、RBD、S2サブユニットにドッキングさせた。合計で、スパイク2量体の3量体は18個のリピドA分子と結合し、スパイク3量体の3量体は27個のリピドA分子と結合していた。次に、この系を標準的なMARTINI水粒子と0.15 m NaCl塩溶液で溶媒和した。衝突した粒子を移動させるために、最急降下エネルギー最小化が行われました。各系で10 nsの平衡化シミュレーションを行い、力定数1000 kJ mol-1 nm-2と500 kJ mol-1 nm-2の位置拘束を、それぞれペプチド骨格と脂質A分子に適用した。生産シミュレーションでは、各系で異なる開始速度を用いて、3回の独立した10μsシミュレーションを行った。温度は310 K、圧力は1 atmに保ち、速度再スケーリングサーモスタット [2] と Parrinello-Rahman barostat [19] を使用した。クーロン相互作用は反応場法を用いて計算し,ファンデルワールス相互作用はポテンシャルシフトVerlet法を用いて切断し,いずれも短距離距離を1.1 nmにカットオフした.どちらのシミュレーションも10fsの積分時間ステップを使用した。
統計解析
ThTアッセイ、TEM画像解析、蛍光顕微鏡画像解析、ブルーネイティブポリアクリルアミド(BN)ゲル画像解析のグラフは、少なくとも4つのSタンパク質独立実験からの平均±SEMとして提示されている。これらのアッセイにおける差異を、Dunnettの多重比較検定を伴う一元配置分散分析を用いて評価した。すべてのデータは、GraphPad Prism (GraphPad Software, Inc., San Diego, CA, USA)を用いて分析した。さらに、0.05未満のP値は、統計的に有意であるとみなされた(P < 0.05、P < 0.01、およびP < 0.001)。
に進む。
結果
Sタンパク質-LPS凝集体の可視化
Sタンパク質の凝集は、ネガティブステイン電子顕微鏡で可視化した(Fig. 1A)。画像解析の結果、LPS添加によりSタンパク質はより大きな凝集体を形成することが明らかになった(Fig.1B)。この結果は、アミロイド形成タンパク質/ペプチドのβシート構造に特異的に結合し、蛍光シグナルを発する色素Amytrackerを用いた蛍光顕微鏡で確認された(図1C,D)。

写真やイラストなどを保持する外部ファイル。
オブジェクト名はFEB2-9999-0-g002.jpg。
図1
LPSをトリガーとするSタンパク質凝集の可視化。(A) TEMネガティブ染色により、大腸菌由来のLPSとインキュベートした後、タンパク質の丸い凝集粒子(サイズ0.2〜2μm)を確認した。未処理のサンプルでは、より小さく丸みを帯びたタンパク質の凝集体も観察された(0.02〜0.2μm)。4つの独立した実験からの1つの代表的な画像を示す(n = 4)。スケールバーは5μmである。(B) TEM画像の解析は、imagej 1.52 kを使用して粒子のサイズを決定するために行われた。 (C) 蛍光顕微鏡 - Amytracker 680染色は、LPS(50μm)に曝露したときにSタンパク質凝集(1μm)が増加することを明らかにした。4つの独立した実験の1つの代表的な画像を示す(n = 4)。スケールバーは2μmである。挿入図は、4つの凝集体を4倍の高倍率で表したものである。(D) Sタンパク質-LPS凝集体におけるAmytracker 680シグナルの画像解析。粒子の大きさは、灰色値/μm±SEMの平均値として表される。グラフにおいて、各ポイントは、1つの実験につき少なくとも10枚の写真の平均測定値を表す。統計解析は、4つの独立した実験(n = 4)からT-testを使用して行った。**P ≤ 0.01.
TCP-25 は LPS 誘発 S タンパク質凝集を抑制する
TCP-25 はトロンビンの C 末端に由来するペプチドで、LPS と高い親和性で結合する[18]。S タンパク質の凝集が LPS 依存的であることを示すために、過剰の TCP-25 を添加した。チオフラビン T1 は、βシート構造の特徴を示すアミロイド生成タンパク質に特異的に結合する蛍光色素である。Sタンパク質の蛍光シグナルはLPS処理により著しく増加したが、TCP-25(2および10μm)はLPSによるSタンパク質の凝集を著しく阻害した(Fig. 2A)。

写真やイラストなどを保持する外部ファイル。
オブジェクト名は、FEB2-9999-0-g003.jpg。
図2
TCP-25によるSタンパク質凝集の阻害。(A) ThT凝集アッセイでは、50μmのLPSで処理したSタンパク質凝集体(1μm)の蛍光が有意に増加することが確認された。このタンパク質の凝集は、2μmと10μmのTCP-25ペプチドの添加により有意にブロックされた。(B) BNゲル/ウェスタンブロットアッセイでは、LPS(50μm)で処理した単量体Sタンパク質が著しく減少し、TCP-25の添加後に著しく増加することが示された。タンパク質-LPS相互作用は、2および10μmのTCP-25ペプチドの両方によって有意にブロックされた。画像は4つの独立した実験からの一例である(n = 4)。(C) LPSおよびTCP-25の存在下または非存在下でのSタンパク質単量体を検出する化学発光シグナル強度の画像解析。蛍光シグナルと強度は、平均値±SEMで表される。統計解析は、4つの独立した実験(n = 4)から、Dunnettの多重比較検定を伴う一元配置分散分析を用いて実施した。*P≦0.05、**P≦0.01、***P≦0.001。
さらに、LPSで処理したSタンパク質の凝集を、ネイティブブルーゲル/ウェスタンブロット解析で検証した。LPS存在下でのSタンパク質の高分子複合体形成を検出するために、非変性条件下で電気泳動を行った。Sタンパク質をLPSと混合すると、Sタンパク質の単量体(非集合体)の著しい減少が検出された。この単量体バンドは、Sタンパク質とLPSの混合物にTCP-25(2および10μm)を添加すると再び増加した(Fig. 2B)。画像解析の結果、LPS単独またはTCP-25を併用したSタンパク質単量体の有意な変化がそれぞれ認められた(Fig.2C)。これらの結果は、LPSがSタンパク質の凝集を誘導し、アミロイドの形成につながることを示している。
LPS結合部位の近くにあるSタンパク質の凝集しやすい領域
LPS存在下でのSタンパク質凝集の分子機構を理解するために、まず、A3Dウェブサーバー[15]を用いてSタンパク質上の凝集しやすい領域の候補を調査した。各Sタンパク質残基の凝集傾向スコア(正の値は凝集傾向が高いことを、負の値は凝集傾向が低いことを示唆)をFig.3Aに示す。正のスコアを持つ残基がいくつかあり、Sタンパク質の凝集に寄与している可能性がある。これらはNTD上のループ246-250とC末端ドメイン2(CTD2)付近のループ621-624である。SARS-CoV-2のSタンパク質ベースのワクチン候補の低温電子顕微鏡構造では、Sタンパク質はこれら2つのループ領域間の相互作用を介して高次の複合体を形成している[11]。ループ621-624は、我々が以前に同定し、SARS-CoV-2の変異体に見られるいくつかの変異の近くにある、潜在的に薬剤投与可能なクリプティックポケットの一部でもある[20]。興味深いことに、このループは、in vitroでSタンパク質を好中球エラスターゼと共培養したときにアミロイド線維を形成するペプチド601-620と隣接している[21]。さらに、2つのLPS結合ポケット、すなわちNTDポケットとRBDポケット[5]は、正の凝集傾向スコアを持ついくつかの領域に近接していることがわかった(図3B)。ループ170-176は、NTD上のLPS結合部位の上縁の一部である。このループと周囲の糖鎖分子は、LPS結合ポケットの開閉を媒介することが示されている[20]。RBDでは、残基364-368が「ゲーティング」ヘリックスを形成し、LPS結合を可能にするために離れていく[5]。RBDポケットへのLPSの高親和性結合は、この領域のヘリカル二次構造に依存している[5]。両方の凝集しやすい領域がLPS結合部位の近くにあり、Sタンパク質-LPS相互作用において重要な役割を担っていることから、LPS結合がこれらの残基を介してSタンパク質の凝集を調節している可能性が考えられる。

画像やイラストなどを保持する外部ファイル。
オブジェクト名は、FEB2-9999-0-g004.jpg。
図3
SARS-CoV-2のSタンパク質の凝集傾向。(A) A3D2.0ウェブサーバーによって予測されたSタンパク質3量体の凝集スコア。網掛け部分はNTDとRBDの位置を示す。点線の円は、我々の以前の研究[2, 5]で決定されたLPS結合に関与する残基を示す(青、NTDポケット、赤、RBDポケット、緑、S2ポケット)。(B) Sタンパク質三量体を、青(最も凝集しにくい)から赤(最も凝集しやすい)へと凝集スコアで色分けしたもの。拡大画像はLPS結合部位を示し、脂質A部位はオレンジ色の棒状で示されている。陽性スコアの高い領域はラベルで表示されている。
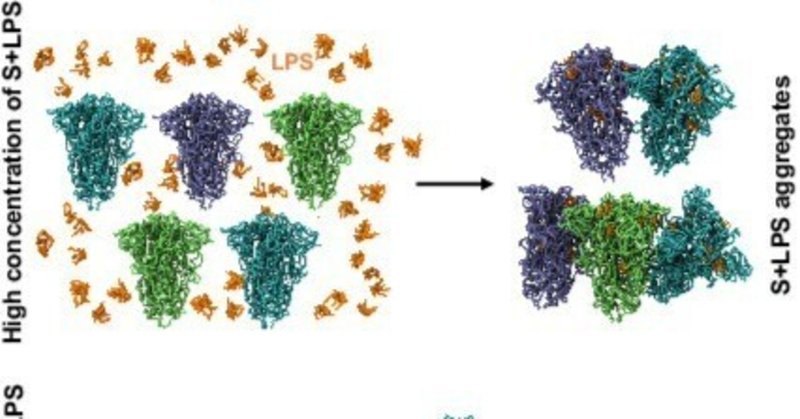
Sタンパク質-LPSは安定な高次オリゴマーを形成する
次に、LPS存在下でのSタンパク質凝集体の安定性を調べるために、TLR4に対する主要な刺激活性を包含するLPSのコア脂質成分であるリピドA存在下でのSタンパク質凝集体のCG MDシミュレーションを行った。現在までに、高次のSタンパク質オリゴマーの低温電子顕微鏡構造が2つある。すなわち、SARS-CoV-2ワクチン候補からの三量体のSタンパク質二量体と三量体のSタンパク質三量体である[11]。LPS 結合部位のそれぞれには、我々の過去の構造研究 [2, 5] に従ってリピド A をロードし、S タンパク質-リピド A 複合体系を 10 μs の間 3 連でシミュレーションした(詳細は Materials and Methods に)。シミュレーションの最初と最後の代表的なスナップショットを、Sタンパク質二量体の三量体とSタンパク質三量体の三量体について、それぞれ図4A,Bに示す。全体として、高次のSタンパク質-脂質A複合体は、両方のシミュレーションセットにおいて安定に結合していた。すべてのケースにおいて、Sタンパク質3量体ユニット間の平均距離は最初の2μs後にプラトーに達し、実際には初期構造と比較して平均で1 nm程度徐々に減少した(図4C)。

写真やイラストなどを保持する外部ファイル。
オブジェクト名は FEB2-9999-0-g005.jpg
図4
脂質Aに結合したSタンパク質凝集体のCG MDシミュレーション (A) 2つのSタンパク質三量体(シアン、紫)と18の脂質A(オレンジ)のCG MDシミュレーションを行った(左図)。Sタンパク質の三量体の出発構造はPDB: 7JJJから取得した。10μsのシミュレーションによる初期と最終のスナップショットを右図に示す。Sタンパク質三量体の各単量体ユニットは、以前に記述したように、NTD、RBD、S2ポケットで3つの脂質A分子と結合している[2, 5]。(B) 3つのSタンパク質三量体(シアン、グリーン、パープル)と27の脂質A分子について同様のシミュレーションを行った(左図)。Sタンパク質の三量体の出発構造は、EMDB: 22355から取得した。10μsのシミュレーションによる初期スナップショットと最終スナップショットを右図に示す。(C) 三量体の二量体(黒)と三量体(赤)のシミュレーションにおける、Sタンパク質三量体の質量中心間の距離。独立した3つのシミュレーションの平均を太線で、標準偏差を斜線で示す。(D) Sタンパク質二量体の三量体(黒)と三量体の三量体(赤)のシミュレーションから得られた脂質A分子のRMSD。RMSD値は、すべての脂質A分子と実施した3つのシミュレーションの平均値である。平均値は太線で、斜線は標準偏差を表している。
次に、すべての脂質A分子がSタンパク質ポケットに結合したままであることを確認することにした。これは、それぞれのSタンパク質複合体の初期構造に最小二乗法を適用した後、各脂質A分子の二乗平均平方根偏差(RMSD)を測定することによって評価された。図4Dに示すように、結合した脂質A分子のRMSDの大きさは、Sタンパク質2量体および3量体のシミュレーション全体を通して、約1.5 nmで安定しており、脂質が結合ポケットから外れることはなかったと思われる。したがって、このシミュレーションは、Sタンパク質-脂質A複合体が原理的に安定な高次のオリゴマーや凝集体を形成できることを示し、上記の実験と一致するものであった。CG法の限界は、タンパク質に適用される弾性ネットワークモデルが、タンパク質の二次構造の大規模な変化を防ぐことである。したがって、Sタンパク質凝集体の形成を促進する可能性のある、脂質Aの結合によって誘発される潜在的な構造変化を観察することはできないだろう。この研究では、システムのサイズが大きいため、原子レベルでのシミュレーションは考慮されなかった。しかしながら、このシミュレーションは、LPS存在下でのSタンパク質凝集の分子レベルでの原理を証明するものである。
次へ
考察
我々は、LPSがSARS-CoV-2のSタンパク質の凝集を誘導し、アミロイド構造を形成することを証明した。LPSの存在下では、Sタンパク質は、Sタンパク質単独で形成される凝集体よりも有意に大きな凝集体を形成した。LPSを隔離するTCP-25はこの効果を逆転させ、Sタンパク質の凝集にLPSが関与していることを確認した。Sタンパク質の凝集しやすい領域は、S1サブユニットのLPS結合部位と重なると予測された。また、高次のSタンパク質オリゴマーと脂質Aを結合させたシミュレーションでは、安定な複合体を形成しており、Sタンパク質がLPS分子と凝集体を形成することをさらに裏付けている。
最近、好中球エラスターゼによるSタンパク質のタンパク質分解で生成されるいくつかのペプチドが凝集体を形成し、そのうちの3つ(ペプチド192-211, 601-620, 1166-1185)はアミロイド線維の基準を満たすことが示された[21]。したがって、これらのペプチドの1つに隣接するCTD2ドメインのループ621-624が凝集しやすいと予測したのは興味深いことである。しかし、折り畳まれた全長のSタンパク質はそれ自体ではアミロイド線維を形成せず、好中球エラスターゼのようなプロテアーゼとのインキュベーションによってアミロイド形成性セグメントが露出することが必要であることを強調する価値がある [21].したがって、本研究は、LPS存在下での全長Sタンパク質の新しい凝集挙動を実証するものである。さらに、ヘパリン結合部位はアミロイド蛋白質に共通する構造的特徴であり、ヘパリンとの相互作用が凝集の引き金になることが報告されている[22, 23]。Sタンパク質には受容体結合領域があり、ヘパリンやヘパリン結合タンパク質と結合する[22]。興味深いことに、タンパク質のヘパリン結合領域はLPSと親和性を持つことが示されており、この観察は、これらのヘパリン結合領域とLPSによるアミロイド形成との間にさらなる関連を与えている[12, 13]。
分子レベルでは、S蛋白質の凝集が低温電子顕微鏡で可視化され、COVID-19 S蛋白質ベースのワクチン候補の構造は、三量体の二量体と三量体の三量体の形成を示した [11]。あるSタンパク質単量体のCTD2付近の短いループが、隣接するSタンパク質単量体のNTDに挿入され、2つのSタンパク質三量体の間に2点の接触点が形成される。同様の挿入は三量体同士の構造でも観察された。したがって、この2つの接触点が高次のオリゴマーや凝集体の核形成の基礎となる可能性がある。これらの2つの接触点とは別に、我々の予測では、S1サブユニット上のLPS結合ポケットを取り囲むいくつかの領域も凝集しやすいと思われる。これらの領域は、LPS結合時に隣接するSタンパク質三量体間のさらなる接触点として働き、LPS存在下でさらに大きな凝集体の形成を促進する可能性がある。NTD上のLPS結合部位にヘム代謝物などの疎水性低分子が結合すると、アロステリックな構造変化を引き起こすことがある[24]。したがって、LPSの結合は、アロステリックなコミュニケーションを通じて凝集しやすい配列を明らかにし、その結果、より大きなS-protein-LPS凝集体を形成する可能性があると思われる。我々のシミュレーションでは、Sタンパク質二量体、三量体の複数の部位に脂質Aが安定に結合することが明らかになった。このことは、Sタンパク質-LPS複合体が実際に安定な凝集体を形成することを示唆している。とはいえ、我々の電子顕微鏡実験や蛍光顕微鏡実験で観察されたように、LPSがSタンパク質単独と比較してより大きなSタンパク質凝集体の形成を促す正確な分子メカニズムについては、さらなる研究が必要であろう。
我々の以前の研究では、SARS-CoV-2のSタンパク質は、TLR4経路においてLPSの追加の運び屋として働き、その結果、過剰な刺激をもたらし、過剰炎症状態に導くことが示された[2, 5]。このことは、ナノモルレベルのSタンパク質がLPSミセルを分解し、TLRを介したLPS反応を著しく高める、超低濃度のLPS(1 nm以下)において重要な生理学的意味を持つ [2, 25]。今回使用した高濃度(それぞれ50μmと1μm)のLPSとSタンパク質におけるアミロイド凝集体の形成の可能性に対するSタンパク質-LPS凝集の影響と、in vivoでの関連性は明らかにさらなる調査が必要である。SARS-CoV-2のSタンパク質は、RBDとfurin切断部位を介してヘパリンやヘパラン硫酸と相互作用することが示されている[26, 27, 28]。このような相互作用は、脳におけるアミロイド蛋白の凝集を促進することが提案されている[23]。さらに、最近の計算機的研究により、Sタンパク質はAβ、α-シヌクレイン、タウ、プリオン、TAR DNA binding protein-43などの凝集しやすいアミロイドタンパク質とも結合することが示された[29]。したがって,高濃度のLPSの存在下で増強されるSタンパク質の凝集が,COVID-19患者の神経変性につながるアミロイドタンパク質の凝集を開始させる可能性がある.興味深いことに、表面糖タンパク質がヘパリンとも結合する単純ヘルペスウイルス1型は、in vitroおよびin vivoでAβ42アミロイド凝集を触媒することが示されている[30]。従って、ある条件下でLPSが生体内で同様にS蛋白の凝集を引き起こし、S蛋白-LPSと他のアミロイド形成蛋白との相互作用を介してアミロイド形成を引き起こすかどうかを調べることが必要である。Sタンパク質の凝集とアミロイド形成の関係を理解することは、SARS-CoV-2感染に伴う神経病理の治療的介入に重要な意味を持つと思われる。
次へ
利益相反について
ASは、トロンビン由来の宿主防御ペプチドに基づく治療法を開発する会社であるin2cure ABの創設者である。ペプチドTCP-25とその変異体は、特許で保護されている。他の著者は、競合する利害関係はないことを表明している。
行く。
著者の貢献
JP と AS がプロジェクトを立案した。JPはThTアッセイ、蛍光顕微鏡、電子顕微鏡、Blue Nativeゲルアッセイを考案し、実験を行った。インシリコ解析の設計と実行は、FSとPJBが行った。原稿は、AS と PJB の協力のもと、JP と FS が執筆した。最終的な原稿は、著者全員で議論し、コメントした。
詳細へ
謝辞
本研究は、スウェーデン研究評議会(プロジェクト2017-02341、2020-02016)、Edvard Welanders Stiftelse and Finsenstiftelsen(Hudfonden)、Torsten Söderberg, Crafoord, Kock, Hedlunds, and Österlund Foundations、およびスウェーデン政府の臨床研究基金(ALF)からの助成金により行われました。Ann-Charlotte Strömdahlの優れた技術的支援に感謝する。Lund University Bioimaging Centrum (LBIC)には電子顕微鏡の設備を利用させていただいたことに感謝する。FS と PJB は BII (ASTAR) のコア資金と ASTAR の助成金 FY21_CF_HTPO SEED_ID_BII_C211418001 によって支援されています。シミュレーションは、National Supercomputing Center, Singapore (https://www.nscc.sg), ASTAR Computational Resource Center (ACRC) のリソース、および理化学研究所のHPCIシステム研究プロジェクト(プロジェクトID:hp200303)によるスーパーコンピュータ「富嶽」で実施しました。
にアクセスしてください。
ノート
Jitka PetrlovaとFirdaus Samsudinはこの論文に等しく貢献した。
編集:Urs Greber
本文へ
データアクセス
本研究の結果を裏付けるデータは、対応する著者(es.ul.dem@avolrtep.aktij)から入手可能であり、合理的な要求があれば利用できる。
詳細へ
参考文献
Felsenstein S, Herbert JA, McNamara PS, Hedrich CM. COVID-19:免疫学と治療法の選択肢。Clin Immunol. 2020;215:108448. [PMC フリーアーティクル] [PubMed] [Google Scholar].
Petruk GPM, Petrlova J, Samsudin F, Strömdahl AC, Cerps S, Uller L, et al. SARS-CoV-2 spike protein binds to bacterial lipopolysaccharide and boosts proinflammatory activity.SARSは細菌リポ多糖と結合して炎症性活性を高める。J Mol Cell Biol.2020;12(12):916-32。[PMC フリーアーティクル] [PubMed] [Google Scholar].
Cinquegrani G, Spigoni V, Iannozzi NT, Parello V, Bonadonna RC, Dei Cas A. SARS-CoV-2 spike protein is not pro-inflammatory in human primary macrophage: Endotoxin contamination and lack of protein glycosylation as possible confounders. Cell Biol Toxicol. 2022;38:667-78. [PMC フリーアーティクル] [PubMed] [Google Scholar].
Ouyang W, Xie T, Fang H, Gao C, Stantchev T, Clouse KA, et al. Variable induction of pro-inflammatory cytokines by commercial SARS CoV-2 spike protein reagents: potential impacts of LPS on in vitro modeling and pathogenic mechanisms in vivo. In vitro modeling. Int J Mol Sci. 2021;22:7540. [PMC フリーアーティクル] [PubMed] [Google Scholar].
Samsudin FRP, Petruk G, Puthia M, Petrlova J, MacAry P, Anand GS, et al. SARS-CoV-2 spike protein as a bacterial lipopolysaccharide delivery system in an overzealous inflammatory cascade.bioRxiv.2021. 10.1101/2021.10.29.466401 [CrossRef] [Google Scholar].
Hariri L, Hardin CC. Covid-19、血管新生、およびARDSエンドタイプ。N Engl J Med. 2020;383:182-3. [PubMed】【Google Scholar】。
Whyte CS, Morrow GB, Mitchell JL, Chowdary P, Mutch NJ. 急性呼吸窮迫症候群(ARDS)における線溶系異常と血栓溶解薬による治療の汎用性 COVID-19. J Thromb Haemost. 2020;18:1548-55. [PMC フリーアーティクル] [PubMed] [Google Scholar].
Awoyemi A, Troseid M, Arnesen H, Solheim S, Seljeflot I. Markers of metabolic endotoxemia as related to metabolic syndrome in an elderly male population at high cardiovascular risk: a cross-sectional study.心血管系リスクの高い高齢男性集団におけるメタボリックシンドロームに関連する代謝性内毒素血症のマーカー。Diabetol Metab Syndr. 2018;10:59. [PMC無料記事】【PubMed】【Google Scholar】。
Vors C, Pineau G, Drai J, Meugnier E, Pesenti S, Laville M, et al. 肥満対痩せ男性におけるカイロミクロンおよびリポポリサッカライド処理と関連した食後内毒素症:脂質の用量効果試験. J Clin Endocrinol Metab. 2015;100:3427-35. [PubMed][Google Scholar].
Toelzer C, Gupta K, Yadav SKN, Borucu U, Davidson AD, Kavanagh Williamson M, et al. SARS-CoV-2 スパイクタンパク質のロックされた構造における自由脂肪酸結合ポケット(Free fatty acid binding pocket in the locked structure of SARS-CoV-2 spike protein). Science. 2020;370:725-30. [PMC フリーアーティクル] [PubMed] [Google Scholar].
また、このような場合にも、「SARS-CoV-2スパイクタンパク質の構造解析」を行うことで、SARS-CoV-2スパイクタンパク質の構造解析を行うことができる。Science. 2020;370:1089-94. [PMC フリーアーティクル] [PubMed] [Google Scholar].
また、このような場合、「臓器移植」を行うことができる。Proc Natl Acad Sci U S A. 2017;114:E4213-22. [PMC フリーアーティクル] [PubMed] [Google Scholar].
Petruk G, Elven M, Hartman E, Davoudi M, Schmidtchen A, Puthia M, et al. グラム陰性菌とそのエンドトキシンのクリアランスにおける完全長アポEの役割. J Lipid Res.2021;62:100086。[PMC フリーアーティクル] [PubMed] [Google Scholar].
Petrlova J, Petruk G, Huber RG, McBurnie EW, van der Plas MJA, Bond PJ, et al. Thrombin由来のC末端フラグメントは細菌とそのプロインフラマー生成物を凝集・捕捉する(The Thrombin-derived C-terminal fragments aggregate and scavenge bacteria and their proinflammatory products. J Biol Chem. 2020;295:3417-30. [PMC フリーアーティクル] [PubMed] [Google Scholar].
Kuriata A, Iglesias V, Pujols J, Kurcinski M, Kmiecik S, Ventura S. Aggrescan3D (A3D) 2.0: prediction and engineering of protein solubility.(タンパク質溶解度の予測とエンジニアリング). Nucleic Acids Res. 2019;47:W300-7. [PMCフリーアーティクル] [PubMed] [Google Scholar].
Cai Y, Zhang J, Xiao T, Peng H, Sterling SM, Walsh RM Jr, et al. Distinct conformational states of SARS-CoV-2 spike protein.(SARS-CoV-2スパイクタンパク質の異なるコンフォーメーション状態). Science. 2020;369:1586-92. [PMC フリーアーティクル] [PubMed] [Google Scholar].
Monticelli L, Kandasamy SK, Periole X, Larson RG, Tieleman DP, Marrink SJ. MARTINI coarse-grained force field: extension to proteins. J Chem Theory Comput. 2008;4:819-34. [PubMed] [Google Scholar].
トロンビン由来 C 末端ペプチドのエンドトキシン中和活性および抗炎症活性の構造的基盤。Nat Commun. 2018;9:2762. [PMCフリーアーティクル] [PubMed] [Google Scholar].
Parrinello M, Rahman A. Polymorphic transitions in single-crystals - a new molecular-dynamics method.(単結晶の多形転移-新しい分子動力学法). J Appl Phys.1981;52:7182-90。[Google Scholar].
Zuzic L, Samsudin F, Shivgan AT, Raghuvamsi PV, Marzinek JK, Boags A, et al. Uncovering cryptic pockets in the SARS-CoV-2 spike glycoprotein. 10.1101/2021.05.442536 [PMC free article] [PubMed] [CrossRef] [Google Scholar].
Nystrom S, Hammarstrom P. SARS-CoV-2 spike proteinのアミロイド形成。J Am Chem Soc.2022;144:8945-50。[PMC フリーアーティクル] [PubMed] [Google Scholar].
SARS-CoV-2の細胞侵入および感染を阻止する新規治療法としてのヘパリン結合ペプチド.Mol Pharmacol. 2020;98:612-9. [PMC フリーアーティクル] [PubMed] [Google Scholar].
Tavassoly O, Safavi F, Tavassoly I. Seeding brain protein aggregation by SARS-CoV-2 as a possible long-term complication of COVID-19 infection(SARS-CoV-2によるCOVID-19感染による長期合併症の可能性)。ACS Chem Nerosci. 2020;11:3704-6. [PubMed] [Google Scholar].
Rosa A, Pye VE, Graham C, Muir L, Seow J, Ng KW, et al. SARS-CoV-2 can recruit a heme metabolite to evade antibody immunity.SARS-CoV-2は、抗体免疫を回避するためにヘム代謝物をリクルートすることができる。Sci Adv. 2021;7:eabg7607. [PMC フリーアーティクル] [PubMed] [Google Scholar].
Puthia MTL, Petruk G, Schmidtchen A. SARS-CoV-2 スパイクタンパク質とエンドトキシンで誘発される肺炎の実験モデル. ACS Pharmacol Transl Sci. 2022;5:141-8. [PMC フリーアーティクル] [PubMed] [Google Scholar].
Clausen TM, Sandoval DR, Spliid CB, Pihl J, Perrett HR, Painter CD, et al. SARS-CoV-2 infection depends on cellular heparan sulfate and ACE2. Cell. 2020;183:1043–57.e15. [PMC free article] [PubMed] [Google Scholar].
Zhang Q, Chen CZ, Swaroop M, Xu M, Wang LH, Lee J, et al. Heparan sulfate assisted SARS-CoV-2 in cell entry and can be targeted by approved drugs in vitro. Cell Discov. 2020;6:80. [PMC フリーアーティクル] [PubMed] [Google Scholar].
Partridge LJ, Urwin L, Nicklin MJH, James DC, Green LR, Monk PN. SARS-CoV-2 スパイクタンパク質の ACE2 に依存しないヒト上皮細胞との相互作用は、未分画ヘパリンで阻害される。Cell. 2021;10:1419. [PMC フリーアーティクル] [PubMed] [Google Scholar].
SARS-CoV-2 スパイクタンパク質とアミロイド生成タンパク質との相互作用:神経変性への潜在的な手がかり。Biochem Biophys Res Commun. 2021;554:94-8. [PMC フリーアーティクル] [PubMed] [Google Scholar].
Ezzat K, Pernemalm M, Palsson S, Roberts TC, Jarver P, Dondalska A, et al. Viral protein corona directs viral pathogenesis and amyloid aggregation.ウイルスタンパク質コロナは、ウイルスの病原性とアミロイド凝集を指示する。Nat Commun. 2019;10:2331. [PMC無料記事】【PubMed】【Google Scholar】。
FOLLOW NCBI
NLMとつながる
この記事が気に入ったらサポートをしてみませんか?
