K-Meansクラスタリング scikit-learnを使わずゼロから実装する(Python)|
K-Meansは個人的にも好きなアルゴリズムで、教師あり学習を必要とせず、混沌とした中からパターンを見つけ出してくれる可能性があります。
実際、実践で使うとそのような都合の良いデータはほとんどなく、むしろ
仮説を否定する場合に使う方が現実的かもしれません。
自分の場合、顧客のセグメント分けに使うことが多いです。
ステップ 1. ランダムに k 個のデータポイントを初期のセントロイドとして選ぶ。
ステップ 2. トレーニングセット内のデータポイントと k 個のセントロイドの距離(ユークリッド距離)を求める。
ステップ 3. 求めた距離に基づいて、データポイントを一番近いセントロイドに割り当てる。
ステップ 4. 各クラスタグループ内のポイントの平均を取ることでセントロイドの位置を更新する。
ステップ 5. ステップ 2 から 4 をセントロイドが変化しなくなるまで繰り返します。
以下、シンプルなトイデータで試した場合です。kの数も2とし、データも2次元です。一行づつ追ってみてください。
import numpy as np
import matplotlib.pyplot as plt
from scipy.spatial.distance import cdist
def kmeans(x,k, no_of_iterations):
#centroidsのindexをk個分ランダムに抽出。
idx = np.random.choice(len(x), k, replace=False)
#Step 1 上記で選んだindexに該当するデータを取り出しcentroidsとする
centroids = x[idx, :]
#Step 2 centroidsと各データ分のユークリッド距離を計算
distances = cdist(x, centroids ,'euclidean')
#Step 3 上で計算した距離からcentroidsに一番近い近いほうデータのindexを入れた配列を作る。
#例:[近い,遠い]この場合は0 [遠い,近い] この場合は1
#結果→[1 1 1 1 0 0 0 0 0 0]を返す
points = np.array([np.argmin(i) for i in distances])
#Step 4 centroids更新処理
for _ in range(no_of_iterations):
centroids = []
for idx in range(k):
#kと同じ(一番距離が近い)データの平均(距離)を取る。
temp_cent = x[points==idx].mean(axis=0)
#k分の平均距離(座標が入る)
centroids.append(temp_cent)
centroids = np.vstack(centroids) # Centroidsを更新
#再度距離を計算
distances = cdist(x, centroids ,'euclidean')
#上で計算した距離で一番近いcentroidsのindexの配列を作る。
points = np.array([np.argmin(i) for i in distances])
#どのラベルがどのデータに紐づいているか表す配列を返す。(データに対応したラベルが入る)
return points
# テストデータ
X = np.array([[0, 0], [1, 1], [1, 0], [0, 1], [2, 2], [2, 1], [1, 2], [3, 3], [3, 2], [2, 3]])
#実行
label = kmeans(X,2, 10)
# 結果を結果をplot
plt.scatter(X[:,0], X[:,1])
plt.show()
#結果を可視化可視化(ラベル付き)
u_labels = np.unique(label)
for i in u_labels:
plt.scatter(X[label == i , 0] , X[label == i , 1] , label = i)
plt.legend()
plt.show()

ちなみにユークリッド距離はPythonで実装するとこうなります。
def euclidean_distance(x1, x2):
return np.sqrt(np.sum((x1 - x2)**2))大量のデータで次数を増やした場合でも、ちゃんと動くか検証してみましょう。
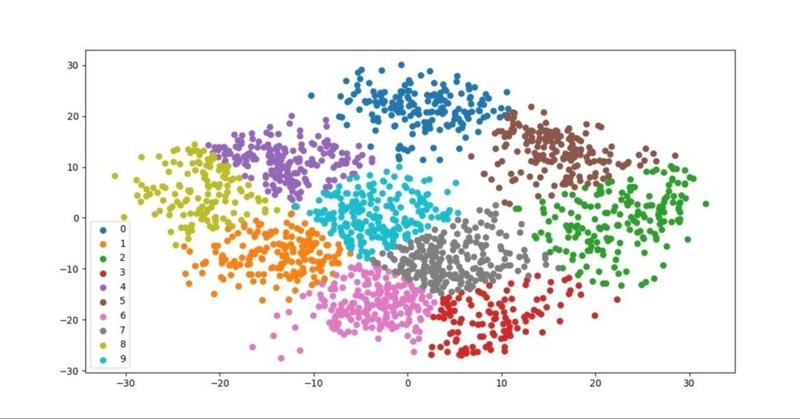
以下、PCA(次元削減)をデータを2次元にした場合です。
from sklearn.datasets import load_digits
from sklearn.decomposition import PCA
import matplotlib.pyplot as plt
#テストデータをロード
data = load_digits().data
#PCAで次元削減
pca = PCA(2)
df = pca.fit_transform(data)
#上記で実装したアルゴリズムを適用
label = kmeans(df,10,1000)
#結果プロット
u_labels = np.unique(label)
for i in u_labels:
plt.scatter(df[label == i , 0] , df[label == i , 1] , label = i)
plt.legend()
plt.show()
機械学習のアルゴリズムのメリット・デメリットが書いてある超オススメ書籍。
この記事が気に入ったらサポートをしてみませんか?
