
主成分分析について
この記事の狙い・目的
機械学習を取り入れたAIシステムの構築は、
①データセット作成(前処理)→ ②モデルの構築 → ③モデルの適用
というプロセスで行っていきます。
その際「データセット作成(前処理)」の段階では、正しくモデル構築できるよう、事前にデータを整備しておくことが求めらます。
このブログでは、その際に問題なることが多い、「次元の呪い」やその対処方法となる「主成分分析(次元削減)」について解説していきます。
主成分分析とは
主成分分析とは、あるデータ群をより少ないデータ群で要約する解析手法のことです。
ここれでその具体的なイメージや、なぜそのような手法が取られるのか、その背景から解説していきます。
次元の呪い
次元の呪いとは、データの次元数が大きくなり過ぎると、そのデータで表現できる組み合わせが飛躍的に多くなってしまい、その結果十分な学習結果が得られなくなることを言います。
結果的に、未知のデータに対する予測精度(汎化性能)を高めづらくなり、また計算負荷が非常に高くなることが問題点としてあげられます。
次元削減
以上の問題点から、データ数が非常に多いデータセットでは、時限削減という手法が有効となる場合があります。
次元削減とは、(ただ単にデータ数を減らすのではなく)可能な限り元のデータの情報を保持したまま、低次元のデータに落とし込むことを言います。
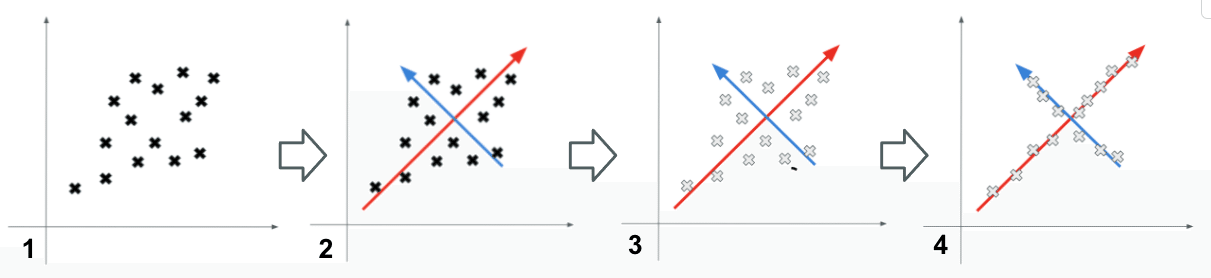
もう少し具体的に解説していきましょう。
まず、特徴量空間上にその、そのデータ群を最もよく表現するベクトルを表します。(※1→2の図)
次に、各データをベクトル(赤・青)上に射影していきます。(※3→4の図)
赤ベクトル上に射影されたデータ群を「第一主成分」、青ベクトル上に射影されたデータ群を「第二主成分」と呼び、これにとり元データを、2次元のデータで表すことができるようになります。
まずはそのままのデータで学習、評価
まずは次元削減を行わず、そのままのデータで学習・評価を行なった結果を見ていきましょう。
プログラム等の実行環境
Python3
MacBook pro(端末)
PyCharm(IDE)
Jupyter Notebook(Chrome)
Google スライド(Chrome)
import numpy as np
import pandas as pd
from sklearn.metrics import confusion_matrix
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
from sklearn.linear_model import LogisticRegressionCV
# データ取得
breast_cancer = pd.read_csv("./breast_cancer_diagnostic.csv", sep=',')
# 混同行列
def plot_confusion_matrix(predict, y_test):
pred = np.where(predict > 0.5, 1, 0)
cm = confusion_matrix(y_test, pred)
matrix = pd.DataFrame(cm)
matrix.columns = [['予測_負例(0)', '予測_正例(1)']]
matrix.index = [['実際_負例(0)', '実際_正例(1)']]
return matrix
# 目的変数の抽出、データ分割
X = breast_cancer.drop('診断', axis=1)
y = breast_cancer['診断']
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.4, random_state=0)
# 標準化
scaler = StandardScaler()
X_train_scaled = scaler.fit_transform(X_train)
X_test_scaled = scaler.transform(X_test)
# ロジスティック回帰
logistic = LogisticRegressionCV(max_iter=1000, random_state=42)
logistic.fit(X_train_scaled, y_train)
# 検証、評価
print('訓練スコア: {:.3f}'.format(logistic.score(X_train_scaled, y_train)))
print('テストスコア: {:.3f}'.format(logistic.score(X_test_scaled, y_test)))
# 混同行列
y_pred = logistic.predict(X_test_scaled)
matrix = plot_confusion_matrix(y_pred, y_test)
matrix
# 学習曲線
from sklearn.model_selection import learning_curve
import japanize_matplotlib
%matplotlib inline
def plot_learning_curve(model, X_data, y_data):
# データ準備
train_sizes, train_scores, val_scores = learning_curve(model, X=X_data, y=y_data, train_sizes = np.linspace(0.1, 1.0, 10), cv=10, n_jobs=1)
train_scores_mean = np.mean(train_scores, axis=1)
train_scores_std = np.std(train_scores, axis=1)
val_scores_mean = np.mean(val_scores, axis=1)
val_scores_std = np.std(val_scores, axis=1)
plt.figure(figsize=[10,5])
plt.title("学習曲線")
plt.xlabel("訓練サイズ")
plt.ylabel("スコア")
# 訓練スコア と 検証スコア をプロット
plt.plot(train_sizes, train_scores_mean, 'o-', color="r", label="訓練スコア")
plt.plot(train_sizes, val_scores_mean, 'o-', color="g", label="検証スコア")
# 標準偏差の範囲を色付け
plt.fill_between(train_sizes, train_scores_mean - train_scores_std, train_scores_mean + train_scores_std, color="r", alpha=0.2)
plt.fill_between(train_sizes, val_scores_mean - val_scores_std, val_scores_mean + val_scores_std, color="g", alpha=0.2)
# Y軸の範囲
plt.ylim(0.8, 1.0)
# 凡例の表示位置
plt.grid()
plt.legend(loc="best")
plt.show()
plot_learning_curve(logistic, X_train_scaled, y_train)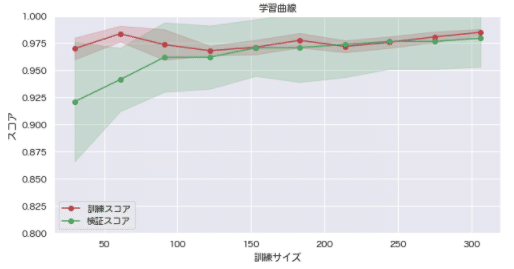
訓練スコアと検証スコアが共に高すぎる気がします。
また、両スコアが非常に近似していることから、訓練データ・検証データの差があまりない可能性が考えられます。
その為、この結果から汎化性能の高さを裏付けることは難しそうです。偶然予測結果が正解と合ってしまっている可能性が懸念されます。
主成分分析を行なった結果と比較して、再度考察してみましょう。
相関分析
本題の主成分分析を解説する前に、相関分析について解説しておきます。
相関分析とは、各データ群がどれほど関係しているか、つまりデータの関連度合い(強さ)を分析する手法です。
その度合いを表す指標に「相関係数」というものがあり、それらを可視化した相関行列というものがあります。実際に見ていきましょう。
# ヒートマップ
import seaborn as sns
plt.figure(figsize=(20, 20))
sns.heatmap(pd.DataFrame(X_train_scaled).corr(), annot=True);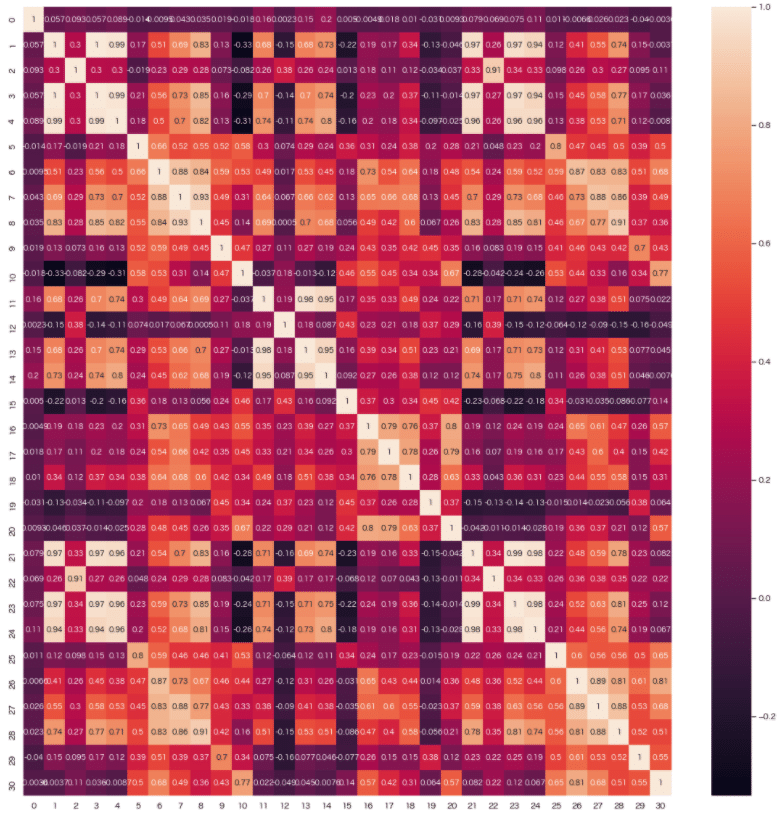
よく見ると相関係数が0.8以上のもが点在しており、変数間の相関が高い項目が多いことがわかります。
また変数間の関係性を散布図で表す散布図行列(ペアプロット)というものがあります。
※対角線は各変数をヒストグラムで表しています。
sns.pairplot(pd.DataFrame(X_train_scaled).corr(), height=2);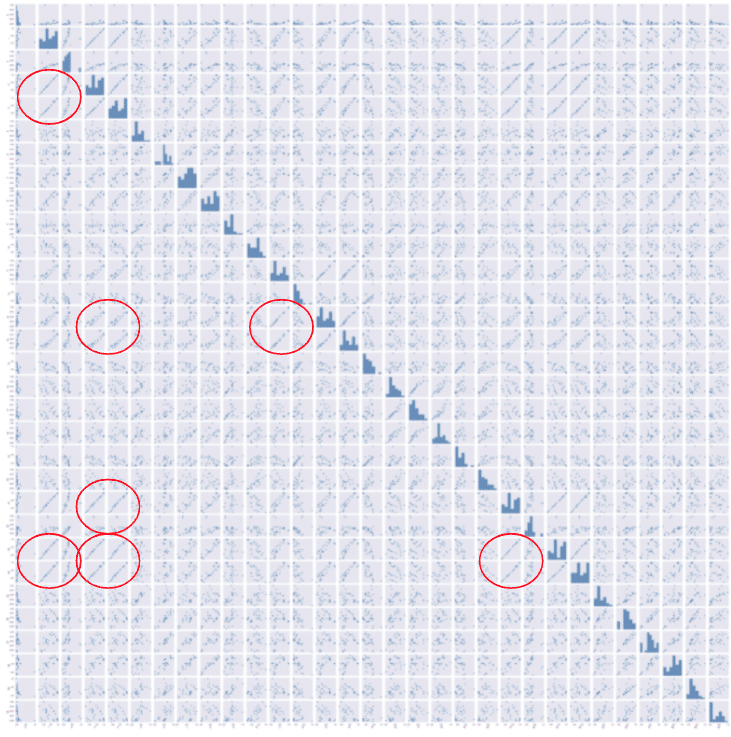
散布図行列により、相関行列の相関係数が信じられるかの確認を行います。よく見ると線形関係の変数がちらほらと見られることがわかります。これらの変数間の相関が強そうです。
問題点
今回のモデル構築では、目的変数(1=正例(乳がん悪性)、2=負例(乳がん良性))を予測する為に、一般化線形モデルの一つ「ロジスティック回帰」を用います。
ロジスティック回帰とは、目的変数が起こる「確率を予測」し、結果的にその予測確率を用いて分類を行うことができるアルゴリズムです。
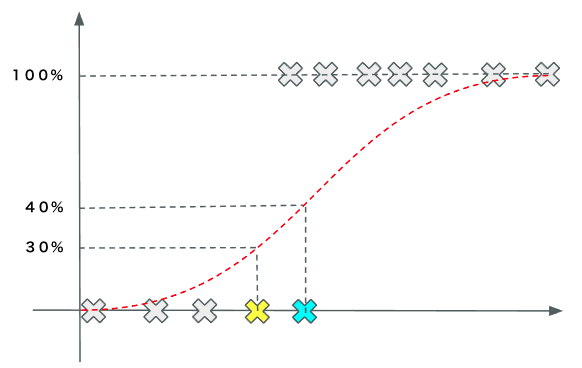
ただし、一般線形モデルを用いる際、説明変数間に強い相関がある場合、「多重共線性」による「オーバーフィッティング(過学習)」の危惧する必要があると思われる。
多重共線性とは、2つ以上の変数間に高い線形関係がある状態のことを言います。
オーバーフィッティング(過学習)とは、学習データに過剰に適合してしまい、未知のデータ(テストデータ等)に対して的確な予測ができなくなってしまった状態のことです。
後々、交差検証を用いて改善を図っていきたいと思います。
※正則化項については、今回は割愛させていただきます。
多重共線性の確認
主成分分析を行う前に、(念の為)多重共線性のチェックを行なっていきましょう。今回はVIFという指標を用いて確認していきたいと思います。
VIF(Variance Inflation Factor)
VIFとは、変数間の多重共線性を検出するための指標の1つです。算出結果に対し10を基準とすることが多いようです。
from statsmodels.stats.outliers_influence import variance_inflation_factor
vif = pd.DataFrame()
vif["VIF Factor"] = [variance_inflation_factor(X.values, i) for i in range(X.shape[1])]
vif["特徴量"] = X.columns
vif.sort_values(by='VIF Factor', ascending=False)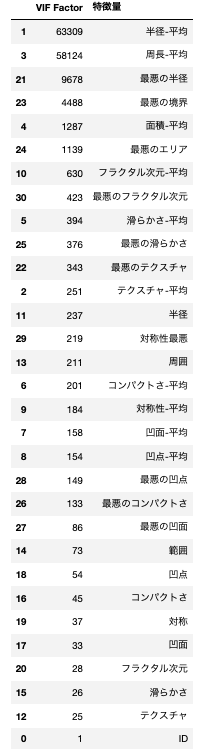
VIF統計量が10以上の変数がほとんどという結果になりました。多重共線性ありと判断できそうです。
主成分分析
いよいよ本題の主成分分析について解説していきます。
主成分分析とは、あるデータ群をより少ないデータ群で要約する解析手法のことです。
そのため主成分分析の狙いは、データ群を要約する際になるべく情報の損失せずに、データを最小限に圧縮するということになります。
その結果、データ構造がわかりやすくなり、同時に計算も負荷も下げられるというメリットがあります。
それでは実際に実装を見ていきましょう。
# 主成分分析
from sklearn.decomposition import PCA
# 寄与率
pca = PCA(n_components=30)
pca.fit(X_train_scaled)
plt.figure(figsize=[10,5])
plt.grid()
plt.bar([n for n in range(1, len(pca.explained_variance_ratio_)+1)], pca.explained_variance_ratio_);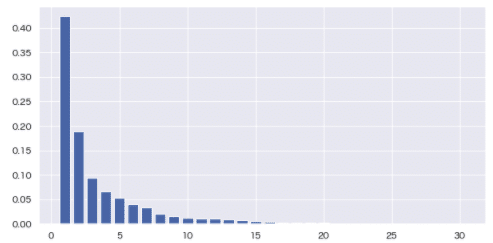
# 累積寄与率
contribution_ratio = pca.explained_variance_ratio_
accumulation_ratio = np.cumsum(contribution_ratio)
cc_ratio = np.hstack([0, accumulation_ratio])
plt.figure(figsize=[8,5])
plt.plot(accumulation_ratio, "-o")
plt.xlabel("主成分")
plt.ylabel("累積寄与率")
plt.grid()
plt.show()
contribution_ratios = pd.DataFrame(pca.explained_variance_ratio_)
contribution_ratios.round(decimals=2).astype('str').head()
print(f"累積寄与率: {contribution_ratios[contribution_ratios.index<5].sum().round(decimals=2).astype('str').values}")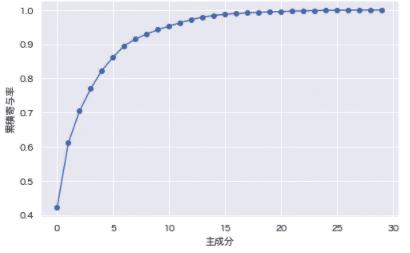

# PCA
# 次元数2まで圧縮
pca = PCA(n_components=2, random_state=42)
X_train_pca = pca.fit_transform(X_train_scaled)
print(f'説明分散比: {pca.explained_variance_ratio_}')
# 散布図
temp2 = pd.DataFrame(X_train_pca)
temp2['目的変数'] = y_train.values
b = temp2[temp2['目的変数'] == 0]
m = temp2[temp2['目的変数'] == 1]
plt.figure(figsize=[10,5])
sns.scatterplot(x=b[0], y=b[1], marker='o', label='良性') # 良性
sns.scatterplot(x=m[0], y=m[1], marker='x', label='悪性') # 悪性
plt.xlabel('PC 1') # 第1主成分:x軸
plt.ylabel('PC 2') # 第2主成分:y軸
plt.legend()
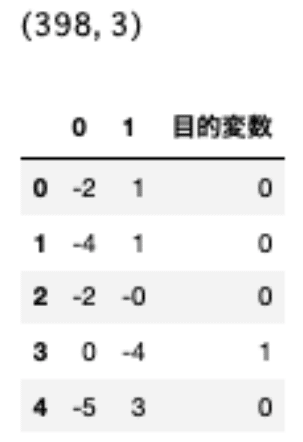
temp2.shape
temp2.head()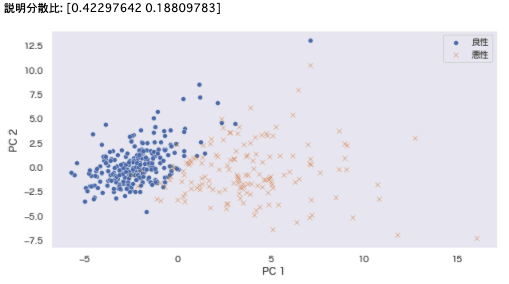
第1・第2主成分は、説明分散比が高く(合計:61%)、よく分離できている。元データに対する説明力が高いことがわかります。
また、累積寄与率が80%までの第1〜第5成分まを抽出しておきます。
pca = PCA(n_components=5, random_state=42)
X_train_pca = pca.fit_transform(X_train_scaled)
print(f'説明分散比: {pca.explained_variance_ratio_}')
# 散布図
temp5 = pd.DataFrame(X_train_pca)
temp5['目的変数'] = y_train.values
b = temp5[temp5['目的変数'] == 0]
m = temp5[temp5['目的変数'] == 1]
temp5.shape
temp5.head()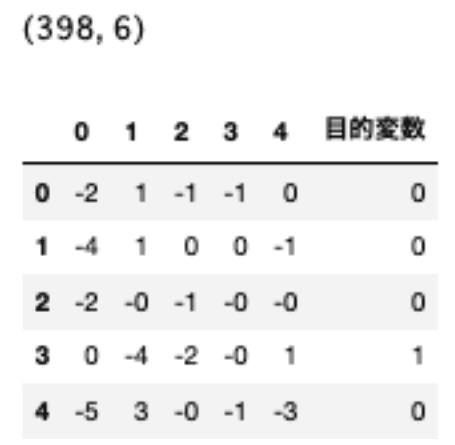
今回は、累積寄与率が60%の第2主成分までと、累積寄与率が80%までの第5成分までをそれぞれ抽出・学習し、結果を比較していきます。
次元削減後のデータで学習
def learning(data):
# 目的変数の抽出、データ分割
X = data.drop('目的変数', axis=1)
y = data['目的変数']
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.3, random_state=0)
# 標準化
scaler = StandardScaler()
X_train_scaled = scaler.fit_transform(X_train)
X_test_scaled = scaler.transform(X_test)
# ロジスティック回帰
model = LogisticRegressionCV(max_iter=100, random_state=42)
model.fit(X_train_scaled, y_train)
# 検証、評価
print('Train score: {:.3f}'.format(model.score(X_train_scaled, y_train)))
print('Test score: {:.3f}'.format(model.score(X_test_scaled, y_test)))
# 混同行列
y_pred = model.predict(X_test_scaled)
matrix = plot_confusion_matrix(y_pred, y_test)
matrix
return model, X_train_scaled, y_train, matrix# 次元数2のデータで学習
model, X_train_scaled, y_train, matrix = learning(dimensio_reduce2)
matrix
plot_learning_curve(model, X_train_scaled, y_train)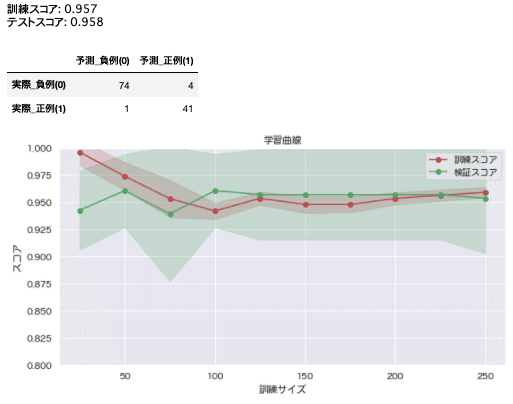
第2主成分まででは、まだ情報不足のようです。
# 次元数5のデータで学習
model, X_train_scaled, y_train, matrix = learning(dimensio_reduce5)
matrix
plot_learning_curve(model, X_train_scaled, y_train)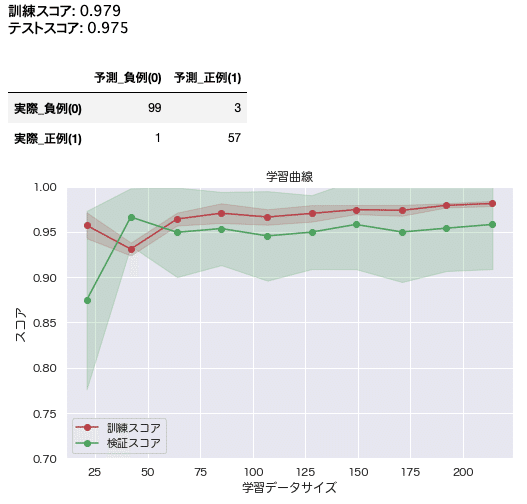
主成分分析を用いた次元削減によりサンプルに含まれたノイズが取り除かれ、訓練データと学習データのスコアの重なりが改善されたように見られます。
ただし依然としてスコアが高すぎる気がする為、(念の為)ROC曲線とAUCも見ていきましょう。
ROC曲線とは、縦軸は真陽性率、横軸は真陰性率を表した図のことです。
AUCは、モデルの判別性能を表す指標です。ROC曲線の曲線下の面積部分のことを指してします。
AUCが0から1に近くにつれて判別性能が高いことを意味しています。
# ROC曲線
from sklearn import metrics
def plot_roc_curve(y_pred, y_test):
fpr, tpr, thresholds = metrics.roc_curve(y_pred, y_test)
plt.figure(figsize=[8, 3])
plt.plot(fpr, tpr, label=f'roc_curve')
plt.xlabel('FPR')
plt.ylabel('TPR')
plt.legend()
plt.grid()
plt.show()
auc = metrics.auc(fpr, tpr)
print(f'AUC: {auc.round(decimals=3)}')
plot_roc_curve(y_pred, y_test)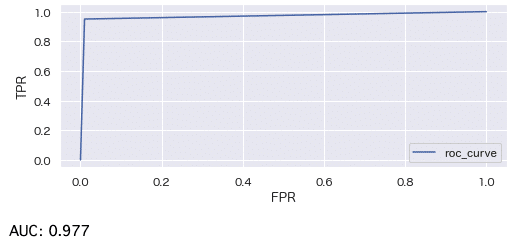
AUCが高く、扱っているデータに対しては判別性能が高いと言えそうです。ただし、更なる未知のデータに対しての判別性能が低いかもしれない。
汎化性能を確認する為、更に交差検証(クロスバリデーション)を行なってみたいと思います。交差検証とは汎化性能を評価する統計的な手法の一つで、いくつかの方法がありますが、今回は詳細を割愛させていただきます。
それでは実装と結果を見ていきましょう。
from sklearn.model_selection import StratifiedKFold
from tqdm import tqdm # プログレスバー
X = dimensio_reduce5.drop('目的変数', axis=1)
y = dimensio_reduce5['目的変数']
sk = StratifiedKFold(n_splits=5, random_state=42, shuffle=True)
aucs = []
for train_index, test_index in tqdm(sk.split(X, y)):
X_train_fld, X_test_fld = X.loc[train_index], X.loc[test_index]
y_train_fld, y_test_fld = y.loc[train_index], y.loc[test_index]
# ロジスティック回帰
model = LogisticRegressionCV(cv=5,
max_iter=1000,
random_state=42,
penalty='l2', # L2ノルム(正則化項)
n_jobs=1)
model.fit(X_train_fld, y_train_fld)
pred = model.predict(X_test_fld)
matrix = plot_confusion_matrix(pred, y_test_fld)
matrix
# 検証、評価
fpr, tpr, thresholds = metrics.roc_curve(y_test_fld, pred)
auc = metrics.auc(fpr, tpr)
auc
aucs.append(auc)
# 検証、評価
np.mean(aucs, axis=0).round(decimals=3)
# AUC: 0.97交差検証の結果でも高い評価点を得られることがわかりました。
"今回扱ったデータセットに対しては"、判別性能は高いと言うことができそうです。
まとめ
各変数の強いデータセットに対して、主成分分析(次元削減)という手法が有効な手法の一つであることを確認できました。
ただし、今回扱ったデータセットはそもそものサンプル数が少ない為、更なる未知のデータに対しての汎化性能を推し量ることは難しそうだ(汎化性能が高いとは言い切れない)と思いました。
その為、サンプル数を増やす、または統計的に母集団を推定してサンプリングする、などの方法を取る必要がありそうです。
また、主成分分析(PCA)は予測に関係のないノイズを多く抽出してしまう傾向がある為、事前に特徴量の精査を行うことも有効と思われます。
今後「理化学研究所:汎化能力を最大化する特徴抽出」で研究されているような、汎化性能の最大化、およびその評価手法なども、ゆくゆく取り入れていきたいと思います。
今回のテーマは「主成分分析」の為、解説はここまでとさせていただきますが、サンプリングなどの改善手法やその考察については、別記事で別途解説したいと思います。
参考文献
統計Web 一般線形モデル
統計Web 多重共線性をチェックする
統計Web VIF / Variance Inflation Factor
統計Web AUC(Area under the curve)
東京大学 Taiji Suzuki's home page > データ解析 第四回「線形回帰分析の拡張:一般化線形モデル」
京都大学大学院工学研究科化学工学専攻 > 主成分分析
流通科学大学:サンプルサイズが小さい場合の統計的検定の比較
理化学研究所:汎化能力を最大化する特徴抽出
解析結果
github/purchase_forecast_feature.ipynb
データセット:Breast Cancer Wisconsin (Diagnostic) Data Set - kaggle
参考資料
この記事が気に入ったらサポートをしてみませんか?
