【R Shiny】easyPubMedとShinyでPubMed論文情報収集アプリを作る
論文を探す時にありがたいPubMed。
ただし、検索結果からabstractが見にくくてどんな論文か分かりにくい、興味のある論文の一覧化がしにくかったりと不便な点もあります。
特定のトピックをルーチンで検索して結果をまとめる…なんてのも大変。
そこでRのパッケージeasyPubMedのPubMed情報収集をShinyでアプリケーション化してみました。
下の記事を参考にさせていただきました。
コードは以下の通りです。
# cache clear
rm(list = ls())
gc()
gc()
library("shiny")
library("shinymanager")
library("shinythemes")
library("shinybusy")
library("easyPubMed")
library("tidyverse")
library("DT")
library("lubridate")
# Define UI for application that draws a histogram
ui <- fluidPage(
# Application title
titlePanel("Pubmed data collection"),
# Sidebar with a slider input for number of bins
sidebarLayout(
sidebarPanel(
width = 2,
dateRangeInput("date", "Date range:",
start = Sys.Date() - 60,
end = Sys.Date(),
max = Sys.Date(),
format = "yy/mm/dd",
separator = " - "
),
textInput("word_to_search",
label = "word to search",
value = "Rstudio"
),
actionButton("get_data", "Get data", class = "btn-primary"),
br(), br(),
"Download",
br(),
downloadButton("download_data", ".csv")
),
# Show results
mainPanel(
width = 10,
DT::dataTableOutput("data_table")
)
)
)
# Define server logic required to draw a histogram
server <- function(input, output) {
pubmed_df <- eventReactive(
input$get_data,
{
show_modal_spinner()
# create search word for pubmed
query <- input$word_to_search
date_to <- input$date[2] %>% as.Date(origin = "1970-01-01")
date_from <- input$date[1] %>% as.Date(origin = "1970-01-01")
to <- gsub("-", "/", date_to)
from <- gsub("-", "/", date_from)
Querytime <- str_c(' AND("', from, '"[PDat] : "', to, '"[PDat])')
Queryt <- query %>% str_c(Querytime)
pubmedidlist <- get_pubmed_ids(Queryt)
# get_data
papers <- fetch_pubmed_data(pubmedidlist, encoding = "UTF-8")
# create for-loop list
articlelist <- papers %>% articles_to_list()
narticles <- length(articlelist)
pmid <- numeric(narticles)
year <- numeric(narticles)
day <- numeric(narticles)
month <- numeric(narticles)
lastname <- character(narticles)
firstname <- character(narticles)
title <- character(narticles)
abstract <- character(narticles)
journal <- character(narticles)
# create dataframe from list
for (i in 1:narticles) {
df <- articlelist[[i]] %>% article_to_df(max_chars = 10000)
pmid[i] <- df[1, ]$pmid %>% as.numeric()
year[i] <- df[1, ]$year %>% as.numeric()
month[i] <- df[1, ]$month %>% as.numeric()
day[i] <- df[1, ]$day %>% as.numeric()
lastname[i] <- df[1, ]$lastname
firstname[i] <- df[1, ]$firstname
title[i] <- df[1, ]$title
abstract[i] <- df[1, ]$abstract
journal[i] <- df[1, ]$journal
}
# bind
result <- data.frame(
pmid = pmid,
date = paste(year, month, day, sep = "-") %>% ymd(),
name = paste(lastname, firstname, sep = " "),
title = title,
journal = journal,
url = paste0("https://www.ncbi.nlm.nih.gov/pubmed/", pmid),
abstract = abstract
) %>%
arrange(desc(date))
return(result)
}
)
# Pubmed_data cleansing
table_data <- reactive({
req(pubmed_df())
x2 <- pubmed_df() %>%
mutate(url = paste0("<a href='", pubmed_df()$url, "'>", pubmed_df()$url, "</a>"))
remove_modal_spinner()
x2
})
# DT output
output$data_table <- DT::renderDataTable(
table_data(),
escape = FALSE,
options = list(
lengthMenu = c(5, 30, 100),
autoWidth = TRUE,
pageLength = 5,
scrollY = "800px",
scrollX = TRUE,
scrollCollapse = TRUE
)
)
# download button server logic
output$download_data <- downloadHandler(
filename = function() {
paste(ymd(Sys.Date()), "_", input$word_to_search, "_PubMed.csv", sep = "")
},
content = function(file) {
write.csv(pubmed_df(), file, row.names = FALSE)
}
)
}
# Run the application
shinyApp(ui = ui, server = server)
# shinyapps
# tmp.enc <- options()$encoding
# options(encoding = "UTF-8")
# library(rsconnect)
# options(encoding = tmp.enc)
# rsconnect::deployApp()
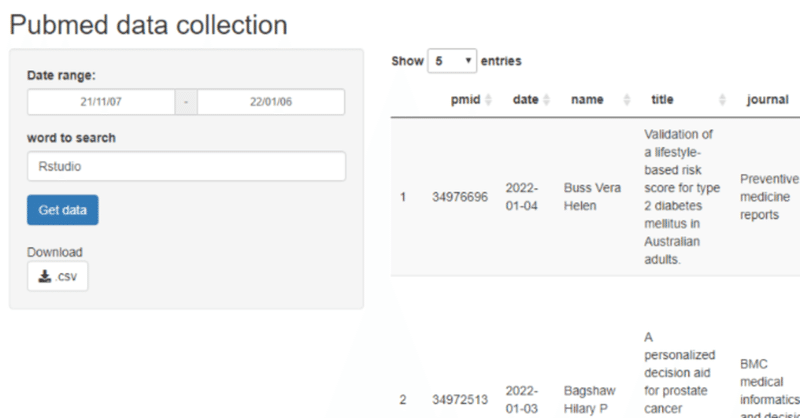
sidebar panelのDate rangeに検索期間を入力、word to searchに検索ワードを入れてGet dataを押すと検索。
main panelにDTでtableを表示します。
得られたデータはDownloadからcsvファイルでダウンロードできるようにしました。
ダウンロードはwindowsの日本環境向けにwrite.csvでエンコードをCP932としていますが、UTF-8でいいならばreadr::write_csvの方が適切かと思います。
関連論文の一覧をcsvで取得したい場合には便利かもしれません。
