R言語:厚生労働省のページのコロナ陽性者数オープンデータを使ってtidyverseでグラフを描く
◆はじめに
厚生労働省のページのコロナ陽性者数オープンデータの方式が変更されました。県ごとのデータが追加されているようです。
以前の記事のコロナ陽性者数prophet予測では最新のデータに対応できなくなってしまいました。
データの内容はともかく、データがあれば触って整形し、処理し、可視化してみたくなるのがR使いの定めというもの。せっかくなのでdplyrとggplotの練習を兼ねて、良い感じのグラフを作成してみます。
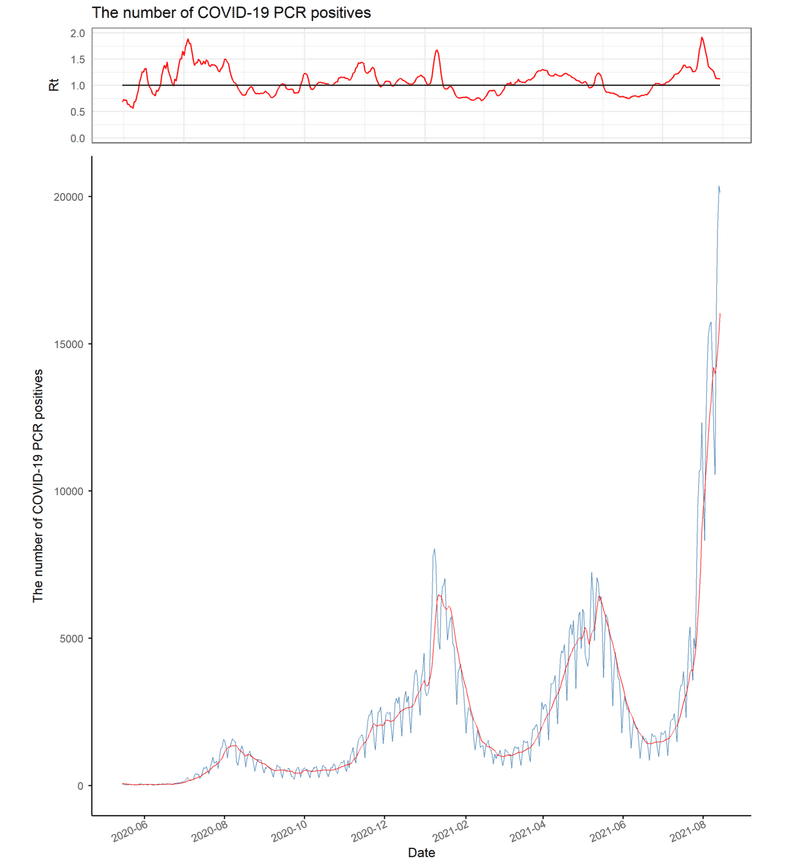
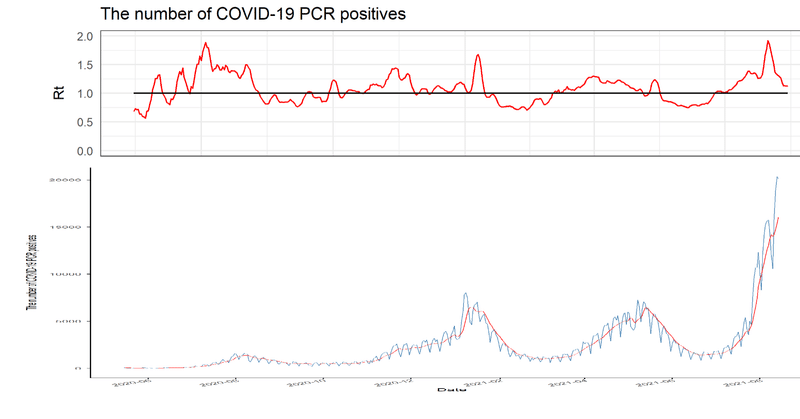
こんな感じのPCR陽性者数の推移+7日間移動平均線+簡易実効再生産数(Rt)を表示するグラフと
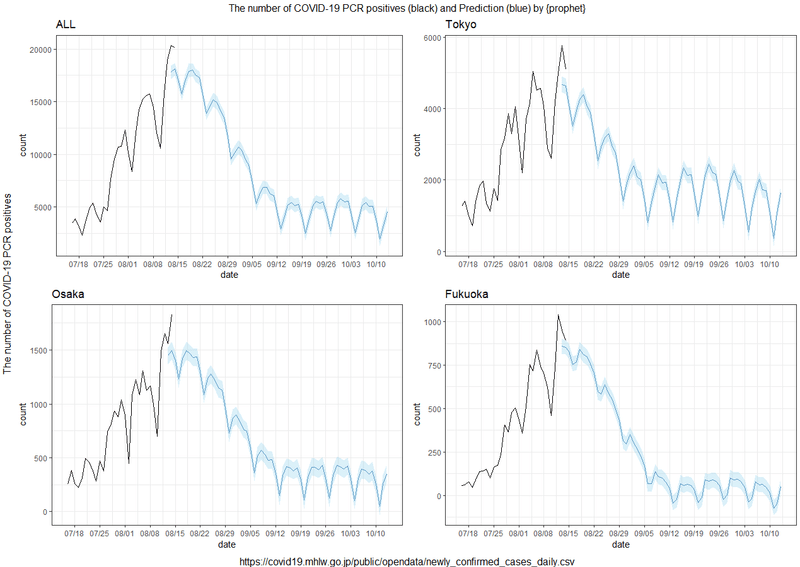
こんな感じの選択された各都道府県の60日予測を並べたグラフを作っていきます。
●ライブラリの読み込み
# No need for the second and subsequent times.
install.packages("tidyverse", dependencies=TRUE)
install.packages("prophet", dependencies=TRUE)
install.packages("lubridate", dependencies=TRUE)
install.packages("zoo", dependencies=TRUE)
install.packages("scales", dependencies=TRUE)
install.packages("gridExtra", dependencies=TRUE)
library("tidyverse")
library("prophet")
library("lubridate")
library("zoo")
library("scales")
library("gridExtra")tidyverse: いつもの。主に使われているのはdplyrとggplot。
prophet: 時系列予測用
lubridate: 時系列処理用
zoo: 移動平均出す用
scales: ggplotの年月日x軸ラベルを良い感じにする用
gridExtra: ggplotのグラフを並べる用
●データの読み込みと整形
url <- "https://covid19.mhlw.go.jp/public/opendata/newly_confirmed_cases_daily.csv"
dat_x <- readr::read_csv(url,
locale = locale(encoding = "utf8"))
dat_l <- dat_x %>%
group_by(Prefecture) %>%
rename(p = 'Newly confirmed cases') %>%
mutate(ma7 = rollmean(p, 7L, align = "right", fill = 0),
Rt = ma7/lag(ma7, n = 5L, default = 0, fill = 0)) %>%
pivot_longer(col = -c(Prefecture,Date), names_to = "item", values_to = "y")厚生労働省のページからデータをRに取り込みます。
県単位でgroup_byでグループ化
陽性者数である列名のNewly confirmed casesを長いのでpに変更
zooパッケージのrollmeanで7日間移動平均、lagで5日ずらして移動平均を割って簡易実効再生産数を計算、mutateで列を足してやり
pivot_longerでggplot-friendlyなtidyデータに変形します。
簡易実効再生産数 (Rt) は国立感染症研究所の以下のページを参考に、7日間移動平均を5日前の7日間移動平均で除した値を採用しました。
COVID-19感染報告者数に基づく簡易実効再生産数推定方法
●推移グラフを作る
まずは本体の陽性者数の推移から
gl <- dat_l %>%
filter(Prefecture == "ALL") %>%
ggplot() +
geom_line(data = subset(dat_l, item == 'p' & Prefecture == "ALL"),
aes(x = ymd(Date), y = y), color = "steelblue", size = 0.3)+
geom_line(data = subset(dat_l, item == 'ma7' & Prefecture == "ALL"),
aes(x = ymd(Date), y = y), color = "red", size = 0.3)+
theme_bw() +
theme(panel.border = element_blank(),
panel.grid.minor = element_blank(),
panel.grid.major = element_blank(),
axis.ticks = element_line(colour = "black"),
axis.ticks.length = unit(1, "mm"),
axis.text.x = element_text(margin = unit(rep(0,4), "mm"), angle = 25, vjust = 1, hjust = 1),
axis.text.y = element_text(margin = unit(rep(1.5, 4), "mm")),
axis.title.y = element_text(margin = margin(t = 0, r = 30, b = 0, l = 0)),
axis.line.x = element_line(colour = 'black', size=0.5, linetype='solid'),
axis.line.y = element_line(colour = 'black', size=0.5, linetype='solid'),
panel.background = element_rect(fill = "transparent",color = NA),
plot.background = element_rect(fill = "transparent",color = NA)) +
scale_x_date(date_breaks="2 month",
labels = date_format("%Y-%m"), # "%Y-%m-%d"
limits = as.Date(c(Sys.Date()-30.5*15, Sys.Date()))) + # x label date_breaks
labs(x = "Date", y = "The number of COVID-19 PCR positives")filter()の中のPrefecture == "ALL"を変えてやる事で都道府県別に作成することができます。
例えばfilter(Prefecture == "Tokyo")などですね。
scale_x_date(limits = as.Date(c(Sys.Date()-30.5*15, Sys.Date())))で15か月分のデータを描写しています。6か月が良ければ30.5*6などにご調整ください。
見た目や後でくっつけるための調整でtheme()の中が激しいことになっていますが、お好みでご調整ください。
次にRt
gRt <- dat_l %>%
filter(Prefecture == "ALL") %>%
ggplot() +
geom_line(data = subset(dat_l, item == 'Rt' & Prefecture == "ALL"),
aes(x = ymd(Date), y = y), color = "red", size = 0.5)+
geom_line(data = subset(dat_l, item == 'Rt' & Prefecture == "ALL"),
aes(x = ymd(Date), y = 1), color = "black", size = 0.5) +
scale_x_date(limits = as.Date(c(Sys.Date()-30.5*15, Sys.Date()))) +
theme_bw() +
ylim(0, 2) +
theme(axis.text.x=element_blank(),
axis.title.x=element_blank(),
axis.text.y = element_text(margin = unit(rep(2,4), "mm")),
axis.ticks = element_line(colour = "black"),
axis.ticks.length = unit(0, "mm"),
panel.background = element_rect(fill = "transparent",color = NA),
plot.background = element_rect(fill = "transparent",color = NA)) +
labs(x = "", y = "Rt",
title = "The number of COVID-19 PCR positives") +
coord_fixed(ratio=40)Rtを描画し、基準値の1のラインを追加してやります。
こちらも上と同様、scale_x_date(limits = as.Date(c(Sys.Date()-30.5*15, Sys.Date())))で15か月分のデータを描写しています。後でくっつける関係上、と同じ値にご調整ください。
y軸範囲はylim(0, 2)、今後Rtが2以上に増えることがあれば(ないことを祈りたいですが)、(0,3)などとしてやる必要があります。
最後に coord_fixed(ratio=40) で横長にしてやります。
●グラフをくっつける
作った二つのグラフをくっつけてやります。
g1 <- ggplot_gtable(ggplot_build(gRt)) %>% suppressWarnings()
g2 <- ggplot_gtable(ggplot_build(gl)) %>% suppressWarnings()
gnew <- gtable_rbind(g1, g2, size="first")
plot(gnew)
ggsave(paste0(Sys.Date(),"_covid_pcr_positive.png"), gnew, bg = "transparent")ggplot_gtable(ggplot_build())でgtableにして、gtable_rbind()でくっつけます。こうすることでx軸がそろって綺麗な感じになります。
plot(gnew)ではgrid線が見えて変な感じですが、ggsave()してやればちゃんとしたグラフが保存されます。
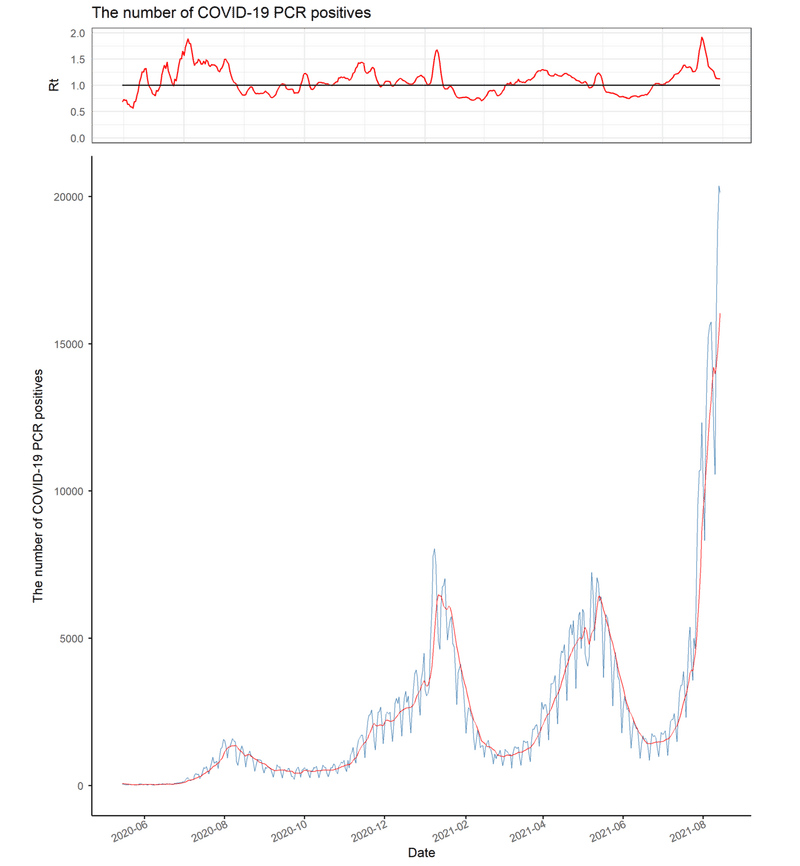
実データ、移動平均、簡易実効再生産数が分かり、傾向分析や過去との比較もしやすい良い感じのデータになりました。
Rtを見ると徐々に上がっているか、急激に上がっているかなどで様々な考察が可能そうです。
●予測データを作る
prophetで選択した都道府県の予測データを作っていきます。
loc <- c("ALL", "Tokyo", "Osaka", "Fukuoka")
date <- 60分析したい都道府県のベクトルをlocに入れ、予想したい期間をdateに入れます。
# Creating model with prophet
prophet_cov<- function(data, pref){
dat <- data %>%
filter(Prefecture == pref) %>%
select(Date, `Newly confirmed cases`)
colnames(dat) <- c("ds","y")
dat <- dat %>% mutate(ds = ymd(ds))
model <- dat %>%
#dplyr::filter(ds >= as.Date("2020-04-01")) %>% # use data after Mar.
# filter(between(ds, as.Date("2020-04-01"), as.Date("2020-12-10"))) # range
prophet(growth = "linear", # or "logistic" 線形トレンドか非線形トレンドか
yearly.seasonality = TRUE,
weekly.seasonality = TRUE,
daily.seasonality = FALSE,
seasonality.mode = "multiplicative", # or "additive"
changepoint.prior.scale = 0.05, # トレンド変化点での傾きの変化の大きさ
changepoint.range = 1, # default0.8, 1で直近のデータまでトレンド変化予想に使用する
n.changepoints = 25 # トレンド変化点候補の数default25
)
# scaleを大きくするならnを小さく、nを大きくするならscaleを小さく調整したほうが良さそう。
future <- make_future_dataframe(model, date) # date日分予想
pred <- predict(model, future)
ftdata <- as_tibble(predict(model, future))
# merge actual and forecast data
forecast <- ftdata %>%
mutate(ds = ymd(ds),
segment = case_when(ds > dat$ds[nrow(dat)]-2 ~ 'forecast',
TRUE ~ 'actual') # SEGMENT ACTUAL VS FORECAST DATA
) %>%
select(ds, segment, yhat_lower, yhat, yhat_upper) %>%
left_join(dat) # JOIN ACTUAL DATA
return(forecast)
}prophetで予測する自作関数を定義してやります。
設定値の詳細は過去の記事かprophetを詳細に解説してくださっているページ様をご参照ください。
lapply(loc, function(x){
prophet_cov(data = dat_x, pref = x)
}) -> res_xlapplyでlocに格納した都道府県ごとに並列して自作関数を処理していきます。
●グラフを作る・並べる
for(i in 1:length(loc)){
g_temp <- res_x[[i]] %>%
dplyr::filter(ds >= Sys.Date()-30) %>%
rename(date = ds,
actual = y) %>%
ggplot() +
geom_line(aes(date, actual)) + # PLOT ACTUALS DATA
geom_line(data = subset(res_x[[i]], segment == 'forecast'),
aes(ds, yhat), color = 'steelblue', size = 0.1) + # PLOT PREDICTION DATA
geom_ribbon(data = subset(res_x[[i]], segment == 'forecast'),
aes(ds, ymin = yhat_lower, ymax = yhat_upper),
fill = 'skyblue', alpha = 0.3) + # SHADE PREDICTION DATA REGION
theme_bw() +
scale_x_date(breaks = function(x) seq.Date(from=ymd(min(res_x[[i]]$ds)),to=ymd(max(res_x[[i]]$ds)),by="7 days"),date_labels="%m/%d") + # x label date_breaks by 7 days
labs(x="date",
y="count",
title = paste0(loc[i])
)
assign(paste("g", i, sep=""), g_temp)
}forループでlocに入れた都道府県ごとに順番にグラフを作ります。
assign(paste("g", i, sep=""), g_temp)でlocごとにg1、g2...と格納してやります。
grid.arrange(g1,g2,g3,g4,
top = "The number of COVID-19 PCR positives (black) and Prediction (blue) by {prophet}",
bottom = url,
left = "The number of COVID-19 PCR positives") %>%
suppressWarnings()gridExtraパッケージのgrid.arrangeで並べ、図の説明を加えてやります。
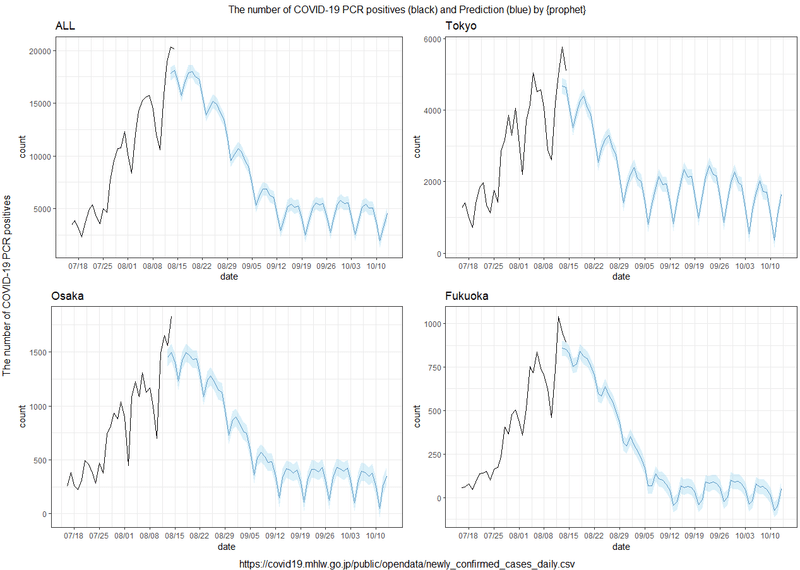
隣接県や仕事で関連のある都道府県で作ってやると便利かもしれません。
以上です。ありがとうございました。
●まとめたコード
Markdownやshinyで表示してやるようにできれば良い感じに出力できるかも。
install.packages("tidyverse", dependencies=TRUE)
install.packages("prophet", dependencies=TRUE)
install.packages("lubridate", dependencies=TRUE)
install.packages("zoo", dependencies=TRUE)
install.packages("scales", dependencies=TRUE)
install.packages("gridExtra", dependencies=TRUE)
library("tidyverse")
library("prophet")
library("lubridate")
library("zoo")
library("scales")
library("gridExtra")
url <- "https://covid19.mhlw.go.jp/public/opendata/newly_confirmed_cases_daily.csv"
dat_x <- readr::read_csv(url,
locale = locale(encoding = "utf8"))
dat_l <- dat_x %>%
group_by(Prefecture) %>%
rename(p = 'Newly confirmed cases') %>%
mutate(ma7 = rollmean(p, 7L, align = "right", fill = 0),
Rt = ma7/lag(ma7, n = 5L, default = 0, fill = 0)) %>%
pivot_longer(col = -c(Prefecture,Date), names_to = "item", values_to = "y")
# https://www.niid.go.jp/niid/ja/diseases/ka/corona-virus/2019-ncov/2502-idsc/iasr-in/10465-496d04.html
gl <- dat_l %>%
filter(Prefecture == "ALL") %>%
ggplot() +
geom_line(data = subset(dat_l, item == 'p' & Prefecture == "ALL"),
aes(x = ymd(Date), y = y), color = "steelblue", size = 0.3)+
geom_line(data = subset(dat_l, item == 'ma7' & Prefecture == "ALL"),
aes(x = ymd(Date), y = y), color = "red", size = 0.3)+
theme_bw() +
theme(panel.border = element_blank(),
panel.grid.minor = element_blank(),
panel.grid.major = element_blank(),
axis.ticks = element_line(colour = "black"),
axis.ticks.length = unit(1, "mm"),
axis.text.x = element_text(margin = unit(rep(0,4), "mm"), angle = 25, vjust = 1, hjust = 1),
axis.text.y = element_text(margin = unit(rep(1.5, 4), "mm")),
axis.title.y = element_text(margin = margin(t = 0, r = 30, b = 0, l = 0)),
axis.line.x = element_line(colour = 'black', size=0.5, linetype='solid'),
axis.line.y = element_line(colour = 'black', size=0.5, linetype='solid'),
panel.background = element_rect(fill = "transparent",color = NA),
plot.background = element_rect(fill = "transparent",color = NA)) +
scale_x_date(date_breaks="2 month",
labels = date_format("%Y-%m"), # "%Y-%m-%d"
limits = as.Date(c(Sys.Date()-30.5*15,Sys.Date()))) + # x label date_breaks
labs(x = "Date", y = "The number of COVID-19 PCR positives")
gRt <- dat_l %>%
filter(Prefecture == "ALL") %>%
ggplot() +
geom_line(data = subset(dat_l, item == 'Rt' & Prefecture == "ALL"),
aes(x = ymd(Date), y = y), color = "red", size = 0.5)+
geom_line(data = subset(dat_l, item == 'Rt' & Prefecture == "ALL"),
aes(x = ymd(Date), y = 1), color = "black", size = 0.5) +
scale_x_date(limits = as.Date(c(Sys.Date()-30.5*15,Sys.Date()))) +
theme_bw() +
ylim(0, 2) +
theme(axis.text.x=element_blank(),
axis.title.x=element_blank(),
axis.text.y = element_text(margin = unit(rep(2,4), "mm")),
axis.ticks = element_line(colour = "black"),
axis.ticks.length = unit(0, "mm"),
panel.background = element_rect(fill = "transparent",color = NA),
plot.background = element_rect(fill = "transparent",color = NA)) +
labs(x = "", y = "Rt",
title = "The number of COVID-19 PCR positives") +
coord_fixed(ratio=40)
g1 <- ggplot_gtable(ggplot_build(gRt)) %>% suppressWarnings()
g2 <- ggplot_gtable(ggplot_build(gl)) %>% suppressWarnings()
gnew <- gtable_rbind(g1, g2, size="first")
plot(gnew)
ggsave(paste0(Sys.Date(),"_covid_pcr_positive.png"), gnew, bg = "transparent")
loc <- c("ALL", "Tokyo", "Osaka", "Fukuoka")
date <- 60
# Creating model with prophet
prophet_cov<- function(data, pref){
dat <- data %>%
filter(Prefecture == pref) %>%
select(Date, `Newly confirmed cases`)
colnames(dat) <- c("ds","y")
dat <- dat %>% mutate(ds = ymd(ds))
model <- dat %>%
#dplyr::filter(ds >= as.Date("2020-04-01")) %>% # use data after Mar.
# filter(between(ds, as.Date("2020-04-01"), as.Date("2020-12-10"))) # range
prophet(growth = "linear", # or "logistic" 線形トレンドか非線形トレンドか
yearly.seasonality = TRUE,
weekly.seasonality = TRUE,
daily.seasonality = FALSE,
seasonality.mode = "multiplicative", # or "additive"
changepoint.prior.scale = 0.05, # トレンド変化点での傾きの変化の大きさ
changepoint.range = 1, # default0.8, 1で直近のデータまでトレンド変化予想に使用する
n.changepoints = 25 # トレンド変化点候補の数default25
)
# scaleを大きくするならnを小さく、nを大きくするならscaleを小さく調整したほうが良さそう。
future <- make_future_dataframe(model, date) # date日分予想
pred <- predict(model, future)
ftdata <- as_tibble(predict(model, future))
# merge actual and forecast data
forecast <- ftdata %>%
mutate(ds = ymd(ds),
segment = case_when(ds > dat$ds[nrow(dat)]-2 ~ 'forecast',
TRUE ~ 'actual') # SEGMENT ACTUAL VS FORECAST DATA
) %>%
select(ds, segment, yhat_lower, yhat, yhat_upper) %>%
left_join(dat) # JOIN ACTUAL DATA
return(forecast)
}
lapply(loc, function(x){
prophet_cov(data = dat_x, pref = x)
}) -> res_x
for(i in 1:length(loc)){
g_temp <- res_x[[i]] %>%
dplyr::filter(ds >= Sys.Date()-30) %>%
rename(date = ds,
actual = y) %>%
ggplot() +
geom_line(aes(date, actual)) + # PLOT ACTUALS DATA
geom_line(data = subset(res_x[[i]], segment == 'forecast'),
aes(ds, yhat), color = 'steelblue', size = 0.1) + # PLOT PREDICTION DATA
geom_ribbon(data = subset(res_x[[i]], segment == 'forecast'),
aes(ds, ymin = yhat_lower, ymax = yhat_upper),
fill = 'skyblue', alpha = 0.3) + # SHADE PREDICTION DATA REGION
theme_bw() +
scale_x_date(breaks = function(x) seq.Date(from=ymd(min(res_x[[i]]$ds)),to=ymd(max(res_x[[i]]$ds)),by="7 days"),date_labels="%m/%d") + # x label date_breaks by 7 days
labs(x="date",
y="count",
title = paste0(loc[i])
)
assign(paste("g", i, sep=""), g_temp)
}
gridExtra::grid.arrange(g1,g2,g3,g4,
top = "The number of COVID-19 PCR positives (black) and Prediction (blue) by {prophet}",
bottom = url,
left = "The number of COVID-19 PCR positives") %>%
suppressWarnings()