【Aidemy X MIT】MITのチュートリアルの教材を利用して、ワインの特性データのクラスタリングを深めてみる!
インフルエンザウィルス研究データーを利用したMITのscikit-learnチュートリアル
数年前に今はノバルティスファーマにいらっしゃるデータサイエンティストEric Maさんが行った機械学習のチュートリアルを受けたことがあったが、当時はpythonが今ひとつ分かっていなかったため、消化不良に終わったのであるが、最近Aidemyでコースを受講して概要がつかめたので、再度その教材(Githubでシェアされている)を見てみるとすごく面白い!
インフルエンザの遺伝子をタンパク配列(文字情報)をアミノ酸の等電点の情報に変換して数値化し、それを機械学習に利用した彼の研究の素材をそのままチュートリアルの教材としてつかっているのだ!
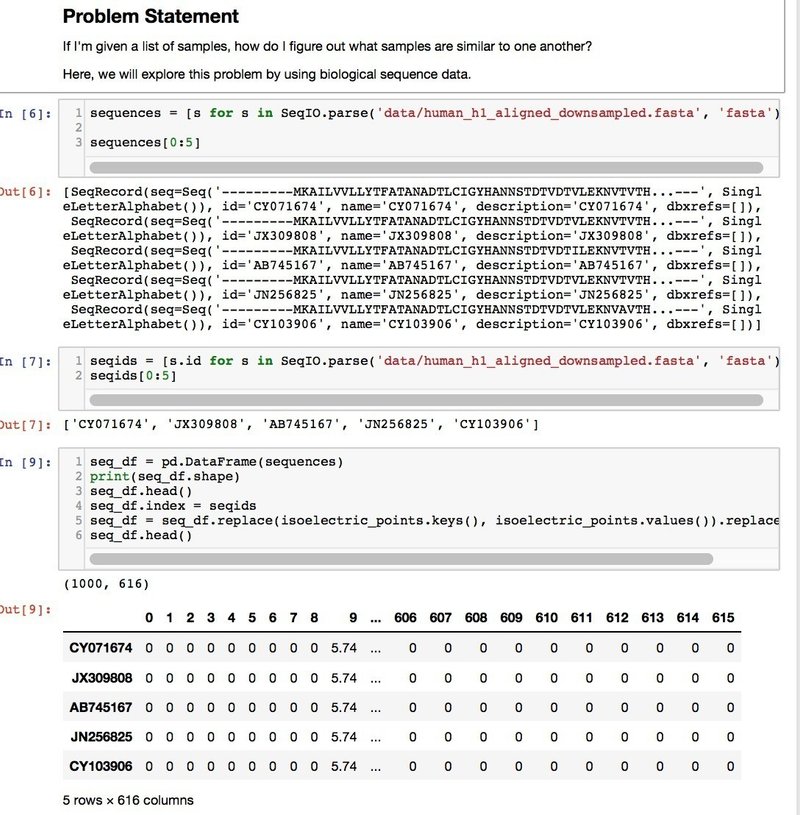
実際の事例をつかっている貴重な教材だし、割と基礎的なところから教師なし学習をみっちり伝えようとしていて今から思うと興味深い。
Aidemy教材のワインのクラスタリング
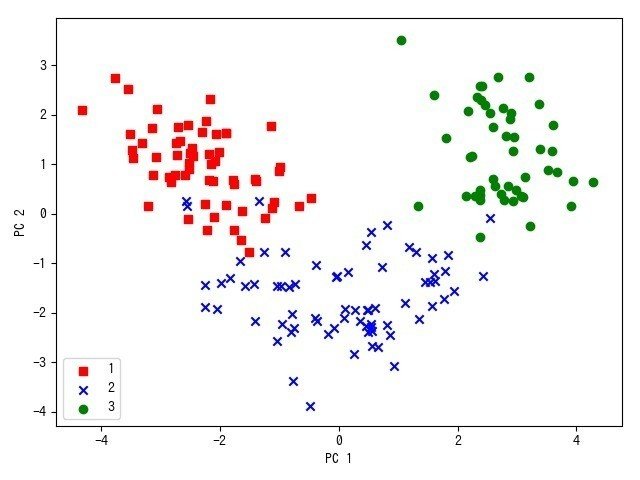
Aidemyの教師なし学習のコースには(上記)、ワインの特性のデータをつかった主因子分析(PCA)の教材があるのだけれど、これが遺伝子情報とか画像情報とか医学系の研究者が現場で行っている解析の事例に近くて、非常に興味深いのだけれど、Eric Maさんの教材をみているのこのワインの特性データーがそのまま使えるということに気づいて組み合わせてみようかなと思った!
このワインの特性データはもともとscikit-learnについているもので、3つのグループに分けられた178種類のワインについて、以下の13個の属性について数値データが載せられているものである。
1) Alcohol
2) Malic acid
3) Ash
4) Alcalinity of ash
5) Magnesium
6) Total phenols
7) Flavanoids
8) Nonflavanoid phenols
9) Proanthocyanins
10)Color intensity
11)Hue
12)OD280/OD315 of diluted wines
13)Proline
MITのクラスタリング教材
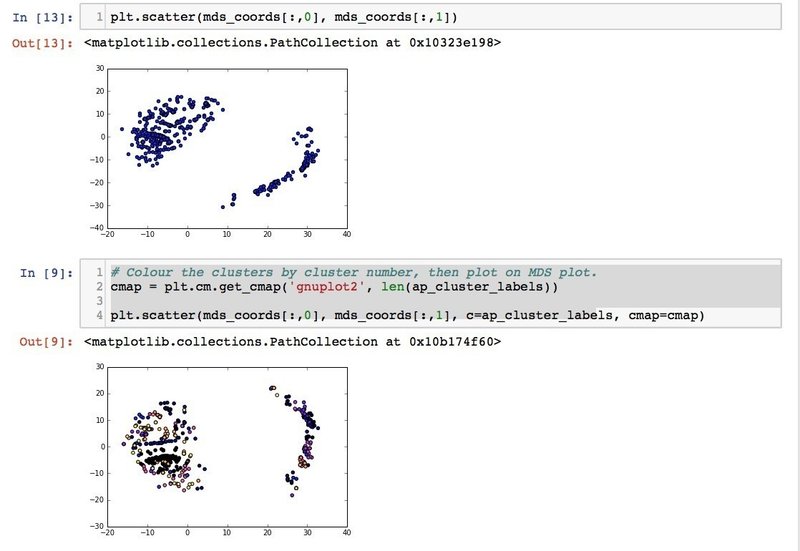
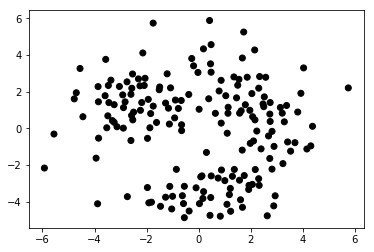
Eric Maさんの教材のうちまずはクラスタリングの部分が扱いやすいので、これを用いてワインの特性データを解析してみた。Eric Maさんはまず、PCAに似た手法であるMulti-dimensional Scaling(MDS)でデータの次元を落としてサンプルの全体像が俯瞰できるようにした後、AffinityPropagation, Kmeans, DBSCANといった各種手法でクラスタリングを行い、クラスタリング結果をMDSのプロットにオーバーレイすることで、手法の違いにより クラスタリング結果に違いがでることをチュートリアルで述べていた!
ワイン特性データでやってみると、
まずはMDSプロット
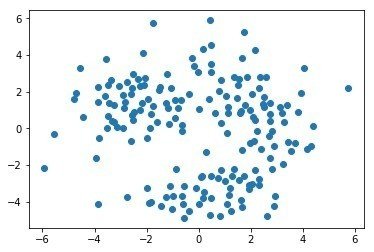
Affinity propagation
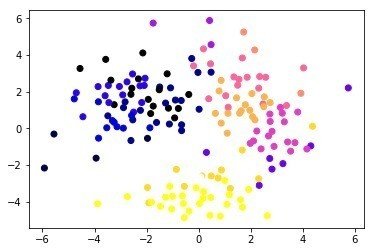
KMeans(n=5)
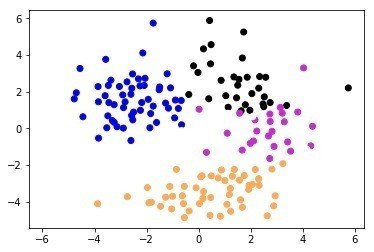
DBSCAN
SpectralClustering
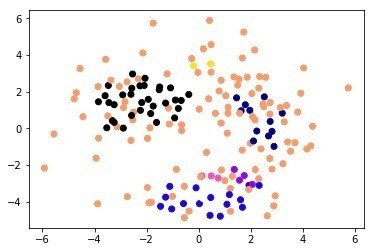
またそれと同時にクラスタリングの妥当性を、シルエットスコアを計算し評価していた
シルエットスコアについては

で計算され、
Affinitypropagation = 0.12094943296335095,
KMeans = 0.23476387776939125,
DBSCAN=NA(分類できずうまくでない),
SpectralClustering=-0.11590011647908852
であり、Kmeansクラスタリングがこの場合一番妥当なのかと思われた。
MDS, tSNE, Isomap(PCA)は意外と違う?
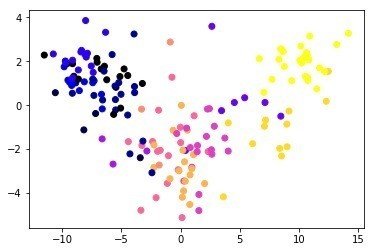
あと以下はおまけであるが、データーの次元を減らす方法(Manifold Learningっていうらしい)としてはtSNE(ティズニーって言います)やIsomapという方法があり、これも応用として試してみるような課題がでていた。tSNEはFACS、マス解析、single cell 解析でもよくつかあれ医学系研究者にはおなじみである。個人的にはPCA(Isomapに近いらしい),MDS, tSNEってほとんど変わらないのではないかと思っていたが、意外と違うものである!
MDS
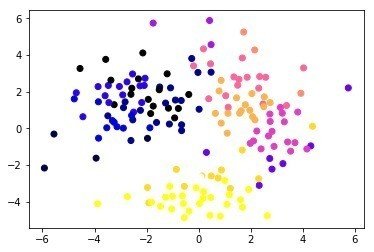
tSNE

Isomap
ここで用いたコードはEric Maさんのシェアされている教材を基にして改変したもので以下で参照できます。
この記事が気に入ったらサポートをしてみませんか?
