GPT index で論文読ませて、超マニアックな専門知識を必要とする質問チャットボット作成
GPT Index で専門知識を必要とする質問応答チャットボットを簡単作成|npaka @npaka123 #note https://t.co/Q35c8HKoZQ
— 布留川英一 / Hidekazu Furukawa (@npaka123) January 14, 2023
最近ChatGPTが大流行りですね。そのChatGPTに似た技術で自分の情報を学習させてカスタマイズさせたチャットボットを作る手法もいくつか出てきました。
今日ツィッターをみていたら、布留川さんという方が、そのカスタマイズチャットボットを作るGPT indexを試されていたnoteを書かれていましたが。。
拝見してみるとすごく簡単そうだったので、自分でも試してみました。
Input ファイルの準備
用意したのは
I'm pleased to share a new review article on functional connections between RNA splicing and the hallmarks of #cancer that I wrote with @OlgaAnczukow, just out in @NatureRevCancer https://t.co/iNJuHOFrEZ
— Robert K. Bradley (@bradleybio) January 10, 2023
このRNAスプライシングの最新の知見を集めたレビューの一節を利用し
Recurrent mutations in splicing factors
Recurrent somatic mutations in SF3B1, SRSF2, U2AF1 and ZRSR2 occur frequently in haematological malignancies, including in myelodysplas- tic syndromes (MDS), chronic myelomonocytic leukaemia (CMML), acute myeloid leukaemia (AML) and chronic lymphocytic leukaemia (CLL)19,20 (Fig. 2a,b). These mutations are frequently termed ‘spliceo- somal mutations’. SF3B1 and U2AF1 are also recurrently mutated in diverse solid tumour types7,21?23. Mutations in SF3B1, SRSF2 and U2AF1 almost always occur as heterozygous missense point mutations affect- ing specific residues in both haematological malignancies and solid tumours, whereas mutations in the X-linked gene ZRSR2 frequently disrupt its open reading frame and preferentially occur in males. Detailed functional studies have revealed that recurrent SF3B1, SRSF2 and U2AF1 mutations cause gain or alteration of function, whereas ZRSR2 mutations cause loss of function, consistent with the spectra of mutations observed in patients. Spliceosomal mutations are almost always mutually exclusive as they elicit redundant and/or synthetically lethal effects due to their cumulative impact on alternative splicing and haematopoiesis24, although there are rare exceptions to this rule25.
SF3B1 is the most frequently mutated spliceosomal component in cancer, with recurrent somatic mutations detected in ~30% of all patients with MDS, including 83% of cases of MDS-subtype refractory anaemia with ring sideroblasts and 76% of cases of MDS-subtype refrac- tory cytopenia with multilineage dysplasia and ring sideroblasts19,26. SF3B1 mutations are also detected in other cancers, including 15% of CLL, 3% of pancreatic cancer, 1.8% of breast adenocarcinomas, 1% of cutaneous melanomas and 20% of uveal melanomas (UVMs)21,22 (Fig. 2a). SF3B1 is a core component of the U2 snRNP that is involved in BPS recognition and spliceosomal complex A assembly (Fig. 1). SF3B1 mutations near-universally occur as heterozygous, missense mutations that affect multiple hot-spot residues within the carboxy-terminal HEAT domains (HDs) (Fig. 2b). These mutations induce altered BPS recognition with consequent changes in 3′SS recognition, resulting in widespread splicing alterations including cryptic 3′SS usage, dif- ferential cassette exon inclusion and reduced intron retention27,28. The prognostic implications of an SF3B1 mutation depend upon the specific mutation and indication. For example, SF3B1K700E is associated with comparatively good prognosis in MDS with ring sideroblasts19,20, whereas SF3B1K666N is associated with disease progression29. In CLL, SF3B1G742D correlates with poor prognosis26. Although how mutant SF3B1 promotes disease phenotypes and tumorigenesis is still under active investigation, numerous cellular pathways have been implicated. For example, SF3B1 mutations cause aberrant inclusion of a poison exon (an exon that contains an in-frame premature termination codon (PTC)) in bromodomain containing 9 (BRD9) across tumour types to promote cell transformation30, induce MAP3K7 mis-splicing to promote hyperac- tive nuclear factor-κB (NF-κB) signalling and disrupt erythropoiesis24,31, and disrupt splicing of genes involve
Recurrent mutations in splicing factors.txt
というファイルを作り、Google ドライブの所定の場所に入れておきます。
あらかじめGoogleドライブの中に
chatGPTというフォルダを作り
その中にdataというフォルダを作り
その中にファイルを入れました
Google colabでの実行
Google driveのマウント
import os
import shutil
from requests import get
from google.colab import drive, files
drive.mount('/content/drive')パッケージのインストール
# パッケージのインストール
!pip install gpt-index# chatGPTフォルダへの移動
os.chdir('/content/drive/My Drive/chatGPT')
cwd = os.getcwd()
print(cwd)
os.listdir("./")APIキーの読み込み
次にGPT indexを使うためにOpen AiのAPIキーを入れます
import os os.environ["OPENAI_API_KEY"] = "YOUR API キー"APIキーはあらかじめ以下のOpen Aiのサイトからもらっておきます。
ここまでできたらいよいよ次はファイルの読み込みとインデックスの作成です。
インデックスの作成
from gpt_index import GPTSimpleVectorIndex, SimpleDirectoryReader
# インデックスの作成
documents = SimpleDirectoryReader('data').load_data()
index = GPTSimpleVectorIndex(documents)dataディレクトリにあるファイルを読み込んで、インデックスを作っておしまい。
このくらいの長さのファイルだと数十秒で作業が終わります。

これが終われば準備万端。
作ったシステムに質疑応答です。
読ませた文章はRNAスプライシングのがんにおける役割をまとめたもの。
まずはRNAスプライシング因子のスーパースターSF3B1について聞いてみましょう!
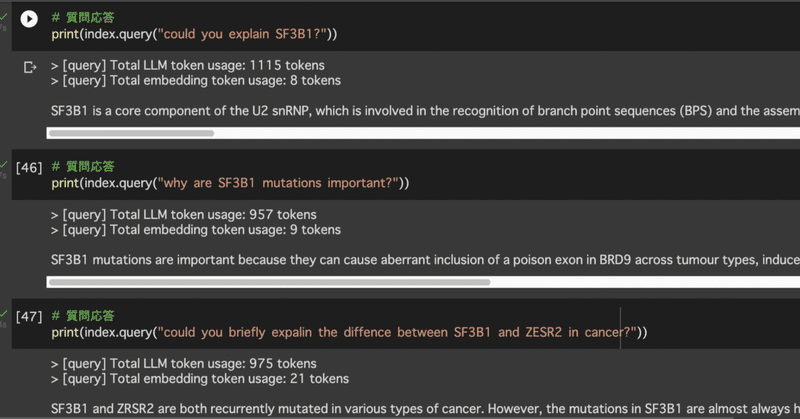
# 質問応答
print(index.query("could you explain SF3B1?"))
SF3B1 is a core component of the U2 snRNP, which is involved in the recognition of branch point sequences (BPS) and the assembly of the spliceosomal complex A. It is the most frequently mutated spliceosomal component in cancer, with recurrent somatic mutations detected in ~30% of all patients with myelodysplastic syndromes (MDS), chronic myelomonocytic leukaemia (CMML), acute myeloid leukaemia (AML) and chronic lymphocytic leukaemia (CLL). SF3B1 mutations are also detected in other cancers, including pancreatic cancer, breast adenocarcinomas, cutaneous melanomas and uveal melanomas (UVMs). These mutations are almost always heterozygous, missense mutations that affect multiple hot-spot residues within the carboxy-terminal HEAT domains (HDs). These mutations can cause altered BPS recognition, resulting in changes in 3SS recognition, cryptic 3SS usage, differential cassette exon inclusion and reduced intron retention. The prognostic implications of an SF3B1 mutation depend upon the specific mutation and indication.
元の文章をエクストラクトしているだけですが、いい感じのSF3B1のまとめになっていますね。優秀な大学生くらいのレベルはあります。
次にちょっと難しめの質問、なんでSF3B1の変異が重要なのか聞いてみます!
# 質問応答
print(index.query("why are SF3B1 mutations important?"))
SF3B1 mutations are important because they can cause aberrant inclusion of a poison exon in BRD9 across tumour types, induce MAP3K7 mis-splicing to promote hyperactive NF-B signalling, and disrupt erythropoiesis and splicing of genes involved in cell transformation, proliferation, and survival. These mutations can also have prognostic implications depending on the specific mutation and indication.
これはかなり高度に情報を要約していますね。SF3B1が色々な腫瘍でBRD9の余分なエクソンを挿入し、MAP3Kのミススプラインシングを誘導し、さまざまな悪影響を与えることをうまくまとめています。またがんの予後に関わることも漏らさず付け加えていますね。
次にSF3B1とZRSR2のがんにおける違いについて手短に説明してもらいます。
# 質問応答
print(index.query("could you briefly expalin the diffence between SF3B1 and ZRSR2 in cancer?"))
この時 ZRSR2をタイプミスして見舞ったのですが、優しくさらっと無視して的確な答えを返してくれました。
SF3B1 and ZRSR2 are both recurrently mutated in various types of cancer. However, the mutations in SF3B1 are almost always heterozygous missense point mutations affecting specific residues, while mutations in ZRSR2 frequently disrupt its open reading frame and preferentially occur in males. Additionally, the mutations in SF3B1 cause gain or alteration of function, while ZRSR2 mutations cause loss of function.
SF3B1と ZRSR2の違いについて簡潔にまとめてくれているのが、わかります。二つともがんでよく変異が見られるが、SF3BF1は点突然変異によるヘテロ変異が多いが、 ZRSR2変異はフレームシフトが多く男性に多い。
またSF3B1変異ははgain of functionもしくは機能の変化が起こるが、 ZRSR2はloss of function変異が多いと。
専門家もビックリのほぼパーフェクトな回答で驚きました(SF3B1がgain-of-functionとははっきり本文に書いていないのがちょっと惜しいです)!これだと専門家を凌駕するようなチャットボットもすぐ作れそうです!
最後に彼の考えも聞いてみましょう!
# 質問応答
print(index.query("what do you think of SF3B1?"))
SF3B1 appears to be a key component of the spliceosome and is recurrently mutated in a variety of cancers, including myelodysplastic syndromes (MDS), chronic myelomonocytic leukaemia (CMML), acute myeloid leukaemia (AML) and chronic lymphocytic leukaemia (CLL). The mutations are almost always heterozygous missense point mutations affecting specific residues, and they can cause gain or alteration of function, or loss of function. The prognostic implications of an SF3B1 mutation depend on the specific mutation and indication. SF3B1 mutations have been linked to aberrant inclusion of a poison exon, MAP3K7 mis-splicing, and disruption of erythropoiesis and other gene splicing. Overall, SF3B1 appears to be an important factor in cancer development and progression.
最初の文はいいですね良い回答です。機能の説明はちょっと拡大解釈し過ぎですが、あとはまあ許容範囲なまとめになっていますね。
ちなみにせっかく作ったindexはまた使えるかも知れないので、セーブしておきます。
# save to disk
index.save_to_disk('index.json')セーブしたインデックスは
# load from disk
index = GPTTreeIndex.load_from_disk('index.json')
というコマンドで読み込んで使うことができるようです。
最後までお読みいただきありがとうございます。
非常に簡単に専門性の高いチャットボットが作れてしまうので、驚きというか脅威を感じてしまいました。
まじかこれ!GPT indexに論文読ましたら、専門的な回答してくれるじゃん?
— Toshi⁷ MD,PhD 🇺🇸|アメリカで研究&開発 (@tokitky) January 15, 2023
SF3B1と ZRSR2のがんでの働きの違いについて聞いてみたら?
論文に則ってきちんと答えてくれる! https://t.co/bGnATNmKBT pic.twitter.com/VlUYKZ0kiq
この記事が気に入ったらサポートをしてみませんか?
