【Aidemy X Bio】がん細胞データをつかって、機械学習最先端手法Constrastive PCAをつかってみる
これまで乳がん細胞と正常細胞の遺伝子発現データをつかって、Aidemyの機械学習のコードを利用しながらいくつかプラットフォームをつくってきた。
【Aidemy X Bio】Aidemyのコースを応用して、遺伝子解析データ用の教師なし学習プラットフォームを作ってみた
【Aidemy X Bio】機械学習はがん細胞を見分けられるか?:遺伝子解析データをもとに教師あり学習(分類)を行ってみる!
教師なし学習の一つの手法であるPCAであるが、データ間のvariationが大きい場合、うまくクラスターがわかれないことがある。これを克服するためにコントロール群のデータvariationを前もって除去しておいて、データ内のクラスター間の分離を良くする手法Contrastive PCAが最近Stanford大のJames Zouらにより
Nature Communications volume 9, Article number: 2134 (2018)
に発表された。
著者による説明は以下のyoutubeで参照できる
このContrastive PCAについて彼らはpython libraryを作り、チュートリアルをGithubで公開しているので、
これを利用して、乳がん細胞と正常細胞の遺伝子発現データを利用して、Contrastive PCAをおこなってみた!
%matplotlib inline
from sklearn.cluster import AffinityPropagation, KMeans, DBSCAN, SpectralClustering
from sklearn.manifold import MDS, TSNE, Isomap
from sklearn.metrics import silhouette_score
import numpy as np
import matplotlib.pyplot as plt
import pandas as pd
from scipy.linalg import logm, expm
from contrastive import CPCAライブラリを読み込み
df_wine = pd.read_csv("TNBC10vNormal10_cpm_2.csv", sep=",",header=0, index_col=0)
df_wine_2 = df_wine.T
print(df_wine_2)
df_wine_2 = pd.DataFrame(df_wine_2)
X,y = df_wine_2.iloc[:, :].values, np.array([1,1,1,1,1,1,1,1,1,1,0,0,0,0,0,0,0,0,0,0])データの読み込み
foreground_data = X[:,:]
background_data = X[10:20,:]
background_data
mdl = CPCA()
pre_cluster_lables = np.array([1,1,1,1,1,1,1,1,1,1,0,0,0,0,0,0,0,0,0,0])
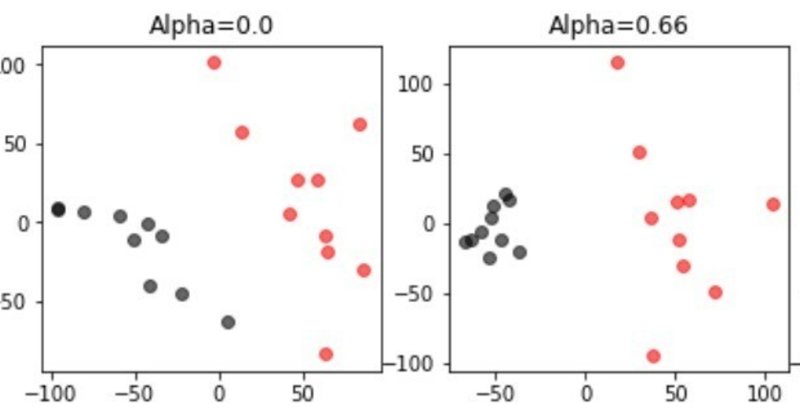
projected_data = mdl.fit_transform(foreground_data, background_data, plot=True,active_labels=pre_cluster_lables)学習用データと、バックグラウンドデータをアサインし、教師なし学習させた。学習用データとして全データを、バックグラウンドデータを正常細胞データとする。

alphaの値が大きくなるに従って、バッククラウンドデータの正常細胞のクラスターが小さくなり、正常細胞(黒)とがん細胞(赤)の差が広がっているのがわかる。
foreground_data = X[:,:]
background_data = X[18:20,:]
background_data
mdl = CPCA()
pre_cluster_lables = np.array([1,1,1,1,1,1,1,1,1,1,0,0,0,0,0,0,0,0,0,0])
projected_data = mdl.fit_transform(foreground_data, background_data, plot=True,active_labels=pre_cluster_lables)正常細胞の一部をバックグラウンドデータとしてみても

同様の傾向が得られる
もともとそこそこよく分かれているデータなのでいまひとつ有効性がわかりにくが、チュートリアルによるデモの図だと、分離の悪いクラスター群がきちんと4群に分かれることがわかり。こうしたPCA等の手法はできるだけ多くのクラスターを見いだすことが重要であるから、この手法はかなりパワフルであると考えられる。
まずはContrastive PCAの導入がうまくいったようなので、今後これを利用していろいろ知見を深めていきたいと思う。またこの3月には機械学習はまったくの素人と言っていいほどだったので、Aidemyのコースをやったおかげで機械学習がある程度できるようになり、こんな風に新しい知見を取り入れていろいろ検討できるようになったのはありがたい。
この記事が気に入ったらサポートをしてみませんか?
