転移学習を用いた肺炎の診断プログラムの作成🫁
1.自己紹介
はじめまして、診療放射線技師(レントゲン技師)3年目の社会人です。
データサイエンティストに興味が湧き、Aidemyデータ分析講座6ヶ月コースを受講しました。
2.目的
本職であるレントゲンとPythonをうまく組み合わせられないか考えたところ、健常者と肺炎患者のデータセットを用いて判定プログラムを作成することにしました。
3.開発環境
Python3
Macbook Air
Google Colaboratory
4.データセット説明
今回のデータセットはnormal(健常者)とpneumonia(肺炎患者)が事前にラベリングされたものになります。
健常者の胸部画像を表示します。
基本的な解剖名称を記載しました。レントゲン画像は見にくいと思いますが、左右が反転していますので注意してください。

肺炎患者の胸部画像を表示します。
右肺野のやや白くもやがかかったようなところが肺炎です。

5.コード解説
必要なライブラリをインポート
!pip install japanize-matplotlib
import japanize_matplotlib
import os
import cv2
import numpy as np
import matplotlib.pyplot as plt
from tensorflow.keras.utils import to_categorical
from tensorflow.keras.layers import Dense, Dropout, Flatten, Input
from tensorflow.keras.applications.vgg16 import VGG16
from tensorflow.keras.models import Model, Sequential
from tensorflow.keras import optimizers健常者、肺炎患者のパスをとってきてそれぞれリストに代入する
今回の学習に用いるデータはnormal(健常者):300枚、pneumonia(肺炎患者):300枚となっています。
#トレーニングデータ
path_normal_train = os.listdir("/content/drive/MyDrive/Aidemy/成果物/chest_xray_300/train/NORMAL/")
path_pneumonia_train = os.listdir("/content/drive/MyDrive/Aidemy/成果物/chest_xray_300/train/PNEUMONIA/")
画像のフォーマットを整える
img_normal_train = []
img_pneumonia_train = []
for i in range(len(path_normal_train)):
img = cv2.imread("/content/drive/MyDrive/Aidemy/成果物/chest_xray_300/train/NORMAL/" + path_normal_train[i])
b,g,r = cv2.split(img)
img = cv2.merge([r,g,b])
img = cv2.resize(img, (50,50))
img_normal_train.append(img)
for i in range(len(path_pneumonia_train)):
img = cv2.imread("/content/drive/MyDrive/Aidemy/成果物/chest_xray_300/train/PNEUMONIA/" + path_pneumonia_train[i])
b,g,r = cv2.split(img)
img = cv2.merge([r,g,b])
img = cv2.resize(img, (50,50))
img_pneumonia_train.append(img)
X_train = np.array(img_normal_train + img_pneumonia_train)
y_train = np.array([0]*len(img_normal_train) + [1]*len(img_pneumonia_train))
rand_index = np.random.permutation(np.arange(len(X_train)))
X_train = X_train[rand_index]
y_train = y_train[rand_index]
y_train = to_categorical(y_train)path_normal_trainから一つずつパスを取ってきてi番目の画像のフォーマットを一枚ずつ整える。まず一枚取ってきて、cv2imread()で画像を読み込むとRGBがBGRとして読み込まれていまうので、これを回避するためにb,g,rにそれぞれ分解して、r,g,bに並べ直している。その後、さまざまな種類の胸部画像が混在しているのでサイズを50×50でリサイズしました。そして、事前に作っていたimg_normal_trainのからのリストに入れていきます。
同様のことをpneumoniaにも行っていきます。
トレーニングデータの作成
img_normal_trainとimg_pneumonia_trainを結合して、ランダムにシャッフルします。こうすることで学習が上手に行うことができるようになります。y_trainに関しては、to_categorical()を用いてone-hotベクトルとしています。
img_normal_train = []
img_pneumonia_train = []
for i in range(len(path_normal_train)):
img = cv2.imread("/content/drive/MyDrive/Aidemy/成果物/chest_xray_300/train/NORMAL/" + path_normal_train[i])
b,g,r = cv2.split(img)
img = cv2.merge([r,g,b])
img = cv2.resize(img, (50,50))
img_normal_train.append(img)
for i in range(len(path_pneumonia_train)):
img = cv2.imread("/content/drive/MyDrive/Aidemy/成果物/chest_xray_300/train/PNEUMONIA/" + path_pneumonia_train[i])
b,g,r = cv2.split(img)
img = cv2.merge([r,g,b])
img = cv2.resize(img, (50,50))
img_pneumonia_train.append(img)
X_train = np.array(img_normal_train + img_pneumonia_train)
y_train = np.array([0]*len(img_normal_train) + [1]*len(img_pneumonia_train))
rand_index = np.random.permutation(np.arange(len(X_train)))
X_train = X_train[rand_index]
y_train = y_train[rand_index]
y_train = to_categorical(y_train)テストデータの作成
上記の内容をテストデータでも行っていきます。
path_normal_test = os.listdir("/content/drive/MyDrive/Aidemy/成果物/chest_xray_300/test/NORMAL/")
path_pneumonia_test = os.listdir("/content/drive/MyDrive/Aidemy/成果物/chest_xray_300/test/PNEUMONIA/")
img_normal_test = []
img_pneumonia_test = []
for i in range(len(path_normal_test)):
img = cv2.imread("/content/drive/MyDrive/Aidemy/成果物/chest_xray_300/test/NORMAL/"+ path_normal_test[i])
b,g,r = cv2.split(img)
img = cv2.merge([r,g,b])
img = cv2.resize(img, (50,50))
img_normal_test.append(img)
for i in range(len(path_normal_test)):
img = cv2.imread("/content/drive/MyDrive/Aidemy/成果物/chest_xray_300/test/PNEUMONIA/"+ path_pneumonia_test[i])
b,g,r = cv2.split(img)
img = cv2.merge([r,g,b])
img = cv2.resize(img, (50,50))
img_pneumonia_test.append(img)
X_test = np.array(img_normal_test + img_pneumonia_test)
y_test = np.array([0]*len(img_normal_test) + [1]*len(img_pneumonia_test))
rand_index = np.random.permutation(np.arange(len(X_test)))
X_test = X_test[rand_index]
y_test = y_test[rand_index]
y_test = to_categorical(y_test)
VGG16のインスタンスの生成
上記のコードでトレーニングデータ、テストデータの準備が終わりました。
続いて、VGG16のインスタンスを作成していきます。
VGG16(Visual Geometry Group-16)とは、コンピュータビジョン分野で広く使用されている深層学習モデルの一つです。VGG16は、2014年にKaren SimonyanとAndrew Zissermanによって開発されたモデルで、イギリスのオックスフォード大学のVisual Geometry Groupに由来する名前です。
VGG16は、主に画像分類タスクに使用され、非常に深いネットワーク層を持っています。具体的には、16層の畳み込み層とプーリング層からなり、最後に完全に接続された3つの全結合層から構成されます。VGG16は非常にシンプルな畳み込みブロックの設計を用いており、3x3の小さなカーネルを持つ畳み込み層と、2x2のプーリング層を交互に積み重ねることにより、深いネットワークを構築しています。
input_tensor = Input(shape=(50,50,3))
vgg16 = VGG16(include_top=False, weights="imagenet", input_tensor=input_tensor)input_tensor
インプットする画像のサイズを指定しています。今回はresizeで50×50として、3はRGB(=カラー画像)を意味しています。
include_top = False
include_topをFalseにし、VGGの特徴抽出部分のみを用いてそれ以降のモデルは自分で作成したモデル(top_model)と結合させます。
weights = "imagenet"
weightsを"imagenet"と指定するとImageNetで学習した重みを用いることができます。
上流モデルの作成
top_model = Sequential()
top_model.add(Flatten(input_shape=vgg16.output_shape[1:]))
top_model.add(Dense(256, activation="relu"))
top_model.add(Dropout(rate=0.5))
top_model.add(Dense(2, activation="softmax"))今回は転移学習を用いて学習を行なっていくので、VGGの特徴抽出部分のみを用いてそれ以降のモデルは自分で作成したモデル(top_model)と結合させます。
転移学習とは、あるタスクで学習したモデルの知識や特徴を、他の関連するタスクでの学習に活用する手法です。通常、新しいタスクに対してゼロからモデルを訓練する代わりに、転移学習はソースモデルから学習した特徴や知識を再利用して、新しいタスクに適応させることができます。
モデルの連結
model = Model(vgg16.inputs, top_model(vgg16.output))Model()クラスを用いてVGG16とtop_modelを連結させていきます。
VGG16の層の重みを変更不能にする
for layer in model.layers[:19]:
layer.trainable = FalseVGG16の18層目までの重みをtrainable = Falseとすることで変更をできなくすることができます。
コンパイルする
コンパイルとは、ニューラルネットワークで学習するときは、まずどんな方式で学習するかを決めることをいいます。
#コンパイルを行う
model.compile(loss="categorical_crossentropy",
optimizer=optimizers.SGD(lr=1e-4, momentum=0.9),
metrics=["accuracy"])loss = "categorical_crossentropy"
損失関数を"categorical_crossentropy"に指定しました。
optimizer = optimizers.SGD(lr=1e-4, momentum=0.9)
最適化アルゴリズムをSGDに指定しました。転移学習する場合、最適化関数はSGDを選択するのが良いとされています。
metrics = ["accuracy"]
評価指標を正解率としています。
学習をする&精度評価
#学習を行う
history = model.fit(X_train, y_train, batch_size=100, epochs=16, validation_data=(X_test, y_test))
# 精度の評価
test_loss, test_acc = model.evaluate(X_test, y_test, verbose=1)
print('Test loss:', test_loss) print('Test accuracy:', test_acc)model.fit()を用いてモデルの学習を行っていきます。batch_sizeを100、epochsを16回として、その学習の評価データ(X_test, y_test)で行います。
モデルの評価をmodel.evaluate()を用いて行います。それぞれの変数に誤差と正解率を代入して出力します。
下記は学習状況を表しています。
Epoch 1/16
7/7 [==============================] - 39s 6s/step - loss: 30.6538 - accuracy: 0.6522 - val_loss: 19.6878 - val_accuracy: 0.5684
Epoch 2/16
7/7 [==============================] - 38s 6s/step - loss: 24.0122 - accuracy: 0.7787 - val_loss: 21.4454 - val_accuracy: 0.7778
Epoch 3/16
7/7 [==============================] - 31s 5s/step - loss: 5.9798 - accuracy: 0.8769 - val_loss: 8.9904 - val_accuracy: 0.8226
Epoch 4/16
7/7 [==============================] - 38s 6s/step - loss: 1.3769 - accuracy: 0.9334 - val_loss: 2.9972 - val_accuracy: 0.7991
Epoch 5/16
7/7 [==============================] - 38s 6s/step - loss: 0.4605 - accuracy: 0.9118 - val_loss: 1.4198 - val_accuracy: 0.8355
Epoch 6/16
7/7 [==============================] - 31s 5s/step - loss: 0.2416 - accuracy: 0.8819 - val_loss: 1.2338 - val_accuracy: 0.8397
Epoch 7/16
7/7 [==============================] - 39s 6s/step - loss: 0.2978 - accuracy: 0.8502 - val_loss: 1.3104 - val_accuracy: 0.8248
Epoch 8/16
7/7 [==============================] - 38s 6s/step - loss: 0.2291 - accuracy: 0.8536 - val_loss: 1.7744 - val_accuracy: 0.8077
Epoch 9/16
7/7 [==============================] - 39s 6s/step - loss: 0.5000 - accuracy: 0.8869 - val_loss: 3.4386 - val_accuracy: 0.7543
Epoch 10/16
7/7 [==============================] - 38s 6s/step - loss: 0.4341 - accuracy: 0.9135 - val_loss: 7.8590 - val_accuracy: 0.7543
Epoch 11/16
7/7 [==============================] - 38s 6s/step - loss: 0.5582 - accuracy: 0.9235 - val_loss: 1.3290 - val_accuracy: 0.8462
Epoch 12/16
7/7 [==============================] - 38s 6s/step - loss: 0.1762 - accuracy: 0.9268 - val_loss: 1.3248 - val_accuracy: 0.8312
Epoch 13/16
7/7 [==============================] - 31s 5s/step - loss: 0.1981 - accuracy: 0.9035 - val_loss: 1.3868 - val_accuracy: 0.8098
Epoch 14/16
7/7 [==============================] - 38s 6s/step - loss: 0.1580 - accuracy: 0.8985 - val_loss: 1.6350 - val_accuracy: 0.8184
Epoch 15/16
7/7 [==============================] - 40s 6s/step - loss: 0.1686 - accuracy: 0.9235 - val_loss: 1.9019 - val_accuracy: 0.8312
Epoch 16/16
7/7 [==============================] - 38s 6s/step - loss: 0.1381 - accuracy: 0.9235 - val_loss: 1.8786 - val_accuracy: 0.8226
15/15 [==============================] - 14s 950ms/step - loss: 1.8786 - accuracy: 0.8226
Test loss: 1.8786150217056274
Test accuracy: 0.8226495981216431今回の学習では以下のような結果になりました。
Test loss: 1.8786150217056274
Test accuracy: 0.8226495981216431
正解率は約82.23%という結果になりました。
学習状況のグラフ化
今回の学習がどのように行われているのか折れ線グラフでグラフかしてみました。
#学習状況のグラフを表示
param = [["正解率", "accuracy", "val_accuracy"],
["誤差", "loss", "val_loss"]]
plt.figure(figsize=(10,4))
for i in range(2):
plt.subplot(1,2,i+1)
plt.title(param[i][0])
plt.plot(history.history[param[i][1]], "-o")
plt.plot(history.history[param[i][2]], "-o")
plt.xlabel("epochs")
plt.legend(["train", "test"], loc="best")
if i == 0:
plt.ylim([0,1])
plt.show()
グラフから、epoch数が増えていくにつれて正解率は上昇しているのがわかります。誤差もかなり低くなっています。
実際に予測してみる
実際に画像を用いて正しく判断できているか、実験していきたいと思います。doctor関数を作成して判定してもらいます。
#健常者か肺炎患者かを判定する関数
def doctor(img):
img = cv2.resize(img, (50, 50))
pred = np.argmax(model.predict(np.array([img])))
if pred == 0:
return "normal"
else:
return "pneumonia"適当にデータを指定します。今回はpneumoniaのデータを読み込ませたいと思います。pneumoniaと表示できれば正解です。
img = cv2.imread("/content/drive/MyDrive/Aidemy/成果物/chest_xray/val/PNEUMONIA/person1946_bacteria_4874.jpeg")
b,g,r = cv2.split(img)
img1 = cv2.merge([r,g,b])
plt.imshow(img1)
plt.show()
print(doctor(img))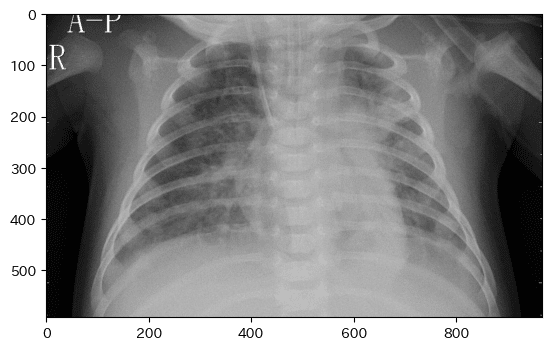
pneumonia
一枚しか表示していないので、信憑性には欠けますがこの画像に関しては正しく判定できているみたいです。
6.考察
今回のデータセットは画像を見てもわかりにくいと思いますが、ほとんどが小児の胸部画像になります。つまり成人の胸部画像とは異なるところが多いです。例えば肺野の大きさ、肋骨の太さ、心臓の大きさなどがあります。よって成人の胸部画像を予測させると正解率が低下する可能性が考えられます。
学習データおよびテストデータを今回の学習では300枚ずつにしました。この量は機械学習を行うに当たって少ないデータ量です。データ量を増加させていくことでさらなる精度向上につながると考えられます。
VGG16はカラー画像を前提に学習を行なっていますが、レントゲン画像はモノクロ画像です。今回は無理矢理カラー画像として学習させてしまいましたが、今の学習状況ではこれが精一杯なのでさらに勉強してこの問題をクリアしていきたいと考えています。
7.感想
医用画像と人工知能を上手に組み合わせることができました。このように成果物を作ることは良いアウトプットになると思いました。
今回のコードをベースとして、たくさんの医用画像とPythonを組み合わせたコードを書いていきたいと思います。放射線治療の際の正確な臓器分類を行うプログラム。患者の体格から瞬時に最適線量を算出するプログラムなどを作成したいと考えております。
※このブログはAidemy Premiumのカリキュラムの一環で、受講修了条件を満たすために公開しています
この記事が気に入ったらサポートをしてみませんか?
