
「夜型」の人が努力しても、決して「朝型」になれない -- とは限らない理由
(本記事はZhubo日本版オリジナル)
# はじめに
先日、WIREDさんがオリジナルで、yahooやlivedoorなどのメディアでも紹介された記事タイトル "「夜型」の人が努力しても、決して「朝型」になれない "に関して。
WIREDさんは個人的に一番と言っても過言ではないレベルに好きなメディアであり、このメディアを立ち上げる間接的なきっかけにもなりました。
いかに魅力的なタイトルで読者を引きつけるかは常に重要ですが、上記の記事タイトルに関しては、少し誤解を招く感が強いかなと思い、本記事で勝手ながら補足出来ればと思っています!
(タイトルの"なれない"の後ろに、せめて?マークの一つでも付けて欲しかったなという印象... 英語版記事が大元ですが.)
# ポイント
## 1. 「遺伝子によって決まる」のがどの程度なのか
夜型か朝型かに関して、遺伝子の影響があるということは先行研究でも示されてきましたし、今回の論文でも確かな事実としてより理解が深められています。が、"「夜型」の人が努力しても、決して「朝型」になれない"と結論づけることの一番の問題点は、効果量 (effect size) を無視した議論をしていること。より身近な言葉で言うと、遺伝子によって決まるのがどの程度なのか (例: 遺伝子の影響がほんの少しだけある、なのか、遺伝子で全て決まってしまう、なのか) を無視した議論であるという点。
元の論文*1 の図5を見ると分かる通り、今回同定された最も効果量(~=影響)の大きいゲノム変異でその効果量はlog odds ratio (LOR)~=0.2。これはどの程度かというと、「このゲノム変異を持っている人と持っていない人で比べて、持っている人の方が朝型である確率がe^0.2 = 1.22倍高い」という程度。
すなわち、「このゲノム変異を持っていない人1000人のうち100人が朝型だとして、ゲノム変異を持っている人1000人のうち120人くらいが朝型」という感じ。確かにこの変異を持っていることであなたが朝型である確率は上がります。しかし、この変異を持っていなかったからといって、"努力しても、決して「朝型」になれない "という印象は受けませんね。
(30億文字分の情報のうち1文字が書きかわるだけで確率が1.2倍にもなるのはかなりすごいことだと思いますが)
さらに、他の多くの変異はLORが0.01~0.02のオーダー (つまり確率が1.01~1.02倍になる程度)。
実際は論文で述べられた351個のゲノム変異の組み合わせ*2で朝型/夜型傾向がわかるわけですが、その程度はというと、本文及び元論文で述べられている通り"上位5パーセントの人は、最も少ない下位5パーセントの人と比べて、平均で25分早く眠りにつく"程度。 イメージとしては、私とあなたで比べて遺伝子によって睡眠開始時間に25分以上の差がある確率は2*0.05*0.05 で 0.5%。大体の場合は25分以下の差しかないという感じでしょうか*3。
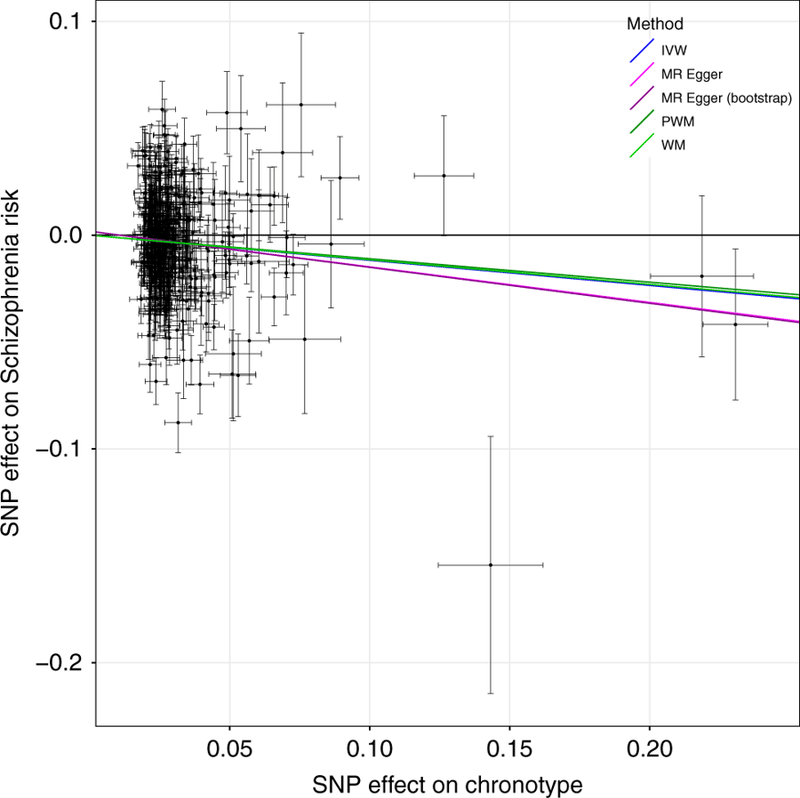
元論文Figure. 5。横軸が朝型傾向に対するeffect size。
(縦軸はSchizophrenia=統合失調症 に対するeffect size. こちらに関して詳細は本記事後半を参照)
## (2. 「遺伝子を持っているか」というより「ゲノム変異を持っているか」)
(これはどのメディアもやっている気がするし、言葉の使い方の問題なのでそこまでシビアではないかもしれませんが、)上で述べた、元記事中の「351個ある時計遺伝子のうち最も多くもっている上位5パーセントの人は、最も少ない下位5パーセントの人と比べて、平均で25分早く眠りにつく」という言い方も少し誤解のもととなるかなと感じます。
(1. で指摘したようにちゃんと定量的な議論をしているのはとても嬉しいのですが)
これは、GWASで同定されるゲノム領域(独立なゲノム領域 ; independent genomic loci)が351個同定されたこと*4と、「関連遺伝子が351個ある」ことはだいぶ異なるため。
ヒトのゲノムには遺伝子と呼ばれる部分と、そうでない部分(非コード領域; non coding regions)があり、遺伝子と呼ばれる部分はゲノム全体の2~3%と言われています。もちろんゲノム上で遺伝子領域は機能的に特に重要なのですが、遺伝子以外のゲノム領域(例: 遺伝子の働きを調節する制御領域; regulatory regions )でも重要な領域は多くあります。実際に今回同定されたゲノム変異も、多くは非コード領域にまたがっています。
また、今回の論文のように集団のゲノムデータを使って特定できる"独立なゲノム領域"が1つの遺伝子や1つの制御領域に丁度合致していることはまずありません*5。
これらの事実から、今回の論文中の"351個"の領域は複数の遺伝子にまたがっているもの / そもそも遺伝子にまたがっていないもの / 複数遺伝子を制御していると思われる制御領域にまたがっているもの などなど多岐にわたるため、「351個の遺伝子」という言い方はかなり不正確になってしまいます*6。
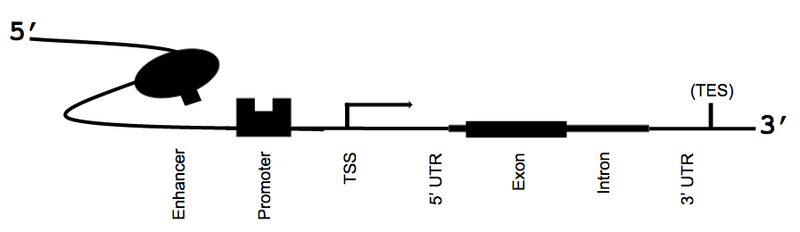
図: ゲノム上の様々な領域のイメージ。https://broad.io/gnomad_mnv より。
# 改めて論文のポイントおさらい
さて、これらの注意点を押さえた上で、改めて今回の論文のポイントをおさらいしてみましょう!
## 使われたデータ
今回の論文では UK Biobank と 23andme の2セットの集団ゲノムデータが使われています。
UK Biobank はイギリスで国家レベルで行われている巨大プロジェクトで、2006~2010年にリクルートされた約45万人分のイギリス人(白人)のゲノム変異データ+健康状態や生活習慣などのかなり密なアンケート結果が集まっていて、今回の研究に使われています。そのかつてないレベルのサンプル数(人数)と、密な周辺情報(phenotyping)から、近年の遺伝子研究で一躍注目を集めているデータセットです。
23andmeは世界最大の遺伝子検査会社の一つであり、ゲノム検査キットを購入して提出した約25万人分(ほぼ白人)のゲノム情報データが今回の研究に使われています。
## GWAS (ゲノムワイド関連解析) とは
当然上記の大規模データのアンケート項目には「あなたは朝型だと思いますか? / 平均起床時間はどれくらいですか?」などが含まれます。今回の論文ではこの大規模データ*7を利用して、「朝型を自負している人と夜型を自負している人で比べて、違いが出ているゲノム上の領域を調べよう」 ということをしました。
大規模データというのがミソで、例えば朝型の確率が約1.11倍になる(らしいけど確信は無い)変異Xがあったとして、

の状況ではXが本当に確率を1.11倍にする効果を持っているのかよく分からない/怪しい (偶然10人多かっただけかも?)ですが、
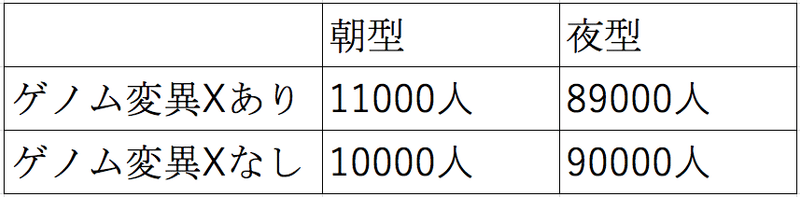
の状況なら、まあ信じていいか(偶然で1000人も多いなんてことは多分無いかな?)と思えますね。
データの量(sample size)が増えるほど、小さな効果に関してもある程度確信を持てるというわけです。
上の表を見つめて、変異Xに効果があるのかどうかを判定する、というのを統計学/数学的にやっているのがGWASというイメージです*8。
## 時計遺伝子 / 脳や網膜が重要 等々がなぜ言える?
今回の研究の意義は実は、新たに朝型に関連するゲノム領域がわかっただけでなく、それを遺伝子機能や生理学的機能とリンクさせた点にもあります。
このために彼らが行った作業のステップを、噛み砕いてみてみましょう。
a) 351個の"独立なゲノム領域"から、実際に"関連遺伝子"を定義する
上でも述べたように、同定された領域の中には、遺伝子や制御領域にまたがっていないものもたくさんあります。今回の手法(や一般的な手法)では、そのような場合においても「多分このゲノム変異はゲノム上である程度距離が近い遺伝子を何かしら制御しているんだな」という仮定を立てて*9、関連遺伝子を決めています。これにより、ただの"領域一覧"が、"遺伝子一覧" に変換されます (当然完璧な変換では無いですが、ある程度うまくいく変換であることは知られています)。

図: 元論文 Figure 1. 横軸がゲノムでの位置、縦軸が重要度 というイメージ
(x: position, y: significance. typical Manhattan plot from figure 1 of the original article)
b) 遺伝子ごとに、「この遺伝子は体のどの部分で特によく働いているのか」を調べ、特に関連する身体的部位を見つける
実は全ての遺伝子が身体の中で平等に働いているわけではなく、脳でだけ働いている遺伝子 / 目の網膜で特異的に働いている遺伝子 などなど遺伝子の活動*10には部位特異的なものも多くあります。これに注目し全ての遺伝子に関してどの部位でどのくらい働いていそうなのかを調べ上げたデータセットが世の中にはあります。今回の論文ではこのデータセット(GTEx)を用い、今回の関連遺伝子が全体的にどの部位で一番よく活動しているのかが調べられました。
これを使って、ただ漠然とした「これらの遺伝子が朝型に関連している」から、「これらの遺伝子が朝型に関連していて、特にこの部位で活動しているのが関連深いだろう」と一歩細かく生物学の理解を深めることができます。
(例: もしランダムに500個の遺伝子をとってきて活動部位を 調べてもわりと均等なはずだが、もし朝型傾向が脳に関連しているなら、朝型傾向に関連する遺伝子500個は全体的に他の部位より脳でよく活動しているだろう)
c) 遺伝子ごとの「関連する機能リスト」を調べ、特に関連する生物学的機能を見つける
ハエやマウス、ヒト細胞など種々の実験を通じた遺伝子の機能を調べる実験は日々世の中で行われています。その積み重ねとして、世の中には、「この機能に関連する遺伝子一覧」という具合にまとめられたデータがあります*11。
(例: 細胞間のシグナル伝達に関連する遺伝子一覧はこれ! など)
これらのデータを活用し、今回の関連遺伝子が全体的にどの生物学機能と関連しているのかが調べられました。
先程と同様、これを使って、ただ漠然とした「これらの遺伝子が朝型に関連している」から、「これらの遺伝子が朝型に関連していて、多分この生物学的機能が背景にあるんだろう」と一歩細かく生物学の理解を深めることができます。
以上のように、
a) ただの"領域一覧"から、"遺伝子一覧" に変換
b) 身体のこの部位(=脳/網膜)で活動している遺伝子が特に多い、そして
c) こういう機能(=生活リズム制御/光受容)を持った遺伝子が特に多い
という具合に、生物学的理解を深めていったわけです。
## 相関から因果へ(Genetic Correlation / Mendelian Randomization)
本文ではさらに、「夜型傾向とうつ病傾向は、遺伝的に相関しているかもしれない / 夜型傾向がうつ病の原因の一部となっているかもしれない」という方向にまで議論を進めています。ただの「関連している」から、これが遺伝的に相関している、さらに、片方が片方の原因となっている、と進めるのは当然簡単ではなく (例: アメリカ人は日本人より肥満率が高い、かつ日本人は遺伝子Xの作用により一般的に黒髪である、からといって、遺伝子Xが肥満の原因になっているとは限らない)、
そのような議論を進めるために主に2つの概念を導入しています。これを順に見ていきましょう。
i) genetic correlation
ゴリラの血液型は全てB型です。ゴリラは肌が黒いです。
つまりB型の人は肌が黒い、なんてことはありませんね。
単純に「特徴Aを持っている人は特徴Bを持っていることが多い」という議論から、「特徴Aと特徴Bは同じ遺伝子Xが影響している」なのか「特徴Aは遺伝子X、特徴Bは遺伝子Yが影響しているが、たまたま遺伝子XとYは共にあらわれがち」なのか(または、特徴Bはそもそも遺伝子が影響していないのか etc)は全然違いますね。genetic correlationはそれらのシナリオを分ける数値、というイメージです*12。
(ゴリラの例では、B型を決めるgenetic要素と肌の黒さを決めるgenetic要素は別なので、phenotypic correlation~=1でも、genetic correlation~=0。
特徴「肌の黒さ」は遺伝子X、特徴「血液型B型」は遺伝子Yが影響しているが、たまたま遺伝子XとYは共にゴリラという個体にあらわれがち、といったところ(?)*12.5 )

Photo by Greg Hume (wikipediaより)
論文ではgenetic correlationを調べることで、
- 朝型傾向とうつ病が遺伝的に(少しは)関連していること
- BMIや糖尿病とは遺伝的に関連していないこと
が示されました。
ii) mendelian randamization
「特徴Aと特徴Bは同じ遺伝子Xが影響している」という場合に関しても、
ア)「遺伝子Xが活動すると特徴Aが現れ、それによって特徴Bが現れる」なのか、
イ)「遺伝子Xが活動すると特徴Bが現れ、それによって特徴Aが現れる」なのか、はたまた
ウ)「遺伝子Xが活動すると特徴Aも特徴Bもそれぞれ個別に現れる」
なのかはまだわかりません。
(アの場合特徴Aが特徴Bの原因、イの場合逆、ウの場合どちらでもない)
これを(ある程度の仮定のもと)"見分ける"手法としてmendelian randamization (MR)という手法が知られています*13。
論文ではmendelian randamizationを用い、
-「夜型が原因でうつ病になる」的要素は微妙にあるらしい ことと
- 「うつ病が原因で夜型になる」ことはそんなに無いらしい こと
を示しました。

教科書でもおなじみのMendelさん. もちろんMendelian Randomizationの名前の由来になっています。
まとめると、genetic correlationを調べ、MRを駆使することで、論文では、
- 朝型傾向とうつ病が遺伝的に関連していること(+ BMIは糖尿病とは遺伝的に関連していないこと) 及び
- 夜型傾向が統合失調症(+もしかしたらうつ病)のリスクを上げる原因になること
が示されました。
(が、effect sizeに関してはまたも、一般的にはかなり小さいと感じる範囲*14なのではないでしょうか。)
# まとめ
UKBBをはじめとする大規模バイオバンクデータ (遺伝情報と健康情報が紐づけられたデータ)は本当に強力で、今まで知られていなかったゲノムの影響を次々と発見できるポテンシャルを秘めていて、研究者コミュニティは存分にワクワクしています。
だからこそ、上で述べたように、「この遺伝子が影響あり」という発見が次々と起こる中で、「じゃあこの遺伝子で運命が決まっているのか」と短絡的に判断せず、(特に睡眠や食事などに関連する身近な形質であるほど)「遺伝子の影響がどの程度*15」なのか、という定量的な部分に関しても自然と目を向けられる世の中になるといいなと思います(ついでに、欲を言えば「このゲノム領域が影響あり」と「この遺伝子が影響あり」も厳密には異なるので、遺伝子、という語の使い方も正確になってほしい)。
異分野の記事などを目にするときにはタイトルを鵜呑みにせず、中身をしっかり吟味しようという自身に対する戒めにもなりました。
WIREDさんが今後も刺激的でワクワクする記事を出してくれることを楽しみにしつつ、結びといたします。
(質問や誤り訂正etc大歓迎です!)
注/出典
1. https://www.nature.com/articles/s41467-018-08259-7
2. 正確にはさらに多くのゲノム変異。QC / genome wide significanceを通過したcommon SNPs からLD pruneをして得たindependent lociの数が351個なので。
3. かなりroughな値です。自分がtop/bottom 5 percentileに入る確率 * あなたがその逆の5 percentileに入る確率 で計算。もちろん平均なので実際は自分が丁度5 percentileのboundaryだったら差は25分より小さくなる / 自分がtop 0.1 percentileに入っていれば相手はもっと真ん中に近くても差は大きくなる などなどの限界あり。そして何より2.で述べたようにGWAS significantでないvariant and/or rare variantの寄与などなど。
4. 論文のsupplementaryを漁っても、筆者がどのようにindependent lociを定義したのかが述べられていないのですが、業界のconsensusはあるのでしょうか...。 rのthresholdを0.2にするか0.3にするか、cM単位で見るのか etcで微妙に変わる気がするので、詳しい人教えてくれたら助かります...。
5. 4で定義したindependent lociがfunctionalに定義したcoding gene / enhancer / promoter などとexactにmatchすることは無いよ、の意。もちろんcoding geneのsubsetになっているケースは今回の351 lociに関してもいくつかありますが。
6. 最初は日本語訳のミスだと思ったのですが、英語版元記事でも同様の表現(351 genes)が使われています。研究者がわかりやすくするために言ったのか、記者が書き間違えたのか、我々が気にしすぎなのか..
7. 日本では対応するのが Biobank Japan / GeneQuest あたりになると思いますが、さすがに欧米のサンプル数には勝てないです..
8. typicalなGWAS paperとして、Fine mappingもやっているのですが、LDの説明など込み入ってしまうので今回は省略
9. 今回彼らが使ったMAGMAもMAGENTAもDEPICTも(多分PASCAL)もcis window assumption (というかHi-Cとかをうまく組み込んでいるtoolあるのか?)。MAGENTAでは110kb upstream, 40kb downstreamをwindow sizeに設定したとのこと。
10. RNA expressionを"活動"とくだけた言い方をしています。もちろんprotein expressionや実際のactivityと完全イコールでは無いですが...
11. linkはGO term.今回の論文ではRNAi screeningなどいくつかのcircadian study結果を自前でまとめあげたらしいですが、まあイメージということでgo term。
12. (手法としてはいくつかありますが、ハートとしては、両者を決める遺伝的要素をそれぞれ同定して、それがどれくらい相関しているかをストレートに調べる、というイメージ。) 手法について議論しだすと闇が深そうなので深入りしません..。
12.5 (4/11追加). True genetic correlation / True phenotypic correlationのこと。もちろん実際にその数値を出すには、ゴリラだけでなく、B型以外かつ異なる肌の色を含む別の類人猿(e.g. ニホンザル)を含むheterogeneousなサンプル集団に対してのGWAS、かつ、PC やその他のcovariateによるcorrectionを行わない、というかなり不自然な状況設定が必要ですが... 。
13. MRも深入りしません笑
14. r_gは朝型とうつ病で0.16。MRに関してはunitが色々な変換を経ているらしくよく分からないが、significanceから推定するにおそらくそこまでcausal effect は強く無い(?) MRでのcausal effectの定量方法を知りたい...。
15. さらに言えば、effect sizeやPRSやheritability などなどがわかったところで、結局、populationの中でのgeneticsの寄与ではなく、個人における「私のこの形質において遺伝的要素で決まるのはnn%」というところまではなかなか行けないのでは....。この辺り詳しくなりたいですね...。
文: 王 青波
