
7年の時を経て再びNatureに: 次世代のがん細胞百科事典
がん細胞株に関する我々の理解は遥かに不足している
がん細胞株 (cancer cell line)*aは、がん研究に関わる人間皆にとって最も馴染み深い名詞である。がんの元凶を実験室で解明しようとするとき、或いは製薬市場でがんに対抗する武器を探求するとき、必ず欠かすことのできない第一歩が、がんを研究するモデルとしてのがん細胞を入手することである。幸いなことに、ときに病人のがん細胞は体内から分離してして以降も旺盛な生命力を持つ。プラスチックの培養皿に、培養液と結成を注いで、適切な温度と二酸化炭素濃度を保ってやれば、これらのがん細胞の新しい家となる。これら実験室で長く生存、分裂そして継代可能ながん細胞は総称してがん細胞株と呼ばれ、がんの原理の研究と新薬開発に関して必須で不可欠なツールとなっている。
細胞継代用の培養皿
1951年、最初のがん細胞株であるHeLaがアメリカのJohns Hopkins大学医学院の一人の子宮頸がん患者から分離され、後に世界中で万単位の実験室で培養されることになった。
今日までに、およそ4000種ものがん細胞株が数千ものがん患者から分離され、実験室中での培養が成功している。これら数千ものがん患者のがん細胞は実験室中で多く存在していて、簡単に用いることのできるツールとして研究者ががん研究をするのに使われている。これらの細胞系を深く分析することは、がんの発生及び発展機構を理解する機会を提供してくれる; 実験室中で薬物を細胞株に投与することで、どの新薬にがんを駆逐するポテンシャルがあるかを知ることができるのである。
科学研究の歴史上初のがん細胞株であるHeLa
がん細胞株の数ある貢献の一方で、ある種の重要な問題が少しずつ浮かび上がってきている:
- これらの細胞系の遺伝子(ゲノム)と分子的特徴は何であるか?
- これら細胞系にはどのようなゲノム 突然変異が起きていて、どのような(染色体や遺伝子の)コピー数の変化が生じているのだろうか?
- 個々の遺伝子のRNA発現量はどの程度なのか?
- 特定のがん細胞から得た情報は、臨床中ではどのような病態に最も関連しているのか?
実験室で数十年以上も使用されたこれらの細胞系に関して、いわゆる自身に対する”身体検査”の結果はまだ存在しないというわけである。
過去数十年間で、発表された数十万本ものがん研究に関連する論文、その全てにおいて少なからずがん細胞株が登場するということを忘れてはならない。すなわち、あらゆる抗がん新薬の研究開発の過程で、がん細胞株を用いた薬の効果と原理を調べる実験が行われている。もしこれらの細胞株の分子機構の詳細を知らないまま使用しても、得られる研究結果ががんに対して一般的にどの程度効果があるのかにも疑問が残ったままになる。ある薬剤はある種のがん細胞株を駆逐する能力があるかもしれないが、他の細胞株は駆逐できないかもしれない。すなわち、実際に患者さんを相手にしてどの程度の効果があるのかがわからないのである。
強力な連携で、がん細胞百科事典を作り上げる
ブロード研究所 (Broad Institute of MIT and Harvard) とノバルティス (Novartis Institutes for Biomedical Research)の科学者たちがこの問題に気づいた。そして学術界と産業界の強力な連携を形成して、がん研究の歴史上過去にない規模の計画が打ち立てられた: がん細胞株百科事典 (Cance Cell Line Encyclopedia = CCLE)である。 彼らの目標は、ハイスループット(=時間当たりの効率が良く大規模)な遺伝学的手法を用いて、よく使われるがん細胞株のゲノム、トランスクリプトーム、エピゲノムとその機能情報を解明し、さらにこれらの情報を無料のリソースとして一般に公開し、CCLEを全世界のがん研究者が使える百科事典とすることである。
2012年、CCLEの第一段階の成果のまとめが、堂々とNatureで発表された[1]。この論文では、BroadとNovartisの研究者たちがDNAマイクロアレイ(microarray)とパネルシークエンシング (panel sequencing)という手法を用い、947種類のがん細胞系において1600以上もの遺伝子の変異、コピー数の変化、そして発現量を分析した。同時に、彼らは479種類のがん細胞株において24種類の抗がん薬の効果を試し、更に、異なる細胞系における抗がん薬に対する感度(sensitivity)の違いと、ゲノム情報との対応を調べた。百科事典の建設が始めて形になった瞬間だった。

CCLEの第一段階を率いたのは、Broad研究所/Dana Farberがん研究所のLevi Garraway教授と、Norvartisのがん研究最高責任者(Global Head of Oncology)のWilliam Sellersであった。CCLE計画を進める過程で、彼らの身分にも興味深い変化があった。Levi氏は学術界から産業界に移り、2017年から製薬会社Eli Lillyの上級副総裁及びがん研究主任となった。William氏は産業界から学術界に移り、2016年にBroad研究所のコアメンバー及びHarvard大学医学院の教授となった。特筆すべきは、William氏は以前のNovartisの更に前にはHarvard大学医学院で主任研究員をしていて、Levi氏は彼の実験室のポスドクであったということだ。かつての師弟であった二人がCCLEで再びタッグを組み、さらには同じ時期に学術界と産業界の”スワップ”を経験したのは、とても不思議な縁である。

CCLEを率いた二人のキャリアパスの変化
NovartisからBroadに移ってから、Williamは継続してCCLEに打ち込んできた。打ち込むこと7年、彼らはついにこの巨大なプロジェクトを完成させ、先週 5/8にNatureに発表した[2]。

今回の最新のCCLE論文では、1079種類のがん細胞株のゲノム情報 (Microarray & Whole Exome Sequence = WES)、トランスクリプトーム (RNA-seq)、エピゲノム (DNAメチル化情報 / ヒストン修飾情報)情報が詳細に記録され、さらに、899種類のがん細胞株でのタンパク質発現と代謝情報が解読された。これらに加えて、それぞれの細胞株と関連する臨床情報、薬に対する感度、遺伝子相互の依存性 (CRISPRゲノム編集によって調べられる)も全てまとめてCCLEに含まれる。7年をかけて磨かれた剣のごとく、CCLEはがん細胞株の情報を調べるための”百科事典”にふさわしいものとなった。
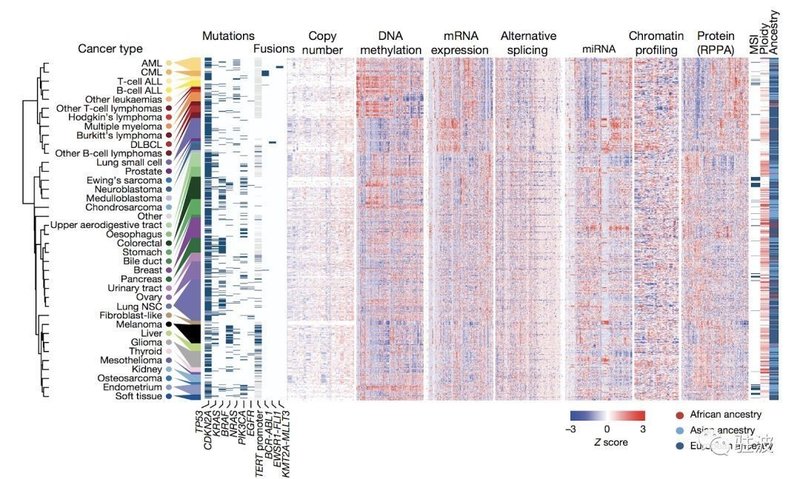
CCLEの情報の例 (論文より図を転載)
CCLEのデータは全てBroadの公開データベースに置かれていて、ダウンロード可能である。もしデータ処理が面倒であれば、depmapというポータル式のプラットフォームも用意されていて、特定の細胞系の名称を入力するだけで、全ての情報を余りなく閲覧することができる。
筆者が最近使用しているA375 melanoma細胞株を例にとってみると、depmapはこの細胞系の由来、形態、特に重要度が高い遺伝子 (preferentially essential gene)、ゲノム変異、ゲノム融合などの関連する情報が提供されていて、この細胞株を理解するのに必要な背景資料が全て含まれている。 (あとは実験を実際に手伝ってくれる機能が備われば完璧なのだが… 冗談。)

CCLE / DepMapを用いて細胞株の情報を調べる例
次の展望: がん研究データのオープンソース化
「我々はこれらのデータを公開することで、がん研究コミュニティが共同でがん研究の全体像を描き上げ、学術界と産業界の全員が抗がん新薬を開発するためにより一層質の高い努力をすることに貢献できればと思っている」とCCLEの責任者であるWilliam氏は語る。Broad研究所のWilliam氏の研究室に所属する人間として、筆者自身も、彼のがん研究データのオープンソース化に対する追求を強く感じた。
過去数年、データ公開はがん研究の領域では新しい潮流となっていた。アメリカ国立がん研究所(NCI)が率いるがんゲノム地図計画(The Cancer Genome Atlas = TCGA)が昨年完結し、一万以上の病人のがん細胞サンプルの種々の生物学的測定結果が全て公開され、全世界のがん研究者と臨床医ががんのゲノム編成の詳細を理解するのに役立てられている。アメリカがん研究協会(AACR)の主導により、アメリカで数十もの主要な医療機関がGENIE計画と呼ばれるゲノム情報集積計画に参加し、今日に至るまで3万人以上ものがん患者のゲノムが読まれ、それらの結果も全て公開されている。
ゲノム以外にも、がんを治療する小分子新薬も”がん研究のオープンソース化”の潮流の一部分になっている。2011年、当時Dana Farberがん研究所に所属していたJay Bradner博士のグループが、BRD4タンパク質を抑制する小分子新薬であるJQ1を開発し、これが種々のがんに有効であることが示された。時間と労力をかけて新薬を開発した後には、多くの研究室が行うのは、できるだけ多くの関連論文を発表し、ライバルたちに対する優位を確立することである。しかし意外なことに、Jay氏は初めにJQ1の構造に関する論文を発表した[3]後に、合成が完了したJQ1を全世界でBRD4タンパク質を研究したがっている研究室に無料で発送することを進め、40ものアメリカの研究室と、30ものヨーロッパの研究室がこれを受け取った。
そしてこれに続くかのように、Novartisは2016年にSHP2タンパク質を抑制する小分子新薬であるSHP099を開発し、抗がんに有効な脱リン酸化酵素(phosephotase)抑制剤がないという従来の”ジンクス”*bを打ち破った。一般には製薬会社はこのように新規制の高い新薬を開発した際には、公開する情報を限定し利潤を上げる方向に走る。しかし今回のNovartisはそのような先例を破り、SHP099の構造、セレクションの過程及び研究開発の実験設計などを全てNatureに発表した[4]。これ以降、SHP2はがん研究における雨後の筍のごとく、数多くの興味深いメカニズムや治療戦略の発見につながった。さらに注目すべきことに、この論文の責任著者はまたもWilliam Sellersであった。

Jay氏のted talkの様子
今こそがん研究を行うのにベストな時代である。Broad研究所、NCI、AACRなどの大型機構がたゆまぬ努力を重ね、がん研究者が触れられるビッグデータが日々蓄積されている。以前のようにデータ自体を探すことの壁に時間と労力をかける必要はなく、がんの未知のメカニズムの解明それ自体により多くの時間を割くことができる。学術界/産業界関係なく、これらの膨大ながん関連のデータは、我々をがんを克服するという目標に一歩近づけてくれる。
今こそがん研究を行うのに最もチャレンジングな時代であるとも言える。がん研究者に言わせれば、十年前のように”強みが一つあれば食っていける”時代、自分たちの独自のデータを論文に発表するだけの時代はすでに過ぎ去ったということ。同時に、日々蓄積する公開データを有効利用するためには、必死に実験室で実験を重ねるだけでは到底足りず、データサイエンス/プログラミング的アプローチや訓練がより重要になってくる。ビッグデータの背後に潜む意味を理解し、膨大なデータの中から生物学的に意味のある仮説を見つけ出すことが、新時代のがん研究者に必要不可欠なスキルとなってきている。
参考文献:
1. https://www.nature.com/articles/nature11003
2. https://www.nature.com/articles/s41586-019-1186-3
3. https://www.nature.com/articles/nature09504
4. https://www.nature.com/articles/nature18621
日本語版注:
*a 癌vsガンvsがんでニュアンスの違いは生じますが、本記事ではひらがなに統一しています
*b 訳に自信なし.. biologicalに間違っている主張かもしれません.. 訂正etc welcomeです
文 / 常亮
校閲 / 袁博,方睿
植字 / 方睿
日本語訳 / 王青波
