
実験!岩波データサイエンス1のベイズモデリングをPyMC Ver.5で⑧状態空間モデル:野生の鳥のカウントデータ
「時系列・空間データのモデリング」章
テキスト「時系列・空間データのモデリング」の執筆者
伊東宏樹 先生
この記事は、テキストの「時系列・空間データのモデリング」章の「野生の鳥のカウントデータを用いた状態空間モデル」の実践を取り扱います。
観測値が正の離散値であること、カウントされない隠れた個体を予測することなど、魅惑のメニューなのです!
たのしくPyMCモデリングを進めましょう!
この章を執筆された伊東先生は「たのしいベイズモデリング2」第5章を執筆されています。
樹木の直径と高さの数理モデルをベイズに取り込んでいます!
こちらもお楽しみくださいませ。
(ご参考)書籍「たのしいベイズモデリング2」
(ご参考)たのしいベイズモデリング2のPyMC化ブログ
はじめに
岩波データサイエンスVol.1の紹介
この記事は書籍「岩波データサイエンス vol.1」(岩波書店、以下「テキスト」と呼びます)の特集記事「ベイズ推論とMCMCのフリーソフト」のベイズモデルを用いて、PyMC Ver.5で「実験的」に実装する様子を描いた統計ドキュメンタリーです。
テキストは、2015年10月に発売され、ベイズモデリングの様々なソフトウェアを用いたモデリング事例を多数掲載し、ベイズモデリングの楽しさを紹介する素晴らしい書籍です。
入門的なモデルから2次階差を取り扱う空間モデルまで、幅広い難易度のモデルを満喫できます!
このシリーズは、テキストのベイズモデルをPyMC Ver.5に書き換えて実践します。
引用表記
この記事は、出典に記載の書籍に掲載された文章及びコードを引用し、適宜、掲載文章とコードを改変して書いています。
【出典】
「岩波データサイエンス vol.1」
第9刷、編者 岩波データサイエンス刊行委員会 岩波書店
記事中のイラストは、「かわいいフリー素材集いらすとや」さんのイラストをお借りしています。
ありがとうございます!
PyMC環境の準備
Anacondaを用いる環境構築とGoogle ColaboratoryでPyMCを動かす方法について、次の記事にまとめています。
「PyMCを動かすまでの準備」章をご覧ください。
PythonとPyMC
テキストで利用するツールは、R、BUGS、JAGS です。
この記事では、PythonとPyMCを用いたコードに変換してベイズモデリングを実践いたします。
イントロ
1. モデルのイメージ
野生の鳥の数を経時的にカウントする場合を想定します。
必ずしも周囲の全ての鳥をカウントできるとは限りません。
隠れている鳥を見逃してしまうことはあるでしょう。

今回取り組むベイズモデリングでは、カウントされなかった鳥を含めた「真の個体数」を推論します!
観測できた「発見個体数」は、真の個体数のうち「発見確率」で見つけられた数だ、と考えるのです。
数式で表現すると次のようになります。
$$
\begin{align*}
N^{\text{obs}}_{ti} &\sim \text{Binomial}\ (N_t,\ p) \\
N_t &\sim \text{Poisson}\ (\lambda_t) \\
\log \lambda_t &\sim \text{Normal}\ (\log \lambda_{t-1},\ \sigma^2) \\
\end{align*}
$$
最初の2行が観測モデル、最後の1行がシステムモデルです。
観測モデルは、観測値である発見個体数$${N^{\text{obs}}_{ti}}$$が真の個体数$${N_t}$$、発見確率$${p}$$の二項分布に従うとしています。
そして、真の個体数$${N_t}$$が期待値$${\lambda_t}$$のポアソン分布に従うと仮定しています。
観測は、50回実施し、1回の観測で5回のカウントを実施します。
発見個体数$${N^{\text{obs}}_{ti}}$$は、$${t}$$回目の観測時の$${i}$$回目($${i=1, \cdots, 5}$$)のカウントです。
システムモデルは、対数スケールに変換した真の個体数の期待値$${\lambda_t}$$が正規分布に従ってランダムウォークすると仮定します。
2. インポート
「時系列・空間データのモデリング」章で利用するパッケージをインポートします。
### インポート
# ユーティリティ
import pickle
# 数値・確率計算
import pandas as pd
import numpy as np
# PyMC
import pymc as pm
import pytensor.tensor as pt
import arviz as az
# WEBアクセス
import requests
# Rdata読み込み
import pyreadr
# 描画
import matplotlib.pyplot as plt
import seaborn as sns
plt.rcParams['font.family'] = 'Meiryo'
# ワーニング表示の抑制
import warnings
warnings.simplefilter('ignore')3. データの作成
観測データはリアルな観測値ではなく、プログラムで生成する架空データです。
Rコードの方法に準拠してデータを作成し、pandasのデータフレームを作成します。
ただし、乱数生成がテキストと異なるため、データ内容やベイズ分析内容がテキストと相違することになります。
### データの作成 テキストのRコードを参考に
## 初期値設定
# 乱数生成器の設定
rng = np.random.default_rng(seed=31415)
# データ生成パラメータ
nt = 50 # 観測回数
nr = 5 # 1回の観測時のカウント回数
p_obs = 0.6 # 発見確率
## 真の個体数の期待値(対数)logλ
# numpy配列の準備
log_lambda = np.zeros(nt)
# 初期値
log_lambda[0] = np.log(30)
# t期のlogλ=t-1期のlogλ+正規分布乱数normal(0, 0.1)
for t in range(1, nt):
log_lambda[t] = log_lambda[t-1] + rng.normal(loc=0, scale=0.1)
## 真の個体数の生成
N_true = rng.poisson(lam=np.exp(log_lambda))
## 発見個体数の生成
N_obs = np.array(
[rng.binomial(n=N_true[t], p=p_obs, size=nr) for t in range(nt)])【実行結果】なし
4. データの外観の確認
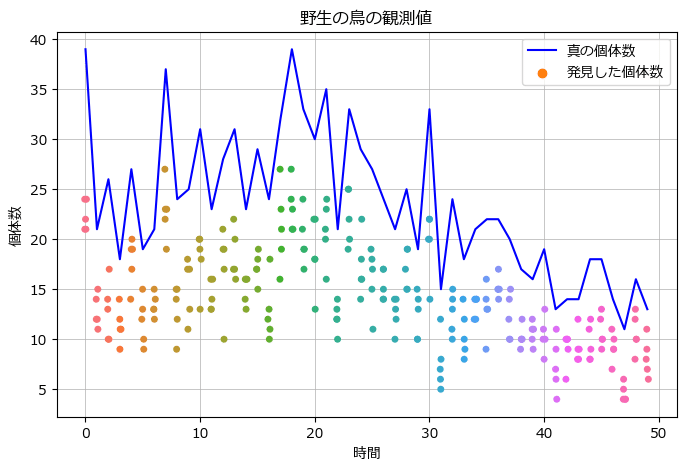
発見個体数と真の個体数(実際には分からない値)の時系列の折れ線グラフを描画します。
テキストの図6の予測前に相当します。
### 真の個体数N、発見個体数Nobsの描画
fig, ax = plt.subplots(figsize=(8, 5))
# 真の個体数の折れ線グラフ
ax.plot(range(nt), N_true, color='blue', label='真の個体数')
# 発見個体数のストリッププロット
sns.stripplot(N_obs.T, ax=ax)
ax.scatter(x=None, y=None, label='発見した個体数')
# 修飾
ax.set(xticks=range(0, nt+1, 10), xticklabels=range(0, nt+1, 10), xlabel='時間',
ylabel='個体数', title='野生の鳥の観測値')
ax.legend()
ax.grid(lw=0.5);【実行結果】
1回の観測につき5回カウントしているので、時間ごとに発見個体数の点が5つプロットされています。
真の個体数を示す折れ線グラフはランダムウォークで生成しています。
真の個体数と比べて発見個体数は少なく、ばらついている感じがします。
5. データの前処理
ベイズモデルで使いやすいようにデータを加工(発見個体数のフラット化)します。
## データ前処理
# 発見個体数を平坦化:shape=(50, 5)からshape=250
N_obs_flat = N_obs.flatten()
print('N_obs_flat.shape: ', N_obs_flat.shape)
print('N_obs_flat[:10] : ', N_obs_flat[:10], '\n'+'-'*50)
# 平坦化した発見個体数の観測回のインデックス
N_obs_idx = np.repeat(np.arange(nt), nr)
print('N_obs_idx.shape : ', N_obs_idx.shape)
print('N_obs_idx[:10] : ', N_obs_idx[:10])【実行結果】

では、PyMCモデリングに進みます。
ベイズモデル
1. モデル数式
前章のモデルのイメージの数式をご覧ください。
2. モデルの定義
数式モデルに準拠してPyMCのモデルを定義しましょう。
システムモデルにはランダムウォーク「pm.GaussianRandomWalk()」を利用します。
### モデルの定義
with pm.Model() as model4:
### データ関連定義
## coordの定義
model4.add_coord('data', values=range(1, len(N_obs_flat)+1), mutable=True)
model4.add_coord('obs', values=range(1, len(N_obs)+1), mutable=True)
## dataの定義
# 平坦化した発見個体数
y = pm.ConstantData('y', value=N_obs_flat, dims='data')
# 平坦化した発見個体数の観測回のインデックス
obsIdx = pm.ConstantData('obsIdx', value=N_obs_idx, dims='data')
### 事前分布
# システムモデルの標準偏差パラメータ
sigma = pm.Uniform('sigma', lower=0, upper=100)
# 観測モデルの二項分布の確率パラメータ
p = pm.Uniform('p', lower=0, upper=1)
### システムモデル:ランダムウォーク
init_dist = pm.Normal.dist(mu=0, sigma=100)
logLam = pm.GaussianRandomWalk('logLam', mu=0, sigma=sigma,
init_dist=init_dist, dims='obs')
### 尤度:観測モデル
# logLamの指数をとる
lam = pm.Deterministic('lam', pt.exp(logLam), dims='obs')
# 真の個体数Nはポアソン分布に従う
N = pm.Poisson('N', mu=lam, dims='obs')
# 観測値=発見個体数は二項分布に従う
Y = pm.Binomial('Y', n=N[obsIdx], p=p, observed=y, dims='data')モデルの内容を表示・可視化してみましょう。
### モデルの表示
model4【実行結果】

続いてモデルを可視化します。
### モデルの可視化
pm.model_to_graphviz(model4)【実行結果】

真の個体数 N は、ランダムウォークする logLam($${\log \lambda}$$、真の個体数の期待値の対数)をパラメータ(指数化後)に用いるポアソン分布で推論されます。
真の個体数 N と発見確率 p(一様分布)は発見個体数の従う二項分布のパラメータになります。
認知できない真の個体数を推論できてしまうベイズって凄いですね!
3. MCMCの実行と収束の確認
■ MCMC
マルコフ連鎖=chainsを4本、バーンイン期間=tuneを1000、利用するサンプル=drawを1chainあたり1000に設定して、合計4000個の事後分布からのサンプリングデータを生成します。テキストの指定数とは異なっています。
事前分布に離散値を含むので、PyMC標準のサンプラーを利用します。
また、MCMCエラーを回避するため、パラメータの初期値 initvals を設定しました。
### 事後分布からのサンプリング 2分40秒
# パラメータの初期値の設定
initvals = {'p_interval__': np.array(0.5), 'N': np.repeat(100, 50)}
# MCMC
with model4:
idata4 = pm.sample(draws=1000, tune=5000, chains=4, random_seed=123,
target_accept=0.9, initvals=initvals)【実行結果】
処理時間は2分40秒です。

■ $${\hat{R}}$$で収束確認
収束の確認をします。
指標$${\hat{R}}$$(あーるはっと)の値が$${1.1}$$以下のとき、収束したこととします。
次のコードでは$${\hat{R}>1.1}$$のパラメータの個数をカウントします。
個数が0ならば、$${\hat{R} \leq1.1}$$なので収束したとみなします。
### r_hat>1.1の確認
# 設定
idata_in = idata4 # idata名
threshold = 1.1 # しきい値
# しきい値を超えるR_hatの個数を表示
print((az.rhat(idata_in) > threshold).sum())【実行結果】
$${\hat{R}>1.1}$$のパラメータは0個でした。
$${\hat{R} \leq1.1}$$なので収束したとみなします。

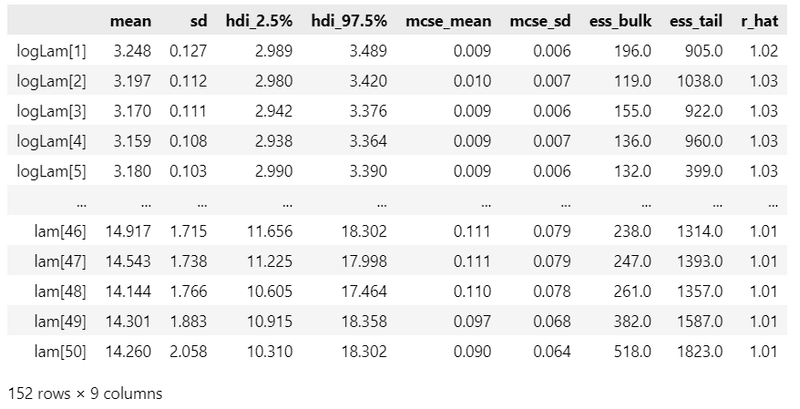
■ $${\hat{R}}$$の値把握と事後統計量の表示
### 事後統計量の表示
pm.summary(idata4, hdi_prob=0.95)【実行結果】
■ トレースプロットで収束確認
トレースプロットを描画します。
こちらの図でも収束の確認を行えます。
### トレースプロットの描画
pm.plot_trace(idata4, compact=True)
plt.tight_layout();【実行結果】
左プロットの線の重なり具合、右プロットの偏りのない乱雑さより、収束している感じがします。
と言いたいところですが、p は4本のマルコフ連鎖 chain がバラけていて、なんだか危うい形状です。

4. 分析
真の個体数の期待値$${\lambda_t}$$の事後分布を描画しましょう。
テキストの図6に相当します。
### 真の個体数N、発見個体数N_obs、真の個体数の期待値λの描画 ※図6に相当
## 描画用データの作成
# λの取り出し
lambda_samples = idata4.posterior.lam.stack(sample=('chain', 'draw')).data
# 真の個体数の期待値λの事後平均
lambda_means = lambda_samples.mean(axis=1)
# 真の個体数の期待値λの95%HDI
lambda_95hdi = az.hdi(lambda_samples.T).T
## 描画
fig, ax = plt.subplots(figsize=(8, 5))
# 真の個体数の折れ線グラフ
ax.plot(range(nt), N_true, color='gray', lw=1, label='真の個体数')
# 発見個体数のストリッププロット
sns.stripplot(N_obs.T, color='tab:blue', size=4, ax=ax)
# 凡例ダミー
ax.scatter(x=None, y=None, color='tab:blue', s=10, label='発見した個体数')
# 真の個体数の期待値λの事後平均の折れ線グラフ
ax.plot(range(nt), lambda_means, color='tab:red', lw=3,
label='真の個体数の期待値 $\lambda$')
# 真の個体数の期待値λの95%HDI区間の塗りつぶし
ax.fill_between(range(nt), lambda_95hdi[0], lambda_95hdi[1],
color='tomato', alpha=0.2, label='95%HDI')
# 修飾
ax.set(xticks=range(0, nt+1, 10), xticklabels=range(0, nt+1, 10), xlabel='時間',
ylabel='個体数', title='野生の鳥の観測値、真の個体の推定値')
ax.legend()
ax.grid(lw=0.5);【実行結果】
赤い線が$${\lambda_t}$$の事後分布の平均値、ピンクの面が$${\lambda_t}$$の95%HDI区間です。
真の個体数(灰色の線)と期待値$${\lambda_t}$$の事後分布を比較すると、真の個体数の飛び抜けた箇所(0回目、18回目、30回目等)を除けば、95%HDI区間に真の個体数が含まれており、まずまずの推論だと思います。

テキスト49ページの発見確率$${p}$$の事後分布の記載を真似してみます。
### 発見率pの事後統計量の表示
display(pm.summary(idata4, hdi_prob=0.95, var_names=['p'], kind='stats'))
pm.plot_posterior(idata4, hdi_prob=0.95, var_names=['p'], figsize=(4, 3),
round_to=3);【実行結果】
発見確率$${p}$$の事後平均は$${0.63}$$、95%HDIは$${0.57}$$~$${0.68}$$と推定されました。

5. 推論データの保存
pickle で MCMCサンプリングデータ idata4 を保存できます。
### 推論データの保存
file = r'idata4_ch3.pkl'
with open(file, 'wb') as f:
pickle.dump(idata4, f)次のコードで保存したファイルを読み込みできます。
### 推論データの読み込み
file = r'idata4_ch3.pkl'
with open(file, 'rb') as f:
idata4_ch3_load = pickle.load(f)ベイズ記事は以上です。
次回予告
「時系列・空間データのモデリング」章
空間自己回帰モデル「1次元の個体数カウントデータ」
結び
今回の真の個体数の期待値$${\lambda}$$のような決して観測されない変数(潜在変数?)を、確率分布を駆使したモデリングで推論できてしまうベイズって凄いですね!
ポアソン分布に従う変数が二項分布の試行回数パラメータにつながるといった分布の組み合わせについても、とても勉強になりました。
おわり
シリーズの記事
次の記事
前の記事
目次
ブログの紹介
note で7つのシリーズ記事を書いています。
ぜひ覗いていってくださいね!
1.のんびり統計
統計検定2級の問題集を手がかりにして、確率・統計をざっくり掘り下げるブログです。
雑談感覚で大丈夫です。ぜひ覗いていってくださいね。
統計検定2級公式問題集CBT対応版に対応しています。
Python、EXCELのサンプルコードの配布もあります。
2.実験!たのしいベイズモデリング1&2をPyMC Ver.5で
書籍「たのしいベイズモデリング」・「たのしいベイズモデリング2」の心理学研究に用いられたベイズモデルを PyMC Ver.5で描いて分析します。
この書籍をはじめ、多くのベイズモデルは R言語+Stanで書かれています。
PyMCの可能性を探り出し、手軽にベイズモデリングを実践できるように努めます。
身近なテーマ、イメージしやすいテーマですので、ぜひぜひPyMCで動かして、一緒に楽しみましょう!
3.実験!岩波データサイエンス1のベイズモデリングをPyMC Ver.5で
書籍「実験!岩波データサイエンスvol.1」の4人のベイジアンによるベイズモデルを PyMC Ver.5で描いて分析します。
この書籍はベイズプログラミングのイロハをざっくりと学ぶことができる良書です。
楽しくPyMCモデルを動かして、ベイズと仲良しになれた気がします。
みなさんもぜひぜひPyMCで動かして、一緒に遊んで学びましょう!
4.楽しい写経 ベイズ・Python等
ベイズ、Python、その他の「書籍の写経活動」の成果をブログにします。
主にPythonへの翻訳に取り組んでいます。
写経に取り組むお仲間さんのサンプルコードになれば幸いです🍀
5.RとStanではじめる心理学のための時系列分析入門 を PythonとPyMC Ver.5 で
書籍「RとStanではじめる心理学のための時系列分析入門」の時系列分析をPythonとPyMC Ver.5 で実践します。
この書籍には時系列分析のテーマが盛りだくさん!
時系列分析の懐の深さを実感いたしました。
大好きなPythonで楽しく時系列分析を学びます。
6.データサイエンスっぽいことを綴る
統計、データ分析、AI、機械学習、Pythonのコラムを不定期に綴っています。
統計・データサイエンス書籍にまつわる記事が多いです。
「統計」「Python」「数学とPython」「R」のシリーズが生まれています。
7.Python機械学習プログラミング実践記
書籍「Python機械学習プログラミング PyTorch & scikit-learn編」を学んだときのさまざまな思いを記事にしました。
この書籍は、scikit-learnとPyTorchの教科書です。
よかったらぜひ、お試しくださいませ。
最後までお読みいただきまして、ありがとうございました。
この記事が気に入ったらサポートをしてみませんか?
