Sタンパク質のN末端ドメイン変異は、SARS-CoV-2株のエピトープ認識に構造的に関与(2021年10月)
N-terminal domain mutations of the spike protein are structurally implicated in epitope recognition in emerging SARS-CoV-2 strains
スパイクタンパク質のN末端ドメイン変異は、新興SARS-CoV-2株のエピトープ認識に構造的に関与している。
Abstract
過去 2 年間、世界は重症急性呼吸器症候群コロナウイルス 2 (SARS-CoV-2) による世界的パンデミックに見舞われてきました。
SARS-CoV-2 ゲノムの獲得変異がウイルスの感染性や免疫原性に影響を及ぼし、元の武漢株に比べて感染力の高い新しい株が数多く生まれています。
SARS-CoV-2 スパイクタンパク質の受容体結合ドメイン (RBD) の変異は、この文脈で広く研究されてきました。
しかし、RBD に隣接する N 末端ドメイン (NTD) 内の変異と欠失についてはあまり研究されていません。
これらの多くは、特定のβシート結合ループ内に見つかっています。 SARS-CoV-2のβシート結合ループは、SARS-CoVや他の関連βコロナウイルスと比較して、驚くほど長いです。
ここでは、これらのループ内に変異や欠失を持つ新しい株の構造的および疫学的研究を実施します。
我々は、NTD ループを安定化させ、回復期血漿からのさまざまな中和抗体による認識に不可欠な重要なエピトープを形成する短距離および長距離の相互作用を特定しました。
これらのループで見つかったさまざまな変異/欠失のうち、Ala 67 および Asp 80 変異、および His 69/Val 70 および Tyr 144 欠失が、さまざまな急速に広がる株で特定されています。
同様に、アミノ酸 241~243 および 246~252 の欠失は、伝染性の高い株の NTD ループのネットワークに影響を及ぼすことがわかっています。
私たちの構造的発見は、これらの変異/欠失がエピトープ構造を変化させ、スパイクタンパク質の NTD 領域の免疫反応性に影響を及ぼす役割についての洞察を提供します。
1. Introduction
コロナウイルス (科: コロナウイルス科) は、異常に大きなゲノム (約 30 Kb) を持つ、ポジティブまたはプラスセンスの RNA ウイルスです。
コロナウイルスは 4 つのサブタイプ (α、β、γ、δ) に分類され、鳥類から哺乳類までさまざまな宿主に感染し、重篤な罹患率と死亡率を引き起こす可能性があります。
過去 20 年間、人への広範な感染ケースが繰り返し発生したため、厳しい監視の対象となってきました。
最も一般的なヒトコロナウイルスは、サブタイプαである229E・ NL63、サブタイプ βであるHKU1・OC43 です。世界中の人々の気道に感染し、風邪の症状を引き起こします。
最近では、人獣共通感染症 (動物) 起源の β コロナウイルス株が、人命に対する深刻な脅威として浮上しています:MERS-CoV (中東呼吸器症候群、MERS)、SARS-CoV (重症急性呼吸器症候群、SARS)、および SARS-CoV-2 (2019 年コロナウイルス感染症、COVID-19)。
MERS と SARS-CoV の全体的な死亡率の方がはるかに高いですが、中国の武漢省で発生したSARS-CoV-2は、世界中で2億550万人以上が感染し、430万人近くが死亡しました。
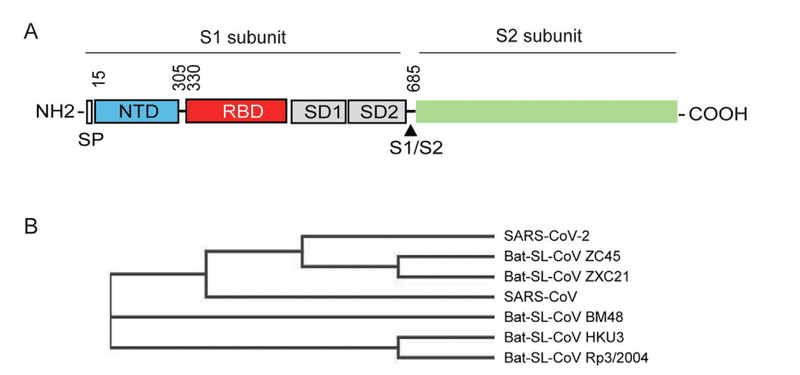
SARS-CoV と SARS-CoV-2 はほぼ 80% の配列同一性を共有しており、どちらも S (スパイク) タンパク質とアンジオテンシン変換酵素 2 (ACE2) の相互作用を通じて宿主細胞に侵入します。
S タンパク質は 2 つの SARS 株間で 80% のタンパク質類似性を共有し、同様の親和性で ACE2 に結合します。
RNA依存性RNAポリメラーゼ(RdRP)はエラー率が高い傾向にあるため、プラス鎖RNAウイルス内の遺伝的多様性と進化は主にRNA複製の忠実度の低さによって引き起こされることが広く受け入れられています。
複製が主に RdRp に依存する他の RNA ウイルスとは異なり、コロナウイルスでは、非構造タンパク質 (nsps) には、処理能力因子 (nsp7-8)、ヘリカーゼ (nsp13)、一本鎖結合タンパク質 (nsp9)、プルーフリーディング エキソヌクレアーゼ (nsp14) およびその他の補因子 (例: nsp 10、nsp16) 、RdRp(校正活性を持つ)と複製複合体を形成します。ウイルスRdRpによってエラーを修正します。
その結果、コロナウイルスは他の RNA ウイルスと比較して変異率が 10 倍低いという特徴があります。
RNA 複製における校正活動にもかかわらず、SARS-CoV の推定変異率は 4x10^-4 ヌクレオチド置換/部位/年ですが、SARS-CoV-2 では 1.12 × 10^-3 変異/部位/年です。
最近の研究により、SARS-CoV-2 スパイクタンパク質 NTD には、宿主の適応免疫応答によって生成される中和抗体によって認識されるエピトープが含まれるという強力な証拠が示されました。
さらに、スパイクタンパク質のこの領域は宿主細胞表面の接着に動的に関与し、細胞の糖環境のグリカン基との相互作用を媒介していることが提案されています。
したがって、SARS-CoV-2 NTD の遺伝的変異を追跡することは、潜在的に免疫回避能力が高い、または感染力が高い新興株を監視するために重要です。
この方向に向けて、我々はβコロナウイルスにおけるNTDの進化を調査しました。
SARS-CoV と SARS-CoV-2 のスパイク構造を比較し、利用可能な SARS-CoV-2 の変異データを分析することにより、私たちは、SARS-CoV-2クレード内で進化的に分岐した特定のNTDループ要素が高い突然変異率を示し、遺伝的変異を引き起こすことを発見しました。
興味深いことに、これらのループの特定の変異と欠失は NTD 構造の変化に関連しており、スパイクタンパク質の NTD 領域を標的とする多くの異なる抗体に共通するエピトープ領域に影響を与えます。
これらの変異体に対応する疫学データから、特定の NTD 変異および欠失が、COVID-19 パンデミックの直近の波にわたって積極的に選択されてきたことが明らかになりました。
3. Results
3.1 特定のループ領域がβコロナウイルスのスパイクNTDタンパク質の進化的分岐を促進する
中国の武漢患者からのSARS-CoV-2ゲノムの配列決定と、代表的なβコロナウイルスゲノム(属:ベータコロナウイルス)の系統解析により、ベータコロナウイルス属のサルベコウイルス亜属が3つの明確なクレードに分類できることが明らかになりました。
クレード 1 は、ブルガリア (BM-48) とケニア (BtKY72) の Rhinolophus sp. に由来する 2 つの SARS-CoV 関連株で構成されています。
ヒト SARS-CoV-2 配列と中国東部の 2 つのコウモリ SARS 様株 (bat-SL-CoVZC45 および Bat-SL-CoVZXC21) はクレード 2 を形成します。一方、ヒト由来のSARS-CoV株と中国南西部由来のコウモリSARS様コロナウイルスはクレード3を形成します。
β コロナウイルス メンバーのスパイク NTD のタンパク質のアラインメントが同様の系統学的プロファイルを示すかどうかを調べるために、代表的な β コロナウイルス株のスパイク NTD 間で比較配列分析を実行しました。
この方向で、SARS-CoV-2 スパイクタンパク質 (図 1A) の残基 15 ~ 305 が SARSとBat-SARS 様コロナウイルス株クレード 1 (BM48)・クレード 2 (BM48ZC45、ZXC21)・クレード 3 (HKU3、Rp3)のメンバー の NTD 配列に対して整列されました。
図 1B が示すように、これらの配列のタンパク質アラインメントにより、SARS-CoV-2 と関連するコウモリ由来株 ZC45 および ZXC21 が一緒にクラスター化していることが示されました。一方、SARS-CoVとコウモリのSARS様配列は別々にクラスター化しており、ゲノム配列で得られたものと同様の系統発生パターンを示しています。
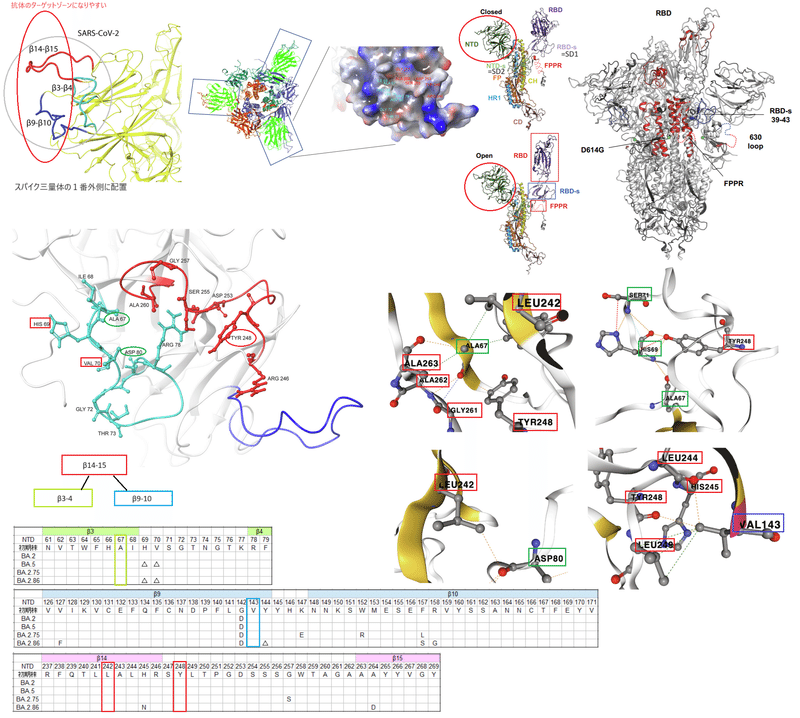
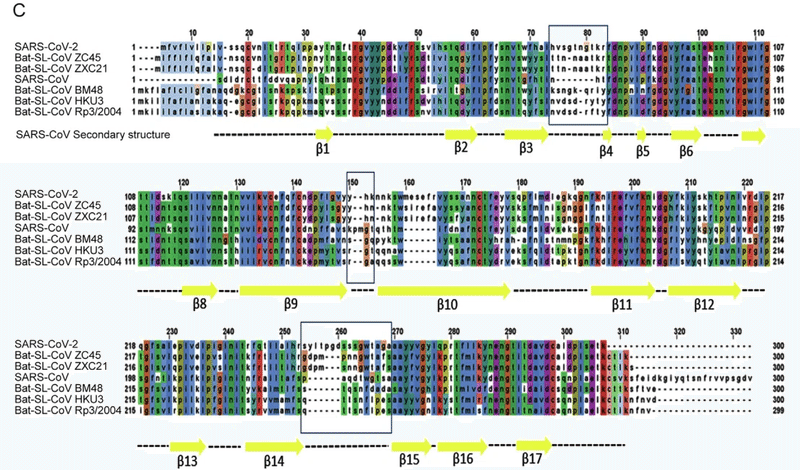
SARS-CoV の二次構造要素 (PDB: 5X4S 構造から特定) をこの配列アラインメントに統合すると、全体として、NTD が大部分の β シートおよびループ内でよく保存されていることを観察しました。ただし、β3-β4、β9-β10、および β14-β15 シートを分離するループは除きます (図 1C)。
より具体的には、SARS-CoV-2 の β3-β4 および β14-β15 ループは、SARS-CoV と比較して長い長さを示します。
拡張されたβ3-β4ループもすべてのコウモリSARS-CoVメンバーの間で共有されていますが、β14-β15 ループの伸長は SARS-Cov-2 のみに限られています。
SARS-CoV-2 と SARS-CoV の間の NTD 分岐領域の構造の違いを特定するために、SARS-CoV-2 スパイクタンパク質の低温電子顕微鏡 (Cryo-EM) 構造を SARS-CoV NTD 結晶構造と比較しました。
この構造比較により、SARS-CoV-2のβ3-β4、β9-β10、およびβ14-β15ループの長さがSARS-CoVと比較して進化的に延長されたことが明らかになりました(図1D)。
3.2 β3-β4、β9-β10、β14-β15ドメインのループ間相互作用と構造安定性
SARS-CoV-2 三量体スパイク複合体の低温 EM 構造とその提案された立体構造状態によると、これらのループは柔軟性が高く、RBD から離れた三量体スパイク複合体の外表面に露出しています。
これらの相互作用ループに対応するタンパク質表面は親水性であり、いくつかの荷電/親水性アミノ酸の存在により正の電位を持っています (図 2A)。
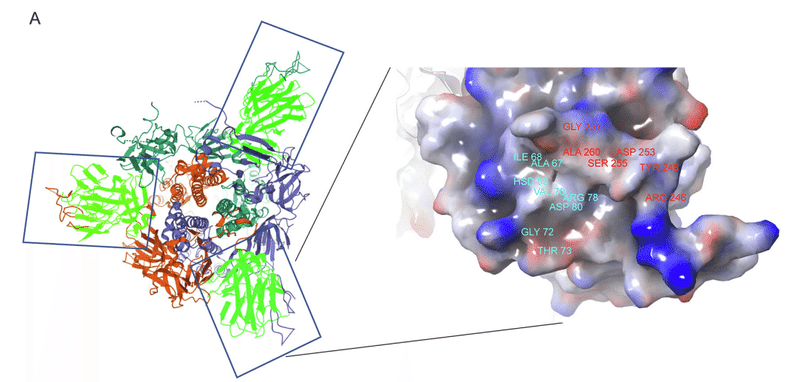
注目すべきことに、β3-β4およびβ14-β15ループ(それぞれアミノ酸62~80および242~263)は近接しており、両方のループのアミノ酸間の静電相互作用によって安定化されています(図2B)。
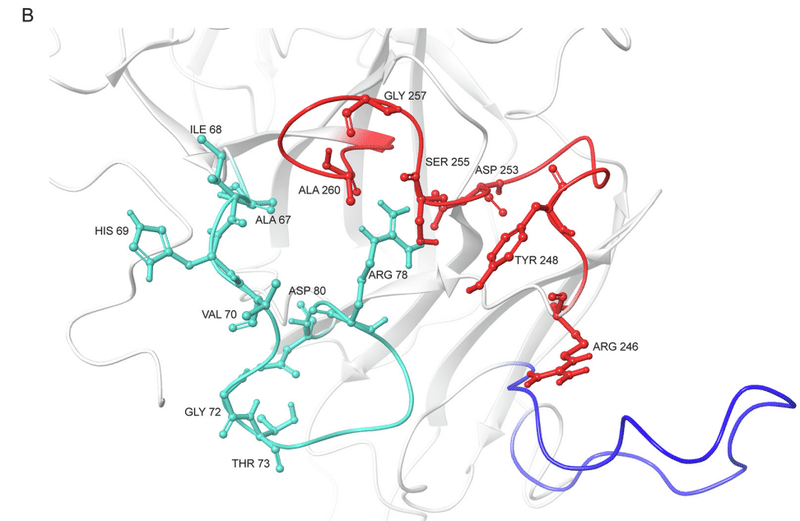
我々の構造解析により、ループβ14-β15およびβ3-β4のいくつかの残基間の静電的および疎水的相互作用のネットワークがループ間コミュニケーションを媒介していることが明らかになった。
より具体的には、β3-β4ループのアミノ酸残基Ala 67およびHis 69は、β14-β15ループのLeu 242、Tyr 248、Gly 261、Ala 262、Ala 263およびTyr 265のアミノ酸と相互作用します(図 3A、B の代表的な相互作用を参照)。
さらに、同じくβ3-β4ループにあるAsp 80は、β14-β15ループのLeu 242と相互作用し、この三次構造をさらに安定化します(図3C)。
最後に、β9-β10 ループ内の Val 143 は、β14-β15 ループの複数のアミノ酸 (Leu 244、His 245、Tyr 248 および Leu 249) と相互作用し、ループ間相互作用をさらに安定化します (図 3D)。

興味深いことに、最もよく特徴付けられた中和抗体の 1 つである 4A8 は、β9-β10 および β14-β15 アミノ酸(Tyr 144、Tyr 145、His 146、Lys 147、Lys 150、Trp 152、His 245、Arg 246、Ser 247、Tyr 248、Leu 249)を含む不連続エピトープ を認識する NTD ターゲティング抗体の最初のメンバーでした。
これらのアミノ酸は、抗体から 4 Å 以内の距離の認識界面に位置しています。
上で述べたように、いくつかのエピトープアミノ酸は、残基 Ala 67、His 69、および Asp 80 (図 4A)、および Val 143 (図 4B) との広範な相互作用ネットワークに参加しています。 これらの相互作用が抗体認識にとって重要であることを示唆しています。
最近の研究では、NTD の免疫原性特性が強調されており、4A8 以外にも、広範囲の中和抗体が NTD スーパーサイトを認識することが確認されています。
COV57、S2L28、S2M28、S2X333、2-17、5-24、4-8、および 2-51 抗体によって認識されるエピトープの構造分析により次のことが明らかになりました。つまり、 β3-β4、β9-β10、およびβ14-β15 ループがこのユニバーサル エピトープの形成に重要な役割を果たしてます (図 5A、B)。
したがって、これらのループ内のアミノ酸に影響を与える変異または欠失は、このエピトープを改造し、これらの抗体の結合親和性を変化させると予想されます。
グリコシル化による露出したアミノ酸残基のマスキングは、ウイルス免疫回避の一般的なメカニズムとして説明されています。
したがって、2 つのグリコシル化部位がループ β3-β4 および β14-β15 の残基 Asn 74 および Asn 149 で同定されたことに言及することが重要です。
3.3 β3-β4、β9-β10、およびβ14-β15ループ上のアミノ酸の多様性は、SARS-COV-2株のスパイクタンパク質NTD領域の進化の動態を調節
NTD二次構造における特定されたSARS-CoV-2変異の分布を調査するため、NTD二次構造における特定されたSARS-CoV-2変異の分布を調査するため、2021年7月9日時点のGISAID SARS-CoV-2データベースを通じて2,022,459の高品質ゲノム配列からの変異データを分析し、SARS-CoV-2スパイクタンパク質(YP_009724390)の1~350アミノ酸配列にアラインメントしました。
さまざまな SARS-CoV-2 株で特定された NTD 非同義変異の約 46.4% が、β3-β4、β9-β10、β14-β15 ループ領域内に見られることが観察されました。これは、他の NTD 二次構造要素と比較して、これらの要素の変異度が高いことを示しています (図 6A-C)。
β3-β4、β9-β10、β14-β15 ループは NTD コアおよび RBD との相互作用面から離れた位置にあるため、これらのループの変異は選択圧が低い可能性があります。
一方、特定されたループ変異のほとんどは、スパイクタンパク質の溶媒アクセス可能な表面に影響を及ぼし、これらの変異は糖または抗体との分子間相互作用のダイナミクスに影響を及ぼす可能性があります。
これはRNAウイルスにとって特に重要です。なぜなら、RNAウイルスのタンパク質の変異率が高いと、宿主の免疫反応からの逃避、毒性の上昇、組織指向性の変化につながることが知られているからです。
興味深いことに、β3-β4およびβ9-β10ループの変異は、β14-β15ループの変異と比較して高い頻度を示しており、SARS-CoV-2の進化において、これら2つの領域での変異がより動的な役割を果たしていることを示唆しています。
3.4 β3-β4およびβ9-β10ループ内の特定の変異は構造変化を引き起こす可能性がある
β3-β4 および β9-β10 ループ内の変異がかなり頻繁に見られることがわかったため、これらの領域における特定の変異が NTD の構造安定性を調節できるかどうかを調べようとしました。
前述のように、スパイクタンパク質の 3D 構造の解析により、Ala 67、Asp 80、Val 143 残基がループ間相互作用の強固なネットワークを維持していることが明らかになりました。
したがって、これらのアミノ酸の変異は、β3-β4/β14-β15 相互作用の安定性を変える可能性があります。
実際、位置 67 の Ala から Val への置換 (A67V) は、Ile 100、Phe 79、および Ala 263 残基との新たな疎水性相互作用の確立に関連しています (補足図 1A)。
さらに、Asp 80 (補足図 1B) の相互作用の多くは、Tyr (補足図 1C) または Ala (補足図 1D) への変異によって失われます。
具体的には、D80Y は His 66 との相互作用をなくしますが、D80A は Phe 65、His 66、および Pro 82 と相互作用しません。
これらの変異体と再配線された相互作用ネットワークとの関連により、構造変化が誘発される可能性があります。
一方、位置 143 の Val を Phe に置換すると (V143F)、Leu 244、His 245、および Leu 249 との芳香族および疎水性相互作用が失われると予測されます。
3.5 疫学的データは、β3-β4、β9-β10、β14-β15ループ内の特定の変異と欠失がSARS-CoV-2の進化の過程で正の選択を受けたという証拠を提供
ウイルスの変異率が高いため、安定性、感染率、免疫回避率が高く、系統の自然淘汰が行われるというユニークな機会が生まれます。
その結果、特定の変異株は、正の選択を通じて、集団内での存在感が高まっています。
2021年6月現在、Nextstrainは13の主要な系統群(19A~B、20A~20J、21A)を特定しています。
SARS-CoV-2 の進化の過程で、これらの系統群内でさまざまな変異株が出現し、高い伝染性を示し、COVID-19 の疫学に影響を与えました。
COVID-19パンデミックへの対応として、懸念される変異株(VOC)や関心のある変異株(VOI)としても知られるこれらの株の世界的な監視が確立されており、世界保健機関(WHO)はラベル付けを容易にするためにギリシャ文字1文字の命名スキームを採用しています。
2021年7月15日までに、アルファ(パンゴ系統指定B.1.1.7)、ベータ(B.1.351)、ガンマ(B.1.1.28.1)、デルタ(B.1.617.2)VOCの世界的分布が高かったことが報告されています。
WHOの疫学データに関する発表された報告書によると、アルファ、ベータ、ガンマ、デルタ株の伝染性は、元の武漢株と比較して、それぞれ29%、25%、38%、97%増加しています。
同様に、イータ(B.1.525)、イオタ(B.1.526)、カッパ(B.1.617.1)、ラムダ(C.37)などの複数のVOIは、その蔓延が拡大していることから、新たなリスクを示すものとして特定されています。
B.1.1.318、B1.1.375、B.1.1.616株のゲノム特性が伝染性に大きな影響を与えるという疫学的証拠も存在し、これらの株は監視対象の変異体として特徴付けられています。
報告された VOC/VOI と現在監視中の指定変異体 B.1.1.318、B1.1.375、B.1.1.616 と、β3-β4、β9-β10、β14-β15 ループの NTD 変異/欠失との関連から、ガンマ株以外のすべての非常に攻撃的な株は、普遍的な NTD エピトープを形成するこれらの 3 つのループ領域に複数の変化があるという特徴があることが明らかになりました (図 7A)。
特に、欠失はすべてのループ領域に分布しており、β3-β4 ループと β9-β10 ループではそれぞれ Δ69-70 と Δ144 が特に多く、これらはいくつかの異なる VOC/VOI (Alpha、Eta、B.1.1.616) に存在します。
さらに、Δ69-70 と Δ144 の共存は、アルファ株とイータ株で見られます。
その結果、患者から採取した SARS-COV-2 の遺伝子型配列におけるこれら 2 つの欠失の頻度は、ここ数か月で非常に高くなっています (図 7B、C)。
NTD 抗原スーパーサイトの形成に重要な役割を果たす β9-β10 ループは、攻撃的な株では異なる欠失パターンを示すようで、Δ144 のほかにも B.1.1.616 株ではΔ144-145 、デルタ株では Δ156-157 が特定されています。
興味深いことに、β14-β15 ループの欠失は、これまで Δ246-252 欠失を持つ Lambda 株でのみ発見されています (補足図 2)。
特に、ベータ株に存在する Δ241-243 は、β14-β15 ループに隣接する領域に影響を及ぼしますが、スーパーサイト形成への影響はまだ研究されていません。
監視中の VOC/VOI および変異体では、上記の欠失パターンが特定のミスセンス変異と共存しています。
具体的には、これらの株の遺伝的特性に関連するβ3-β4ループの変異には、A67V、G75V、T76I、およびD80Aが含まれます。
一方、β9-β10 内の G142D、W152C、E154K、および β14-β15 ループ内の R246I および D253G は、他の 2 つのループで特定されたミスセンス変異です (図 7A)。
株間で変異/欠失が重複しているにもかかわらず、遺伝子変異のさまざまな組み合わせにより複雑なウイルス遺伝子型が生成されますが (図 7B、C、補足図 2)、これらの組み合わせの機能的意味を評価することは困難です。
これらの獲得された変化は、ループ間およびループ内相互作用のネットワークを変更し、NTD抗原スーパーサイトの構造リモデリングを誘発すると予想されます。
実際、上記のループ内の欠失は、COVID-19 患者の回復期血清またはモノクローナル抗体 (mAbs) の結合力と中和力を低下させると提案されています。
このため、これらの欠失は免疫回避を促進し、その結果、正に選択されると考えられています。
一方、最近の研究では、NTD 抗原スーパーサイトの構造変化における特定のミスセンス変異の役割が強調されています。
機能データでは、これらの変化と免疫回避との相関も示されています。
たとえば、W152C 変異は、新しいジスルフィド結合を形成できる遊離システインを導入します。
イプシロン(B.1.427/B.1.429)株では、C136とW152Cの間の代替ジスルフィド結合がNTD構造変化を引き起こし、NTDを標的とする中和抗体からの免疫回避につながると提案されています。
さらに、G142D は NTD を標的とする抗体の結合を変化させることも示されています。
特に、NTD スーパーサイトにおけるループ間相互作用を維持すると予測される β3-β4 ループ内の Ala 67 および Asp 80 残基は、Eta (A67V) および Beta (D80A) 株で変異しています。
私たちの分析と一致して、D80A によって誘発される NTD リモデリングも最近提案されています。
4. Discussion
SARS-CoV-2 は、2002 年後半に出現し、重症急性呼吸器症候群の発生を引き起こした致死的なコロナウイルスである SARS-CoV と遺伝的に関連しています。
SARS-CoV は致死率が非常に高かったが、強力な公衆衛生対策の後、2003 年に根絶されました。
新しいコロナウイルス SARS-CoV-2 は致死性は低いものの、伝染性ははるかに高い。
さらに、SARS-CoV は肺細胞と小腸の腸管上皮細胞に感染するようです。一方、SARS-CoV-2 は腸、肝臓、腎臓、血管など複数の臓器に感染する可能性があります。
SARS-CoV-2 スパイクタンパク質 NTD 上の、宿主との相互作用を調節する可能性のある異なる構造要素を特定するために、SARS-CoV と SARS-CoV-2 NTD の比較配列および構造解析を実施しました。
予想通り、SARS-CoV と SARS-CoV-2 の NTD 配列は非常に類似しています。
最も顕著な違いは、SARS-CoV-2 と SARS-CoV-2 に密接に関連するゲノムを持つ特定のコウモリコロナウイルスでは、ループ β3-β4、β9-β10、β14-β15 の長さが著しく長いことです。これは、これらの要素の構造的進化が SARS-CoV-2 系統のアイデンティティの特徴であることを示しています。
コウモリとヒトのβコロナウイルスのそれぞれのループに対応する配列間の相同性が低いことから、これらの要素のアミノ酸変異がSARS-CoV-2系統内のスパイクタンパク質の分岐に大きな影響を与えていることが示唆されます。
クライオ電子顕微鏡データでは、これらの分岐ループ領域は柔軟性の高い NTD 領域の一部であることが示唆されていますが、私たちの分子モデリング データでは、β3-β4 および β9-β10 ループのいくつかの残基と β14-β15 ループの残基との間の静電的および疎水性相互作用のネットワークが、相対的な安定性を提供するループ間通信を仲介していることを示しています。
β3-β4 の Ala 67、His 69、Asp 80 残基と、β9-β10 ループの Val 143 残基が、これらの相互作用で重要な役割を果たすことが確認されました。
RBD を標的とする抗体が回復期血清の中和活性に大きく寄与することは広く認められています。
しかし最近の研究で、Voss 氏らは回復期血清中の IgG のプロテオーム プロファイルを分析し、反応は主に (>80%) RBD 外に存在するエピトープに向けられており、NTD 領域と S2 ドメインが含まれていることを実証しました。
同研究では、抗NTD抗体が中和に決定的に寄与し、その防御は血漿中の相対レベルに関係していることも報告されています。
この方向に向けて、数多くの免疫学的研究により、COVID-19 患者で誘発された NTD を標的とする抗体は有効な中和抗体 (nAbs) であることが実証されています。
Liu らによる最近の研究では、COVID-19 患者 40 名由来の血漿から分離された中和抗体のほぼ半数が NTD に結合したが、RBD には結合しませんでした。
中和抗体 4A8、COV57、2–17、5–24、4–8 およびおそらく他の抗体は、SARS-CoV-2 スパイクタンパク質の露出した NTD 表面を標的とします。
さらに、RBD を標的とする中和抗体は異なるエピトープを認識するのに対し、NTD に対するさまざまな中和抗体は、主にループ β9-β10 と β14-β15 (Chi らによると N3 と N5) で構成される共通部位を標的とします。
この領域は、グリカンが含まれておらず、エピトープ認識が可能であるため、免疫原性が高いと考えられています。
さらに、ループの柔軟性が高いため、ペプチドは複数の構造をとることができ、複数の抗体による認識に対応できます。
NTD 抗体の中和活性は妨害行動に依拠しています。 つまり、スパイクタンパク質が ACE-2 受容体に結合するのを防ぎます。
SARS-CoVに対する抗体反応の中和活性にはエピトープの立体構造が不可欠である一方、特定の変異体は特定の抗体の中和活性を逃れることができることは広く認められています。
β3-β4、β9-β10、β14-β15 ループの構造変化により NTD 抗体の認識が変化する可能性があるため、SARS-CoV-2 分岐ループの配列変異プロファイルを調査するために GISAID 変異データを分析しました。
私たちの分析により、これらのループの突然変異率が不釣り合いに高いことが明らかになりました。これは、SARS-CoV-2系統内のスパイク配列の分岐と進化において、免疫回避を強化する動的な役割を果たしていることを示しています。
スパイク 3D 構造の分子モデルに基づくと、Ala 67、Asp 80、Val 143 残基は相互作用の強固なネットワークを維持します。
この文脈では、A67V、D80Y、D80A、および V143F の変異体は、これらの相互作用のネットワークを再配線し、新しい疎水性相互接続の確立を促進するか (A67V)、ループ内水素結合 (D80Y、D80A) およびループ間疎水性相互作用 (V143F) の喪失を誘発すると予測されます。
これは、回復期血漿からの抗体を中和することでNTDの認識を妨げる可能性があります。
これに関連して、最近の研究では、SARS-CoV-2株のNTDアミノ酸配列のフレーム内欠失がβ3-β4(Δ69-70)、β9-β10(Δ141-144、Δ146)、β14-β15ループ(Δ243-244)に影響を及ぼすことが、患者の免疫逃避と関連していることが明らかになりました。
NTD ループに変異を持つ SARS-CoV-2 株が、より高い占有率と関連しているかどうかを特定するために、WHO が報告した VOC (アルファ、ベータ、ガンマ、デルタ)、VOI (イータ、イオタ、カッパ、ラムダ)、および高い伝染性を特徴とする監視中の株 (B.1.1.318、B1.1.375、B.1.1.616) に焦点を当てて Nextstrain データを調査しました。
注目すべきことに、これらの蔓延している株の多くについて、最近の機能研究により、それらの高い伝染性と、RBD および NTD を標的とする中和抗体から逃れる能力との間に関連性が確立されています。
NTD を標的とする抗体からの逃避に関しては、アルファ株とベータ株、およびデルタ株とイプシロン株について強力な実験的証拠が存在します。
この分析により、ガンマ株を除き、他のすべての非常に攻撃的な株は、β3-β4、β9-β410、およびβ14-β15ループに複数の変異と欠失を抱えていることが明らかになりました。
これらのループ領域における欠失の頻度が高いことは、欠失がミスセンス変異よりも抗原ドリフトの強い選択を受ける可能性があることを示している可能性があります。
中和抗体とNTDとの間の複合体の構造分析は、β9-β410およびβ14-β15ループは、β3-β4ループよりも抗体結合表面に近いことを示唆しています。β3-β4ループは抗体残基との相互作用に直接関与していません。
しかし、構造解析により、β3-β4 残基 Ala 67、His 69、Asp 80 が、β14-β15 ループを安定化するループ間相互作用において重要な役割を担っていることが明らかになりました。
β14-β15 ループは NTD 抗原性スーパーサイトの重要な部分であるため、β3-β4 ループの変異は免疫回避において重要な役割を果たす可能性があります。
興味深いことに、Ala 67 と Asp 80 は Eta (A67V) と Beta (D80A) 株で変異していますが、His 69 は Alpha、Eta、B.1.375 株で削除されています (Δ69-70)。
また、私たちの分析では、Val 143 が β14-β15 ループの複数のアミノ酸 (Leu 244、His 245、Tyr 248、Leu 249) と相互作用し、抗原性にとって重要な折り畳みで安定化していることも示されました。
Val 143 は、監視中の VOC/VOI および他の変異体のグループ内では変異していませんが、隣接する残基 Gly 142 および Tyr 144 の変異は、Alpha (Δ144)、Eta (Δ144)、Delta (G142D)、B.1.1616 (Δ144-145)、および B.1.617.1 (G142D) 株に存在します。
ここで提示されたすべての計算データは実験的検証に値すると認識しています。
それでも、私たちはウイルスと宿主の免疫システムの相互作用に関する重要な側面を明らかにしました。
私たちの研究結果は、急速に広がる新たなSARS-CoV-2変異体に対するより優れたプロファイルを備えたモノクローナル抗体およびワクチンの生成を促進する可能性があります。
以下省略。
この記事が気に入ったらサポートをしてみませんか?
