RBD 構造およびVOC間の共受容体結合における N343 グリコシル化の役割(2023年12月)
#GotGlycans: Role of N343 Glycosylation on the SARS-CoV-2 S RBD Structure and Co-Receptor Binding Across Variants of Concern
#GotGlycans: SARS-CoV-2 S RBD 構造における N343 グリコシル化の役割と懸念される変異体間の共受容体結合

Abstract
SARS-CoV-2 スパイク (S) タンパク質のグリコシル化は、ウイルス回避と適応度の両方に影響を与えるため、ウイルス進化の重要な標的となります。
ただし、タンパク質のグリコシル化はフォールディングと構造の安定性にとっても重要であるため、グリカンシールドの変化を成功させるのは困難です。
この枠組みの中で、構造的に不要なグリコシル化部位を同定することで、シールドの進化メカニズムへの洞察が得られ、免疫監視に情報を提供することができます。
この研究では、従来の分子動力学 (MD) シミュレーションと強化された分子動力学 (MD) シミュレーションからの 45 μs を超える累積サンプリングを通じて、免疫優性 S 受容体結合ドメイン (RBD) の構造が N343 の N-グリコシル化によってどのように制御されるか、このグリカンの構造的役割が WHu-1、アルファ (B.1.1.7)、ベータ (B.1.351) からデルタ (B.1.617.2) およびオミクロン (BA.1 および BA.2.86) バリアントにどのように変化するかを示します。
より具体的には、N-グリカンの両親媒性の性質がRBD疎水性コアの構造的完全性を維持するのに役立ち、N343でのグリコシル化の喪失が特異的かつ一貫した構造変化を引き起こすことを我々は見出しました。
我々は、RBD構造を強化する変異により、この変化がWHu-1、アルファおよびベータRBDの受容体結合モチーフ(RBM)の立体構造をアロステリックに制御するが、デルタおよびミクロン変異体では制御しないことを示します。
これらの発見を裏付けるように、モノシアル化ガングリオシド共受容体への RBD の結合は、WHu-1 の N343 グリコシル化に大きく依存するが、デルタ RBD では依存しないこと、および親和性が VoC 間で大きく変化することを示します。
最終的に、この研究で私たちが提供する分子的および機能的洞察は、タンパク質の構造と機能におけるグリコシル化の役割についての理解を強化します。 また、N343 のグリコシル化部位が SARS-CoV-2 S グリカン シールドの変異のホットスポットになり得る構造的制約を特定することもできます。
Introduction
SARS-CoV-2 スパイク (S) 糖タンパク質は、ウイルスと宿主細胞の融合を担い、COVID-19 につながる感染を開始します。
S は、2 つのトポロジカル ドメイン、つまり S1 と S2 に細分される構造を持つホモ三量体です (図 1a を参照)。フューリン部位によって分離されており、融合前構造で切断されます。

武漢-Hu-1株(WHu-1)、そして依然として懸念されるほとんどの変異株(VoC)では、 宿主細胞融合は主に、宿主細胞表面にあるアンジオテンシン変換酵素 2 (ACE2) 受容体への S 結合によって引き起こされます。
このプロセスは、細胞外マトリックス内のヘパラン硫酸 (HS) などのグリカン共受容体と、宿主細胞の表面から覗くモノシアル化ガングリオシド オリゴ糖 (GM1os および GM2os) によってサポートされています。
ACE2 との相互作用には、「開口部」として知られる S の劇的な構造変化が必要であり、S1 サブドメイン内の 1 つ以上の受容体結合ドメイン (RBD) が露出します。
ACE2 表面と直接接触する RBD の領域は、受容体結合モチーフ (RBM) として知られています。
最終的に、S が ACE2 に結合すると、S1 サブドメインの脱落と融合後立体構造への移行が引き起こされ、宿主細胞表面近くの融合ペプチドが露出し、ウイルスの侵入が引き起こされます。
その機能を発揮するために、S はウイルスのエンベロープから突き出て認識にさらされます。
宿主免疫系を回避するために、エンベロープウイルスは宿主細胞のグリコシル化機構を乗っ取り、グリカンシールドとして知られる宿主炭水化物の緻密なコートでSを覆います。
SARS-CoV-2 では、グリカンシールドが S タンパク質表面の 60% 以上を効果的に遮蔽します。RBD が開いている場合、N 末端ドメイン (NTD) の領域は免疫認識に対して脆弱になります。
RBD は血清中和抗体の約 90% を標的とするため、抗体の特異性や宿主細胞の共受容体との相互作用をスクリーニングするだけでなく、COVID-19ワクチンのタンパク質足場としても非常に効果的なモデルです。
その直接的な結果として、RBD は大きな進化圧力にさらされています。
免疫回避を引き起こす RBD の変異は、特にそのような変化によって ACE2 への結合親和性が強化される場合や、別の侵入経路へのアクセスが可能になる場合に特に懸念されます。
突然変異のホットスポットの特定と、RBM 内およびその周囲の突然変異の影響については、これまでも、そして現在も厳しい精査が行われています。
しかし、感染力や免疫逃避に劇的な変化をもたらすことがわかっているグリカンシールドの変異にはあまり注目されていません。
糖鎖シールドの変化を成功させることは進化的に困難です。これは、S のグリコシル化の性質とパターンがウイルスの侵入と回避の効率にとって重要であるだけでなく、フォールディングを促進し、機能的なフォールディングの構造的完全性を維持するためにも重要であるという事実によるものです。
したがって、Sシールド内の潜在的な進化的ホットスポットを特定することは複雑な問題ですが、免疫監視にとって非常に重要です。
新しいグリコシル化部位の喪失、シフト、獲得などのシールドの潜在的な変化は、基礎となる機能的なタンパク質構造の完全性に悪影響を及ぼさない場合にのみ発生する可能性があります。
この研究では、RBD 上の重要なグリコシル化部位である N343 のケースを提示し、議論します。
45 μs を超える分子動力学 (MD) シミュレーションからの大規模なサンプリングの結果は、次を示しています。つまり、 N343 での N-グリコシル化の喪失が RBD の構造、動態、共受容体結合にどのような影響を与えるか、そしてこれらの効果が、WHu-1株から指定されたVoC(アルファ株・ベータ株・デルタ株・オミクロン株)に至るまで、基礎となるタンパク質の変異によってどのように調節されるのかということです。
さらに、我々は、オーミクロン BA.2.86 RBD の構造とダイナミクスについての重要な洞察を提供します。
モニタリング中の変異体(VUM)として指定され、一般に「ピロラ」と呼ばれるこの変異体は、N354に新たに獲得されたN-グリコシル化部位を有しており、これは祖先株以来のRBDシールドの最初の変化を表しています。
WHu-1 (中国、2019) から EG.5.1 (中国、2023) までの SARS-CoV-2 S RBD (aa 327-540) は、高度に保存された 2 つの N-グリコシル化部位 (1 つは N331、もう 1 つは N343) を示しています。
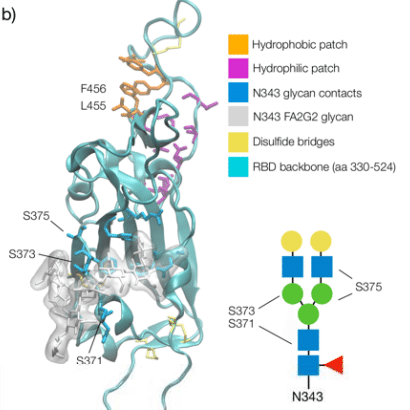
N331 のグリコシル化は RBD を NTD に連結する柔軟性の高い領域に位置しますが、N343 グリカンは RBD の大部分を覆い、タンパク質表面全体に伸びて、ベータ シート コアを構成する 2 つのらせん領域を接続する橋を形成します。 図 1b を参照してください。
この研究では、N343 グリカンの除去が WHu-1 の構造変化を誘導することを示します。アルファとベータは、RBM の構造とダイナミクスをアロステリックに制御します (図 1c を参照)。

デルタおよびミクロンでは、RBD アーキテクチャを強化する突然変異によってこれらの効果が大幅に減衰します。
この分子的洞察に加えて、我々は、N343 グリカンの酵素的除去が WHu-1 RBD のモノシアル化ガングリオシド共受容体の結合に影響を与えるが、デルタ株では影響しないことを示します。
また、GM1 GM1os および GM2 GM1os に対する RBD の親和性が VoC 間で大きく変化し、ベータとオミクロンが最も弱い結合を示すことも観察されました。
最終的に、この研究で我々が提供する分子的洞察は、タンパク質のフォールディングと構造安定性におけるグリコシル化の役割を裏付ける証拠をさらに増やします。
この情報は構造生物学の中心であるだけでなく、糖鎖を含む場合と含まない場合がある新しいCOVID-19ワクチンの設計にも重要であり、シールドを制御する進化メカニズムを理解するのにも役立ちます。
Results
このセクションでは、RBD の構造の簡単な概要から始めて、次に RBD 構造が ACE2 との相互作用によってどのように調節されるか、そしてなぜ N343 グリカンがその安定性に不可欠であるかを説明します。
次に、N343 グリコシル化の喪失が VoC の RBD 構造と GM1os および GM2os に対する結合親和性にさまざまな程度で影響を与える方法と理由を説明します。
SARS-CoV-2 S RBD structure and antigenicity.
SARS-CoV-2 S RBD の構造と抗原性。
SARS-CoV-2 S RBD には、構造化された領域と本質的に無秩序な領域の両方が含まれます。
構造化領域は、N343の架橋N-グリカンによって連結された2つの隣接する部分的に螺旋状のループ(aa 335-345およびaa 365-375)で囲まれた、主に疎水性のベータシートコアによって支えられています(図1bを参照)。

aa 335-345 ループには N343 グリコシル化部位があり、クラス 2 およびクラス 3 抗体の標的となる重要な抗原領域の一部です。
架橋構造では、N343 グリカン五糖は RBD ベータ シート全体に伸びてアミノ酸 365 ~ 375 ループに到達し、バックボーンおよび残基 365 ~ 375 の側鎖との高密度の水素結合および分散相互作用を形成します。 図 1b、c、および S.2 を参照してください。
架橋N343グリカンはRBDの疎水性ベータシートコアを周囲の水から守り、エネルギー的に好ましくない接触を防ぎます。
その両親媒性の性質により、N343 はそのコア GlcNAc-β(1-4)GlcNAc を介してベータ シートの疎水性残基と分散相互作用を形成します。 同時に、周囲の水およびaa 365-375のらせんループと水素結合します。
注目すべきことに、このループ内の重要なアンカー残基 S371、S373、および S375 はすべて、すべてのオミクロン株 (BA.1-2、BA.4-5、BQ.1.1、EG.5.1、XBB.1.5) で疎水性残基に変異しています。
受容体結合モチーフ (RBM) は aa 439-506 を包含し、ACE2 と直接接触するすべての RBD 残基をカウントします。
RBM はクラス 1 とクラス 2 の両方の抗体によって強く標的にされ、高い進化圧力下にあり、すべての VoC がこの領域に変異を持っています。
以前の MD シミュレーション研究で示されているように、非結合 S の RBM は大部分が構造化されておらず動的であり、この洞察はこの領域の低解像度クライオ EM マップによっても裏付けられています。
RBM の固有の柔軟性は、RBD の開閉メカニズムにおける重要な特徴であると考えられます。 ここで、隣接する RBD からの N343 が閉じた立体構造でタンパク質と結合し、RBD が開きます。
RBM の唯一の比較的構造化された領域は、ここで親水性パッチとして定義されるものです (図 1b を参照)。 ヘアピンは、連結した塩橋と極性残基、すなわち R454、R457、K458、K462、E465、D467、S469、および E471 のネットワークによって安定化され、RBD が閉じているときに S の内部に面します。
ACE2 または抗体と複合体を形成すると、RBM は SARS-CoV-1 S RBD と共有される構造化された折り畳みを採用します。
この立体構造では、この研究や他の研究者によって示されているように、RBM の末端ヘアピン (aa 476 ~ 486) のみが高度な柔軟性を保持しています。
RBM結合フォールドは、ACE2とのタンパク質界面の一部である芳香族残基および脂肪族残基L455、F456、Y473、A475の積み重ねによってサポートされる疎水性パッチによって安定化されます(図1bを参照)。
注目すべきことに、疎水性および親水性パッチ内のすべての残基は、おそらく RBD を ACE2 結合立体構造に誘導および/または安定化する重要な機能のため、VoC 全体で高度に保存されています。
興味深い観察として、EG.5.1 変異体 (中国、2023) における最近の F456L 変異による疎水性パッチでのスタッキングの喪失は、適切に名付けられた FLip 変異体における L455F 変異によって回復されています。
スクリーニングからの証拠に基づいて、我々はRBDを、異なるクラスの抗体の標的となることが知られている3つの異なる抗原領域に細分しました。図1dを参照してください。

領域 1 は、N343 グリコシル化部位を含む aa 337-353 に広がり、クラス 2 および 3 抗体の標的となる残基をカウントします。
領域 1 の aa 配列はこれまでのところ高度に保存されており、S309 などのすべての VoC にわたって特定の抗体が中和活性を保持できます。S309 の結合モードには N343 グリカンも直接関与します。
領域 1 におけるこの高度な保存性の注目すべき劇的な例外は、「ピロラ」として知られる BA.2.86 変異体 (デンマーク、2023) です。この変異体では、K356T 変異により N354 に新しい N-グリコシル化セクオンが導入されています。
領域 2 は RBM と一致しており、ACE2 および中和抗体への結合に加えて、隣接する RBD からの N370 グリカンへの結合にも使用されます。
SARS-CoV-2 では N370 グリコシル化が失われており、特にグリコサミノグリカン、血液型抗原、モノシアル化ガングリオシドなどのグリカン共受容体に結合するために利用できる RBM 結合裂が存在します。
領域 3 は、aa 411 ~ 426 の間に伸びる短い比較的構造化されたループで、領域 1 に対して RBD の反対側に位置します (図 1d を参照)。
Effect of the loss of N343 glycosylation on the structure of the WHu-1, alpha and beta RBDs.
WHu-1、アルファおよびベータ RBD の構造に対する N343 グリコシル化の喪失の影響。
WHu-1 株とアルファ (B.1.1.7) およびベータ (B.1.351) VoC について得られた結果は、それらの配列と構造の類似性により一緒に議論されます。 WHu-1 RBDと比較して、アルファは1つの突然変異(N501Y)のみをカウントし、ベータは3つの突然変異(K417N、E484KおよびN501Y)をカウントします。
従来のMDによる広範なサンプリング、 つまり、アルファおよびベータ VoC では 4 μs、WHu-1 RBD では 8 μs とガウス加速 MD (GaMD) の追加 4 μs(表S.1を参照)は、N343 グリコシル化の喪失が RBD の劇的な構造変化を誘導することを示しています。疎水性ベータシートコアに隣接する一方または両方の螺旋ループが互いに引き寄せられます。 図 2 を参照してください。

この構造変化は、N-グリカンの除去時に非常に急速に起こることもあれば、構造エネルギー状況の複雑さにより長い遅延の後に起こることもあります。
分析に使用されるデータは、構造的安定性、つまり平衡に達したシステムに対応します; 構造遷移に対応する時間枠を破棄しました。
WHu-1 RBD における N343 グリコシル化の喪失の影響を調べるために、さまざまな立体構造から MD シミュレーションを開始しました。
従来のサンプリング MD 軌道 (MD1) の 1 セットと GaMD シミュレーションでは、開始構造は以前の研究で得られた MD 平衡化外部ドメインからの開いた RBD に対応します。
このシステムでは、RBM は展開され、最大限の柔軟性が維持されます。
MD2 は、ACE2 結合構造に対応する立体構造から開始されました。
MD2 および GaMD から得られた結果は、MD1 の結果と完全に一致しています。 したがって、図 S.2 に補足資料として含まれています。
GaMD シミュレーションでは、N343 グリカンと反対側のループの aa 365-375 ストレッチとの接触度が低いことが示されています (図 1c を参照)。これは、ほとんどの接触が 365 位からさらに下流の残基とであるためです。
それにもかかわらず、N343 はシミュレーション全体を通じて架橋構造を維持したままになります。
図 2 のカーネル密度推定 (KDE) によって表される RMSD 値の分布が示すように、WHu-1 RBD の領域 1 の構造は安定しています。
グリコシル化された RBD では、領域 1 の安定性は主に、シミュレーションを通じてループ aa 365 ~ 375 の残基と水素結合を形成する架橋 N343 FA2G2 グリカンの寄与によるものです。図 1c および S.2 を参照してください。
N343 グリコシル化の喪失は、WHu-1 RBD の領域 1 の構造変化を引き起こします。これは、図 2a のより広い KDE ピークによって示されています。
水素結合による Y351 (領域 1) と S469 または T470 (領域 2) の間の非共有相互作用ネットワークの破壊、および CH-π スタッキングによる Y351 と L452 (領域 2) の非共有相互作用ネットワークの破壊によって、この構造変化は最終的に領域 1 からの親水性ループの完全な分離を引き起こします (図 1c を参照)。
N343 でのグリコシル化の喪失による領域 3 の構造変化は無視できるほどであるようです。
アルファ RBD のシミュレーションに使用される開始構造は、N501Y 変異で修飾された WHu-1 RBD (PDB 6M0J) の ACE2 結合立体配座に由来します。
N343 の再構成されたグリカンは、軌道全体を通じて aa 365 ~ 375 と相互作用しますが、図 2b に示すデータの収集を開始した 830 ns 以降にのみ安定した立体構造をとります。
ここでも、N343 でのグリコシル化の喪失により、aa 335-345 ループと aa 365-375 ループが互いに近づく急速な構造変化が引き起こされることがわかります (図 2b を参照)。

この構造変化は主に領域 1 に関係し、前のケースと同様に、最終的には領域 1 の Y351 からの親水性パッチの剥離を決定します。
また、図2bのKDEプロットに示されているように、N343グリコシル化αRBDの軌道中に、らせんターンの部分的な破壊と再折り畳みを伴う領域3の小さな構造変化が観察できます。
前のケースと同様に、少なくともこの研究で蓄積されたサンプリング内では、領域 3 の構造は N343 グリコシル化の影響を受けないようです。
ベータ RBD (PDB 7LYN からの開始構造) では、再構築された N343 グリカンは非常に急速に架橋構造を採用し、わずかな偏差のみで軌道全体にわたってこの構造を維持します。
領域 1 ~ 3 の対応する RMSD 値 KDE 分布 (図 2c を参照) は、この構造安定性を反映しています。

前述したように、RBM (領域 2) の安定性は、Y351 (領域 1) と親水性ループの間の相互作用によってサポートされています。
N343 でのグリコシル化の喪失により、RBD コアのらせんループが互いに急速に緊密になります。この場合も、最終的には、MD 軌道の終わりに向かって、つまりサンプリングの 1.9 μs 後に、領域 1 の Y351 から親水性ループが分離します。
Effect of the loss of N343 glycosylation on the binding affinity of GM1os /2 os for the WHu-1, alpha and beta RBDs.
WHu-1、アルファおよびベータ RBD に対する GM1os /2 os の結合親和性に対する N343 グリコシル化の喪失の影響。
私たちの何人かによる以前の研究では、WHu-1 RBDがSARS-CoV-2感染における共受容体としてモノシアル化ガングリオシドに結合することが示されています。
GM1os と WHu-1 RBD 間の低親和性複合体の 3D 構造は、実験的に取得することが困難であることが証明されています。それでも、WHu-1 RBD結合N370に対するGM1osの複数の構造アラインメントと広範なMDシミュレーションの組み合わせを通じて、有望なモデルを得ることができました。詳細は補足資料および参考文献を参照してください。
予測される GM1os 結合部位は、WHu-1 RBD の領域 1 と領域 2 の間の接合部に位置し、この領域を安定化するすべての残基、すなわち Y351、L452、S469 および T470 が関与します。図 1c と 2e を参照してください。

SARS-CoV-2 RBD に結合するグリカン共受容体に関する研究の一環として、直接 ESI-MS アッセイを使用して、N343 グリコシル化の喪失が GM1os および GM2os の結合に及ぼす影響を調べました。
ここでは、endoF3 処理を使用して、フコシル化二分岐および三分岐複合 N-グリカンをコアの非フコシル化またはフコシル化 GlcNAc (それぞれ Gn または GnF) にトリミングしました。
LC-MS 分析は、WHu-1 RBD の N331 ではなく N343 上の N-グリカンがトリミングされたことを示唆しています (図 S.6)。
endoF3 処理 WT RBD のゼロ電荷質量スペクトルから (図 S.5)、 グリカンの割り当てを実行したところ (表 S.8)、検出されたグリコフォームの 31% には N343 に Gn/GnF が含まれており、残りは無傷のグリコフォームであることがわかりました。
図 2d の親和性データは、WHu-1 RBD から N343 グリカンを酵素的に除去すると、GM1os/2os 結合が完全に失われることを示しています。 これは、領域 1 と 2 の間の結合の関与、結合と RBM ダイナミクスのアロステリック制御の両方と一致します。

さらに、アルファ RBD への GM1os および GM2os の結合は、WHu-1 と比較してわずかに減少しているように見えますが、ベータ RBD への結合は劇的に減少しています。
この発見は、E484 と GM1os の間の重要な相互作用を変化させる、ベータ版の変異 E484K と一致します。図 2 を参照してください。 また、変異によって引き起こされる RBM 末端ヘアピンの構造と動態の変化も、S 開口速度に影響を与えることが示唆されています。
Effects of N343 glycosylation on the structure of the delta RBD.
デルタ RBD の構造に対する N343 グリコシル化の影響。
デルタ (B.1.617.2) RBD は、WHu-1 株と比較して、2 つの突然変異、すなわち L452R および T478K を保有しています。
クライオ EM 構造 PDB 7V7Q のオープン RBD は、グリコシル化デルタ RBD と非グリコシル化デルタ RBD の両方の MD シミュレーションの開始立体構造として使用されました。
デルタの変異が RBD 構造にどのように影響し、N343 でのグリコシル化の喪失に対する応答を調節するかを理解するために、グリコシル化システムと非グリコシル化システムの両方について、相関のない従来の MD シミュレーション (2 μs) と 1 つの GaMD シミュレーション (2 μs) を実行しました。 合計 (累積) サンプリングは 12 μs です。
結果を図 3に示します。

グリコシル化デルタ RBD では、N343 グリカンが WHu-1、アルファ、およびベータ RBD よりもはるかに動的であることが観察され、ループ aa 365 ~ 375 に加えて RBD のさまざまな領域と接触しています。
これらの変動に応じて、RBD の立体構造は領域 1 および 3 の平均構造からわずかに逸脱するだけで安定した状態を保ちます。
すべての軌跡、特に図 3c の GaMD シミュレーションの結果は、デルタの RBM (領域 2) が非常に動的であることを示しています。
この柔軟性には、T478K 変異を含む RBM の末端ヘアピン (aa 476 ~ 486) が特に関与しているようです。 一方、残りの RBM は L454R 変異によりしっかりと固定されています。
より具体的には、デルタの R452 は、S469 および T470 に加えて、Y351 と新しい水素結合を確立することができ、領域 1 と 2 の間の結合を強化します。さらに、デルタRBMでは、Y351へのCH-πスタッキングにおけるL452の役割は、ベータシートのねじれを通じて近位のL492によって引き受けられます(図3eを参照)。
これらの相互作用は、R454 の配向の強化にも寄与し、RBM 親水性パッチとのつながりを強化します。
デルタ RBD に対する N343 でのグリコシル化の喪失の影響は、2 つの無相関 MD シミュレーションを実行することによって評価されました。1 つは従来のサンプリング (3 μs の MD1) によるもので、もう 1 つは強化されたサンプリング (2 μs の GaMD) によるものです。
図 3 に示す L452R 変異の結果として、N343でのグリコシル化の喪失時に起こるRBDの疎水性コア上のらせんループaa 335-345およびaa 365-375の締め付けは、領域1と2の間の接合の構造および動態に影響を与えません。 図S.3を参照してください。
従来の MD シミュレーションの結果は、ループの締め付けは主に、領域 1 ではなく aa 365-375 ループのより大きな変位によって達成されることを示しています。 一方、GaMD の結果は両方のループの締め付けを示しています (図 S.3 を参照)。
すべてのシミュレーションにおいて、領域 1 と 2 の間の接合部の構造は乱されず、収集したサンプル内の親水性パッチが剥離することはありませんでした。
Effect of the loss of N343 glycosylation on the binding affinity of GM1/2 for the delta RBD.
デルタRBDに対するGM1/2の結合親和性に対するN343グリコシル化の喪失の影響。
デルタ RBD のグリカン結合に対する N343 グリコシル化の影響を調べるために、直接 ESI-MS アッセイを使用して、endo F3 処理デルタと GM1os および GM2os の間の結合親和性を定量しました。
endoF3 処理 RBD のゼロ電荷質量スペクトル (図 S.5 を参照) から、グリカンの割り当てを実行しました (表 S.9 を参照)。その結果、N331 グリカンと N343 グリカンの両方が Gn/GnF までトリミングされたことがわかりました。
図 3f の直接 ESI-MS データは、N343 グリコシル化の喪失時にデルタ RBD における GM1/2 結合の喪失がないことを示しており、これはシアル化グリカン認識における領域 1 と 2 の接合部の関与をさらに裏付けています。

Effects of N343 glycosylation on the RBD structure in the omicron BA.1 SARS-CoV-2.
オミクロン BA.1 SARS-CoV-2 の RBD 構造に対する N343 グリコシル化の影響。
オミクロン BA.1 RBD は、WHu-1 株と比較して 15 個の変異を持っています。 すなわち、S371L、S373P、S375F、K417N、N440K、G446S、S477N、T478K、E484A、Q493R、G496S、Q498R、N501Y、Y505H、およびT547Kです。
最近流行した XBB.1.5、EG.5.1、BA.2.86 を含むすべてのオミクロン VoC に保持されている、S371L・S373P・S375F 変異は、WHu-1、アルファ、ベータおよびデルタ RBD の N343 グリカンとの水素結合相互作用に関与していることが確認されているすべてのヒドロキシル側鎖を除去します。図 1c および S.3 を参照してください。
我々は、2 セットの相関のない従来の MD シミュレーション (MD1 および MD2) と 1 セットの GaMD を介して、BA.1 RBD の構造における N343 でのグリコシル化の損失の影響を調査しました。合計累積シミュレーション時間は 12 μs でした。
開始構造は、PDB 7QO7 (MD1) および PDB 7WVN (MD2 および GaMD) のオープン RBD に対応します。 ここで、N343 グリカンは、N343 側鎖の空間的配向に応じて、異なる立体構造で再構築されました。
MD1 および GaMD シミュレーションの結果は、S371L、S373P、および S375F 変異にもかかわらず、N343 グリカンが依然として aa 365-375 ループと安定した接触を形成していることを示しています (図 1c および S.3 を参照)。 これらの相互作用は RBD 構造の安定性に寄与します (図 4 を参照)。

MD2 に使用した出発構造では、N343 グリカンは、RBD 疎水性コアから離れる方向を向いたコア五糖で構築されました。
その結果、N グリカンは MD2 軌道中に異なる一時的な構造をとり、RBD の疎水性内部および N331 グリカンとの相互作用で終了します。図 S.6 を参照してください。
すべてのシミュレーションにおいて、N343 でのグリコシル化の喪失により、aa 335-345 および aa 365-375 ループの締め付けが引き起こされます。 これは、L371およびF375側鎖の埋め込みによって駆動され、疎水性コア内のaa 365-375ループのより効率的なパッキングによってオミクロンで安定化されます。
非グリコシル化 RBD は安定な立体構造をとり、親水性パッチの剥離は見られません。
オミクロンにおけるaa 365-375ループ変異の安定化効果は、GM1os/2osasオミクロン(BA.1)に対する親和性によってはテストできませんでした。これらのエピトープには弱くしか結合しません。図3fを参照してください。
以前の研究で MD シミュレーションによって予測され(図 1d と 2e を参照)、ベータで観察された結合部位に基づいて、オミクロンにおける E484A 変異による E484 の喪失は、GM1os/2os 結合を無効にする可能性があります。
RBD 構造の安定性は、N354 に追加のグリコシル化部位が存在することによってさらに強化されます。この部位は、最近検出されたオミクロン BA.2.86 の「ピロラ」変異体に現れています。
図 4d ~ f に示すように、N343 と N354 の N-グリカンは領域 1 を安定させる軌道全体にわたって緊密に絡み合っており、その領域を非常に効果的にシールドしています。
N354 に追加の N-グリコシル化部位が存在すると、MD シミュレーションを実行するためのテンプレートとして使用した BA.1 開始構造と比較して、その部位をホストするループの立体構造も変化します (図 4f を参照)。

以前の糖プロテオミクス分析と、N354 で再構築されたグリカン構造の溶媒への曝露に基づいて、すべてのグリコシル化部位を FA2G2 N-グリカンで占めることを選択したことに注意してください。
Discussion
タンパク質のフォールディングと構造安定性におけるグリコシル化の役割を定量化することは、グリカン構造の動的な性質と特性評価を妨げるタンパク質の機能化におけるミクロおよびマクロの不均一性により複雑な作業です。
しかし、タンパク質のフォールディングは、グリコシル化の種類と占有率がその場で変化する可能性があるという状況の中で発生するという事実は、タンパク質が天然の折り畳みを達成して保持するには、すべてのグリコシル化部位が必須ではなく、機能に影響を与えることなくそれらの部位が置換される可能性があることを示唆しています。
この研究では、ウイルス系統発生において最も高度に保存されている部位の 1 つである SARS-CoV-2 S RBD の N343 における N-グリコシル化の構造的役割を調査しました。
この研究および我々や他の研究者による以前の研究における広範な MD シミュレーションは、RBD コアがこのグリカンによって効果的に保護されていることを示しています。
さらに、N343 グリカンは S の開閉に機構的に関与していることが示されており、このグリコシル化部位はウイルス感染に対して機能的に不可欠となっています。
この研究では、従来のスキームと拡張スキームの両方を使用して 45 μs を超える累積 MD サンプリングを実行し、次のことを示しました。 N343 グリカンは WHu-1 SARS-CoV-2 においても基本的な構造的役割を果たしており、この役割はこれまでに流行している変異株では変化していることがわかっています。
RBD フォールディングに向けて N343 グリコシル化がどの程度基礎的であるかを測定することはできませんが、 N343 の複合 N-グリカンの両親媒性の性質が RBD 構造の安定性を高め、疎水性の高いベータ シート コアを構成する 2 つの部分的に螺旋状のループの間を橋渡ししていることがわかります。
注目すべきことに、我々は以前の研究でN343のオリゴマンノース型N-グリカンについても同じ架橋構造を決定しました。
すべての変異体において、N343 のグリカンの除去により、疎水性コアへの水のアクセスを制限することを目的とした応答としてループの締め付けが引き起こされることが観察されています。
WHu-1、アルファおよびベータ RBD では、このイベントが RBM のダイナミクスをアロステリックに制御し、最終的には親水性パッチの剥離と ACE2 が認識する立体構造からのミスフォールディングを引き起こします。
これらの結果は、WHu-1 株における N331 および N343 グリコシル化の両方の欠失によって観察されたウイルス感染力の大幅な低下と一致しています。ここで、構造の損失は、ゲート制御による機能の損失に追加される可能性があり、またはその逆も同様です。
この分子洞察を裏付ける機能アッセイとして、モノシアル化ガングリオシド GM1os および GM2os のオリゴ糖に対する RBD の結合親和性が N343 グリコシル化によってどのように調節されるかを決定しました。
これらは、我々や他の研究者らによる以前の研究で、WHu-1 感染において共受容体として機能することが示されています。
我々は広範なMDサンプリングを通じて、GM1osおよびGM2osがN370の祖先N-グリカンの6アームが占める位置に正確に対応する部位にRBDを結合すると予測しました。
N370 部位は依然として人獣共通感染症サベルコウイルスに占有されていることに注意してください。
GM1os結合部位(図2eを参照)は、領域1と領域2の間の接合部に正確に位置しており、WHu-1のN343グリコシル化の喪失によって破壊されています。

したがって、N343 グリカンの酵素的除去により、WHu-1 RBD における GM1os および GM2os の結合が消失することがわかりました。図 2d を参照してください。

WHu-1 RBDと比較して親和性が低いという状況の中で、アルファでも同様の結合の喪失が予想される一方で、ベータRBDは、グリコシル化状態に関係なく、GM1osおよびGM2osに結合しないことがわかりました。
私たちが同定したGM1-RBD複合体の構造に基づくと(図 2e を参照)、 E484 がオリゴ糖との重要な接触を表す場合、ベータの E484K の変異は、この変異と同じ領域内の変異に関連する RBM 動態の変化とともに、結合喪失の鍵となる可能性があります。
デルタ変異体では、L452R 変異が RBD の構造安定性の向上に関与し、領域 1 と RBM の間の非共有結合的相互作用ネットワークが強化されることが観察されました。
実際、N343 グリコシル化の喪失によって生じるループの緊密化は、RBM のミスフォールディングを引き起こすわけではありません (図 3 を参照)。
したがって、N331およびN343グリカンをトリミングしたデルタRBDは、完全にグリコシル化された形態と比較して、GM1osおよびGM2osに対する結合親和性に有意な変化を示さないことがわかります。図3fを参照してください。

現在流通しているすべての VoC および VUM を含むすべてのオミクロン変異体では、N343 グリカンがフックするループ aa 365-375 に同様の変異が含まれています。 高度に保存された S371、S373、および S375 はすべて疎水性残基に変異しています。 図S.1を参照してください。

BA.1 および BA.2.86 RBD に関する我々の MD 結果は、371、372、および 373 位の疎水性残基が RBD コア内に詰め込まれる可能性があることを示しています。 バックボーンとの相互作用を通じて N343 グリカンの分岐をサポートできるループ構造をもたらします (図 4 を参照)。

我々は、すべての変異体について、N343 グリカンと反対側のループの aa 365-375 ストレッチの間の接触が、親水性 (水素結合) と疎水性 (分散またはファンデルワールス) タイプの相互作用の間にかなり均等に分布していることを示しました。 図 1c を参照してください。
したがって、アンカー水素結合残基の喪失は、他の相互作用を通じてサポートされ得ることが予想されます。
これに関連して、N343 グリカンの除去は依然としてループの引き締めを引き起こしますが、他の変異体とは異なるメカニズムを介して、最終的には RBM ダイナミクスに影響を与えないようです。
ベータと同様に、オミクロンについては、おそらく E484A 変異が原因で、N343 グリコシル化 RBD の GM1os および GM2os への結合は無視できます。これは、GM1os/RBD複合体の予測構造に基づくと(図 1d と 2e を参照)、予測結合部位内の重要な相互作用を否定することになります。
まとめると、我々の結果は、WHu-1、アルファおよびベータ株以来、N343 の N-グリコシル化部位が構造的に不要になるように RBD が進化したことを示しています。
この枠組み内では、N343 のすぐ近くの N-グリコシル化部位がフォールディングと機能に必要である場合、配列内の部位のシフトが潜在的に発生する可能性があります。
このような修飾は、中和抗体による認識と結合に悪影響を及ぼし、回避を促進する可能性があります。
我々はまた、BA.2.86 について、N354 の新しいグリコシル化部位が領域 1 の安定性に効果的に寄与し、同時にシールドを大幅に向上できることを示しました。
さらに、特定の VoC は、領域 1 と RBD 上の N370 グリカン結合溝の一部である RBM の間の接合部に位置する結合部位と一致する傾向で、モノシアル化ガングリオシド オリゴ糖に対する親和性を失ったことを示します。
この結論は、MD の結果と一致して、N343 グリコシル化の喪失により GM1os および GM2os に対する結合親和性がどのように変化するかによってさらに裏付けられます。
これに加えて、突然変異として、モノシアル化ガングリオシドオリゴ糖への結合が弱まることを確認しました。 また、さらなる変異によって親和性が再びオンになったり、N370 クレフト内で依然として認識できる他のグリカンに対する RBD の優先順位のシフトが決定される可能性もあります。
この分野ではさらなる作業が進行中です。
最後に、この研究の結果は、タンパク質の構造と安定性におけるN-グリコシル化の影響を理解することの重要性を示しており、これはCOVID-19ワクチンの設計に即座に影響を及ぼします。
実際、初期の研究では、SARS-CoV-2 S ベースのタンパク質ワクチンが N-グリカンの除去により有効性が向上したことが示されています。 使用中および開発中の RBD ベースのワクチンについては、N-グリカンの有無にかかわらず設計できます。
このような構築物の設計には、どの N-グリコシル化部位が構造的に必須で、どの部位が不要であるかを理解することが有益となる可能性があります。
これに加えて、我々の結果は、特定のタンパク質配列の文脈におけるタンパク質の構造安定性と動態に対するN-グリコシル化への影響を考慮することが、RBD残基間のエピスタティック相互作用を理解する鍵となる可能性があることを示しています。それはそれで難しいことですが、 不可能ではない場合でも、解読することは可能です。
以下省略。
この記事が気に入ったらサポートをしてみませんか?
