XBB株のスパイク構造、立体構造、抗原性、および受容体認識(2024年3月)
SARS-CoV-2 Omicron XBB lineage spike structures, conformations, antigenicity, and receptor recognition
SARS-CoV-2 Omicron XBB 系統のスパイク構造、立体構造、抗原性、および受容体認識
Summary
XBB と名付けられた SARS-CoV-2 オミクロン変異体の組換え系統は 2022 年後半に出現し、子孫を進化させ、地域および世界の人口を次々と席巻しました。
XBB 系統のメンバーは、免疫回避性と伝染性が向上していることで注目されました。
ここでは、XBB.1.5、XBB.1.16、EG.5、および EG.5.1 スパイク (S) 外部ドメインの低温 EM 構造を決定し、強化された 3-RBD ダウン受容体がアクセスできない閉鎖状態を明らかにします。これは、BA.1 および BA.2 で以前に観察されたプロトマー間受容体結合ドメイン (RBD) 相互作用によって媒介されます。
XBB.1.5 および XBB.1.16 RBD の安定性の向上により、初期の Omicron 変異による安定性の損失が補われましたが、F456L 置換により EG.5 RBD の安定性が低下しました。
S1 サブユニットの変異は、S2 サブユニットの立体構造とエピトープ提示に広範囲の影響を及ぼしました。
我々の結果は、安定性、受容体結合、免疫回避を含む複数のパラメータの同時最適化によるSタンパク質の継続的な進化と、Sタンパク質の立体構造の変化における比較的少数の残基置換の劇的な効果を明らかにしました。
Introduction
SARS-CoV-2 オミクロン B.1.1.529 変異株は、2021 年 11 月に発見されて以来、以前の懸念される変異株 (VOC) と比較して、スパイク (S) タンパク質の変異数が多いことで注目されてきました。 変異を続け、一連の世界的な優位性の変化を経て多くの亜系統に多様化しました(図S1)。
2022 年後半に同定された、高度に多様化した 2 つの BA.2 亜系統の組換え体である XBB 系統は、SARS-CoV-2 変異株が置換ではなく組換えによって適応度を高めた最初の文書化された例となってます。
XBB.1 は、XBB 系統の初期拡大を引き起こした初期の XBB 系統メンバーであり、ワクチン接種または以前の Omicron 亜変異体のブレイクスルー感染によって誘導される抗ウイルス液性免疫に対して顕著な耐性を示しました。 その免疫抵抗特性は、複数の受容体結合ドメイン (RBD) 置換と、繰り返し観察され、もともとアルファ VOC で注目されていた Y144del N 末端ドメイン (NTD) 変異に起因すると考えられます。
XBB.1 はシュードウイルス感染力アッセイにおいて BA.2 よりも感染力が高く、その RBD は BA.2 RBD よりも強く ACE2 に結合しました。
BA.2 および BA.2.75 よりも著しく融合性が高い一方で、XBB.1 の病原性は BA.2.75 に匹敵し、デルタ株よりも低かった。
RBD に追加の F486P 置換を含む XBB.1 の子孫である XBB.1.5 は、2022 年 10 月頃に特定されました。
XBB.1.5 は、以前は最も普及していた Omicron BA.5 の子孫である BQ.1.1 亜種を上回り、世界中で最も普及した系統になりました (図 S1)。
XBB.1.5 は 2023 年半ばまでに衰退し始めたため、XBB.1.16 と追加のマイナー リネージの複雑な配列がそれに置き換わりました。
2023 年 7 月頃、EG.5 はアジアと北米の一部の国で急速に普及し始めました。
EG.5 の実効再生産数 (R0) は、XBB.1.9.2 (XBB.1.5 と同じ S タンパク質アミノ酸配列を持つ親)、XBB.1.5、および XBB.1.16 よりも高く、免疫回避特性が強化されたため、世界的な蔓延に貢献したと考えられています。
EG.5 の直接の子孫である EG.5.1 は、NTD 内にさらに 1 つの Q52H 置換を持ち、最も急速に拡散した EG.5 亜系統でした。
配列類似性から予想されるように、XBB 系統メンバー、XBB.1.5、XBB.1.16、および EG.5 の S タンパク質は、 系統樹上にクラスターを形成し、他の VOC やその親 BA.2 とは区別されます (図 S2)。
ワクチンの評価と製造に必要なリードタイムを考慮すると、 2023 年春の主要なバリアントである XBB.1.5 は、2023 年の秋に利用可能になるワクチン更新プログラム (ファイザー、モデルナ、ノババックス) の一価ブースターとして使用される S バリアントとして選択されました。
初期の研究では、ブースターが現在流行している変異種に対して保護効果があることが示されています。
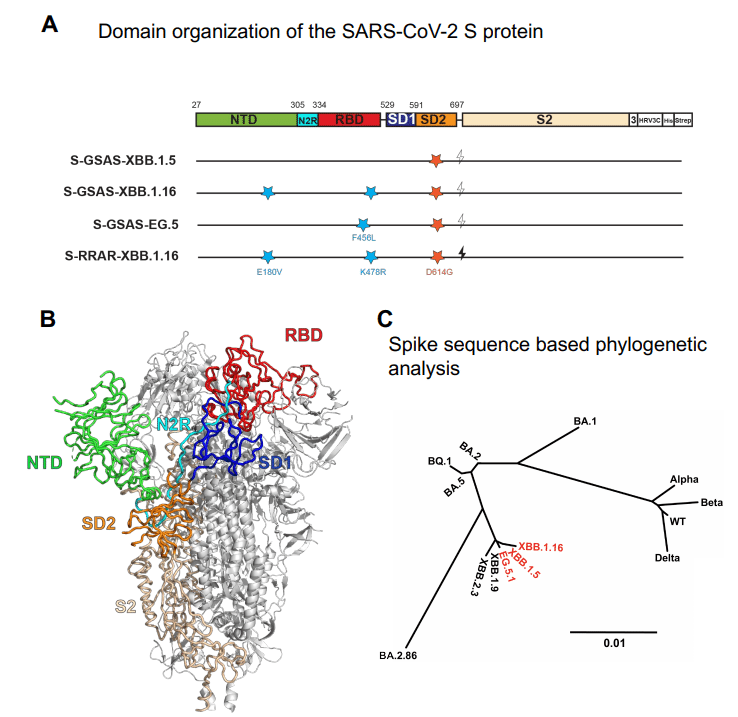
S1 サブユニットと S2 サブユニットで構成される SARS-CoV-2 表面の S タンパク質は、その受容体結合と免疫回避特性の主な決定因子です (図 S2)。
S1 サブユニットは受容体結合を担当し、S タンパク質の融合前立体構造において融合サブユニット S2 の上にキャップを形成します。
S1 NTD サブユニット (残基 27 ~ 305) はリンカー (N2R; 残基 306 ~ 334) を介して RBD (残基 335 ~ 521) に接続されています。続いてサブドメイン 1 (SD1; 残基 529 ~ 591) 領域とサブドメイン 2 (SD2; 残基 592 ~ 697) 領域が続きます。
RBD には、ACE2 や TMEM106B などの宿主受容体に結合する受容体結合モチーフ (RBM; 残基 483 ~ 506) が含まれています。
RBDは、受容体がアクセスできない「ダウン」:閉じた立体構造、または受容体がアクセスできる「アップ」:開いた立体構造のいずれかを採用します。
SD2 には、S1 サブユニットと S2 サブユニットの間の境界を定義するフューリン切断部位が存在します。
別の切断部位 S2’ は S2 サブユニットにあります。
受容体結合と S タンパク質のタンパク質分解プロセシングに続いて、S2 サブユニットは S1 の分離時に再構成され、疎水性融合ペプチドの宿主膜への挿入と S2 ヘリックスの融合後立体構造への移行を可能にし、細胞侵入につながります。
初期の オミクロン株 BA.1 および BA.2 は、受容体がアクセスできないダウン状態で RBD-RBD インタープロトマー接触部に安定化変異を蓄積しました。 その結果、RBD間の直接相互作用またはプロトマー間RBD相互作用に関与する構造的特徴の安定化によって、受容体がアクセスできない3-RBDダウン状態をより多く占める方向へSタンパク質集団がシフトします。
これらの変異は、XBB.1.5、XBB.1.16、および EG.5 S タンパク質に保持されています (図 1A および S2)。
これらの進化した Omicron サブバリアントで S タンパク質の構造がどのように変化したかを理解するために、ここでは、クライオ EM 構造を決定し、XBB.1.5 の受容体結合、抗原性、安定性を研究します。プロリン安定化変異を組み込むことなく調製された、以前に確立された S-GSAS-D614G (非切断) または S-RRAR-D614G (フリン切断) プラットフォームを使用した、密接に関連する XBB.1.16 および EG.5及びEG.5.1 S 外部ドメイン、 各変異体の RBD のみを発現する構築物も同様に研究します(図 S2、データ S1 および S2)。
Results
Results
Thermostability of the XBB.1.5, XBB.1.16 and EG5 RBD
XBB.1.5、XBB.1.16、および EG5 RBD の熱安定性
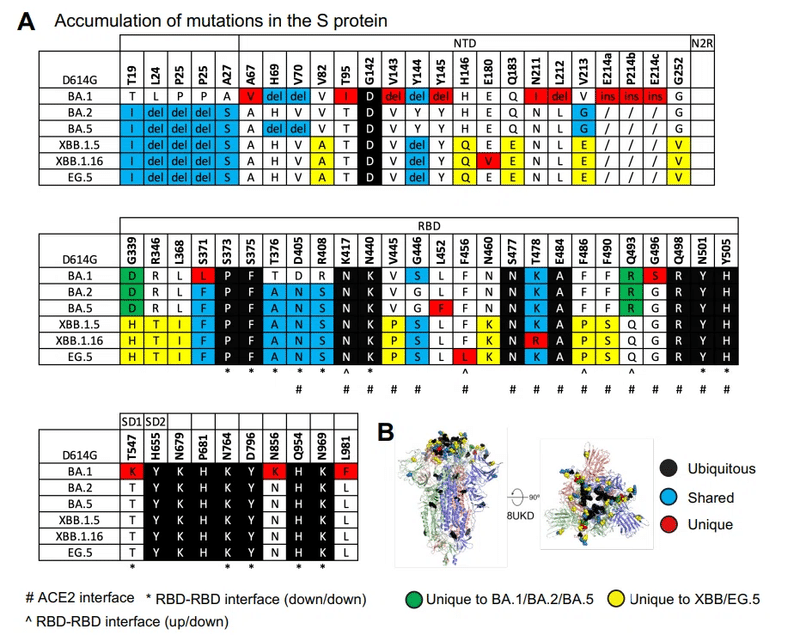
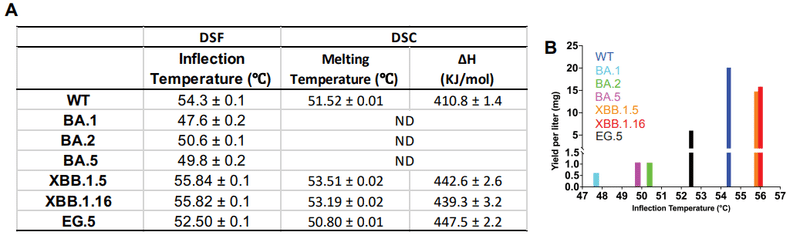
元の Omicron 系統 BA.1、BA.2、および BA.5 と比較した XBB.1.5、XBB.1.16、EG.5、および EG.5.1 S タンパク質の新しい変異は、NTD および RBD に位置します。いくつかはACE2相互作用部位に現れます(図1AおよびB)。
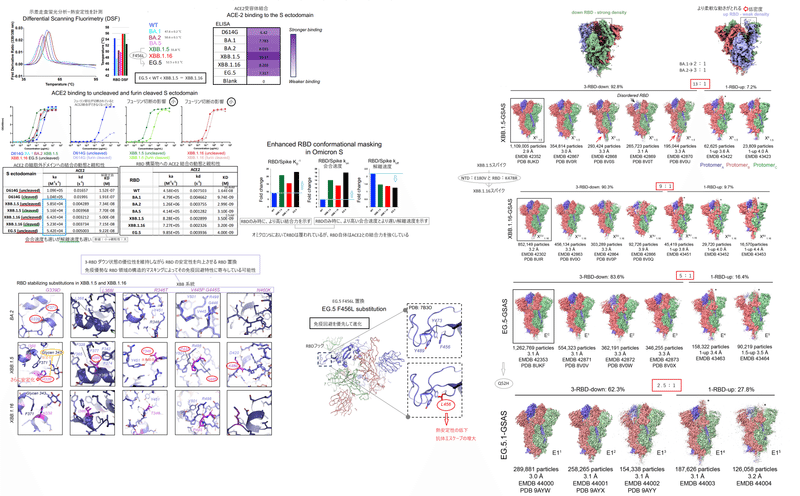
我々は以前、WT RBDと比較してBA.1 RBDの安定性が劇的に低下し、その後のBA.2 RBD変異によりRBDの安定性が部分的に回復することを報告しました。
追加で獲得した RBD 変異がドメインの安定性に及ぼす影響を評価するために、サンプルに熱ランプを適用した際のタンパク質固有蛍光の変化を測定する示差走査蛍光分析 (DSF) アッセイを使用しました (図 1C および S3)。
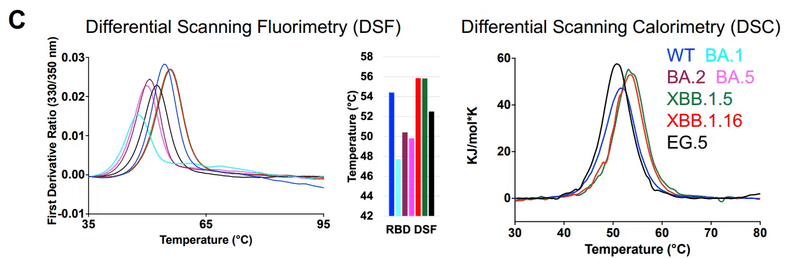
BA.1 RBD は、以前のレポートと同様に、47.6 ± 0.2 °C の変曲点 (Ti) を示し、WT RBD よりも約 7 °C 低くなりました。
この熱安定性の損失は、BA.2 RBD (Ti 50.6 ± 0.1 °C) では部分的に回復しました。
XBB.1.5 は、Ti が約 55.8 °C の WT RBD よりも高い熱安定性を示しました。
したがって、XBB.1.5 で獲得された突然変異は、初期のオミクロン株で観察された熱安定性の低下を回復しただけでなく、XBB.1.5 RBD は WT RBD の熱安定性も上回りました。
XBB.1.5 と XBB.1.16 の RBD は、478 位の単一残基置換が異なります。
オミクロン株BA.1 で発生した T478K 置換は、BA.2、BA.5、および XBB.1.5 では保持されましたが、XBB.1.16 ではアルギニンに置換されました (図 1A および S2)。
XBB.1.5 と XBB.1.16 RBD の DSF プロファイルはほぼ同一であったため (図 1C)、われわれは、XBB.1.5 と XBB.1.16 RBD の間で唯一異なる残基である K478R 置換は RBD の熱安定性に影響を及ぼさないと結論付けました。
EG.5 RBD は、52.5 ± 0.1 °C の Ti を示しました。これは、WT RBD の Ti よりも約 1.8 °C 低く、XBB.1.5 および XBB.1.16 RBD よりも約 3 °C 低かったです。 これは、XBB.1.5 と EG.5 の間の唯一の残基置換である F456L が、EG.5 RBD の安定性の低下に関与していることを示しています。
示差走査熱量測定 (DSC) を使用して測定された WT、XBB.1.5、XBB.1.6 および EG.5 RBD の融解温度 (Tm) は、DSF 実験で観察されたのと同じ熱安定性傾向、つまり EG.5 < WT < XBB.1.5 ~ XBB.1.16 を示しました (図 1C および S3A)。
最高のタンパク質収量は XBB.1.5 および XBB.1.16 RBD コンストラクトから得られ、どちらもテストしたパネルの中で最も熱安定性が高くなります。 一方、BA.1 RBD は熱安定性が最も低く、発現収率も最も低かった (図 S3B)。
要約すると、RBD の熱安定性は SARS-CoV-2 オミクロンの進化中に変化しました。
BA.1 RBD で観察された熱安定性の低下は、後続のバリエーションでは徐々に回復しました。
XBB.1.5 および XBB.1.16 RBD は WT RBD よりも熱安定性が高かったのに対し、EG.5 は WT RBD と比較して熱安定性の低下を示しましたが、依然として BA.1、BA.2、および BA.5 RBD よりも熱安定性が高いままでした。
RBD の安定化は、RBD と宿主受容体とのより生産的な出会いをもたらし、それによってウイルスの伝播性を改善する可能性があります。
RBD は S タンパク質の最も免疫優勢な領域の 1 つであるため、その安定性はワクチンにも影響を与える可能性があり、S タンパク質と RBD のみのワクチン形式の両方において、安定性の向上によりより耐久性の高い免疫原が得られます。
ACE2 receptor binding of XBB lineage S protein ectodomains
XBB系統 Sタンパク質細胞外ドメインのACE2受容体結合
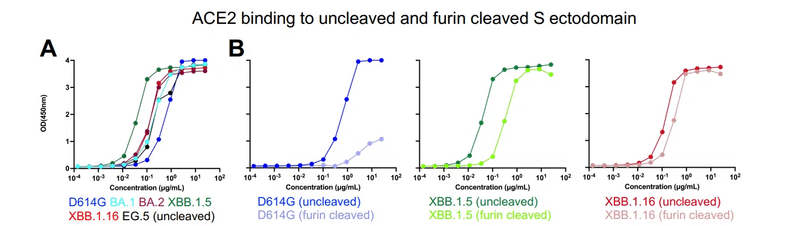
受容体結合に対する獲得変異の影響を評価するために、ELISAおよび表面プラズモン共鳴(SPR)によって、ACE2受容体外部ドメインへのSタンパク質外部ドメインの結合を測定しました(図1D、2A〜GおよびS3C)。
以前の研究に基づいて予想されたように、RBD の ACE2 結合領域の広範な変異にもかかわらず、すべての S タンパク質は ACE2 への強力な結合を保持していました。
実際、試験したすべてのオミクロン S タンパク質の結合レベルは D614GS S タンパク質よりも高く、XBB.1.5 が最も強い ACE2 結合を示しました (図 1D および 2A)。
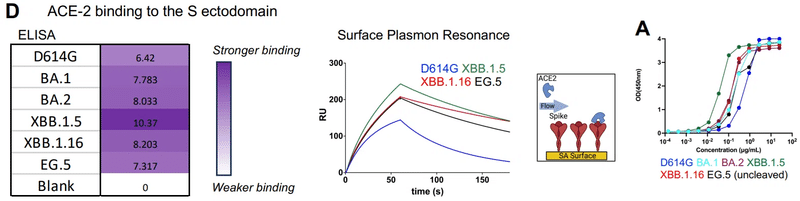
SPR を使用して、Twin-Strep タグを介してセンサー表面に S タンパク質を結合すると、 非切断 (S-GSAS) およびフリン切断 (S-RRAR) D614G スパイクについて、それぞれ 152 nM および 192 nM の ACE2 結合親和性 (または解離定数; KD) を測定しました (図 2F-G)。
XBB.1.5、XBB.1.16、および EG.5 の未切断の細胞外ドメインに対する ACE2 親和性は、それぞれ 73 nM、50 nM、および 92 nM でした。 一方、XBB.1.5 および XBB.1.16 のフューリン切断 S 外部ドメインの ACE2 親和性は、それぞれ 74 nM および 71 nM でした。
EG.5 および EG.5.1 S 細胞外ドメインは、同様の親和性および反応速度で ACE2 に結合しました (図 S3E)。
D614G と比較して、XBB.1.5、XBB.1.16、EG.5および EG.5.1 S 細胞外ドメインの ACE2 親和性がより強固であるのは、解離速度 (koff) が遅いためであり、会合速度 (kon) の低下も補われました。 (図 1D、2C、および 2F ~ G)。
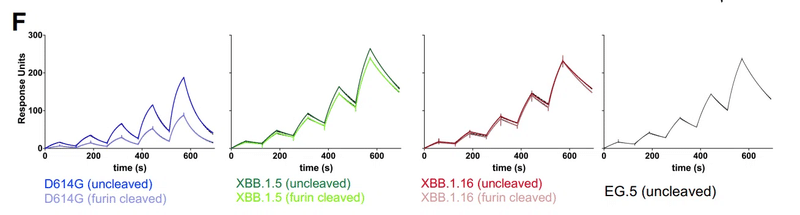
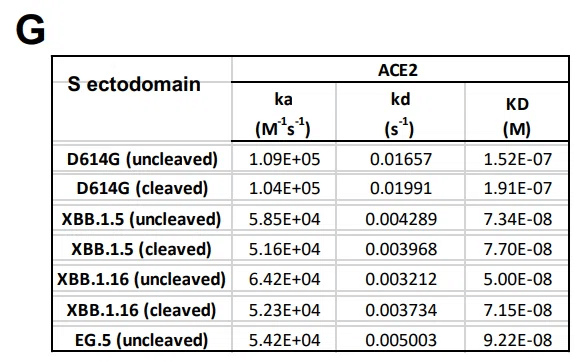
ACE2 結合親和性と反応速度は、D614G、XBB.1.5、および XBB.1.16 S 細胞外ドメインの未切断バージョンとフリン切断バージョンで類似していました。
D614G バリアントでは、同様の ACE2 結合親和性と反応速度にも関わらず、フューリン切断 S タンパク質と非切断 S タンパク質の間で実質的かつ再現性のある ACE2 結合レベルの差が観察されました。
未切断の D614GS S タンパク質は、フリンで切断された D614GS S タンパク質と比較して、より高いレベルの ACE2 結合を示しました。これは、未切断のバージョンと比較して、フューリンで切断された S タンパク質では ACE2 がアクセス可能な「アップ」RBD の利用可能性が低下していることを示唆しています。
この観察は、未切断のS外部ドメインと比較して、フリン切断D614G S におけるRBDアップ集団の減少に関する我々の以前の報告と一致します。
対照的に、XBB.1.5 および XBB.1.16 S タンパク質のフリン切断バージョンと非切断バージョンでは、SPR によって結合レベルの差ははるかに小さく測定されました。これは、XBB.1.5 および XBB.1.16 S タンパク質における RBD のup/downの性質が、D614GS S タンパク質と比較してフリン切断に対する感受性が低いことを示唆しています。
切断されていないS外部ドメインとフューリン切断されたS外部ドメインとの間のこれらの差異は、ELISAによって測定されたACE2結合レベルにも反映されていた(図2A、BおよびE)。
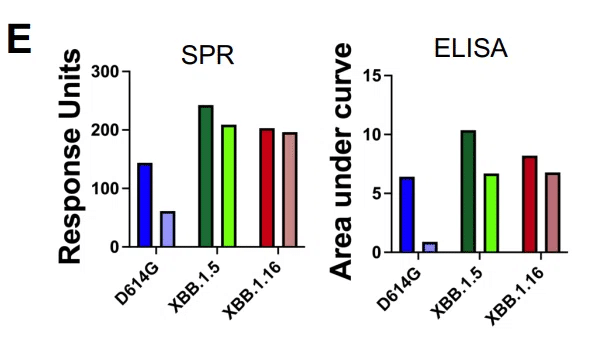
要約すると、公開されたレポートと一致して、我々のデータは、XBB.1.5、XBB.1.16、EG.5およびEG.5.1バリアントのSARS-CoV-2 S外部ドメインに対するACE2外部ドメインの結合親和性がD614Gよりも高いことを実証し、これは解離速度の低下に寄与しました。
さらに、フューリン切断対非切断 S 外部ドメインで観察された ACE2 結合レベルの差は、XBB.1.5 および XBB.1.16 スパイクのドメイン性質に対するフューリン切断の影響が D614G スパイクよりも小さいことを示唆しました。
ACE2 receptor binding of XBB lineage RBDs
XBB系統RBDのACE2受容体結合
S タンパク質の文脈内では、RBD は構造マスキングの影響を受けます。 そして、RBMが露出されて宿主受容体に結合できる「アップ」または受容体アクセス可能な状態、またはRBMが構造的にマスクされている受容体アクセス不可能な「ダウン」状態をとることができます。
これらの立体構造上の制約なしにRBDへのACE2の固有の結合を評価するために、D614G (または WT)、BA.1、BA.2、BA.5、XBB.1.5、XBB.1.16、および EG.5/EG.5.1 S タンパク質に由来する RBD コンストラクトのパネルに対する ACE2 の結合を測定しました (図2H〜IおよびS3C)。
WT および XBB.1.5 RBD は、それぞれ ~16.4 nM および 3.5 nM の親和性で ACE2 に結合しました。これは、公開された研究とほぼ一致しています。
オミクロンRBD に対する ACE2 親和性は、WT RBD に対する親和性よりも強く、BA.1 RBD の結合は約 1.7 倍強く、他の オミクロン RBD コンストラクトの結合は、WT RBD と比較して約 4 ~ 5.5 倍強く結合しました。
RBD と S 外腺細胞の両方をテストしたバリアントでは、対応する S タンパク質よりも RBD 構築物の ACE2 親和性の向上が観察されました。 S外部ドメイン親和性に対するRBD親和性の変化倍数は、D614G変異体と比較して、Omicron XBB.1.5、XBB.1.16およびEG.5変異体の方が高かった(図2I~J)。
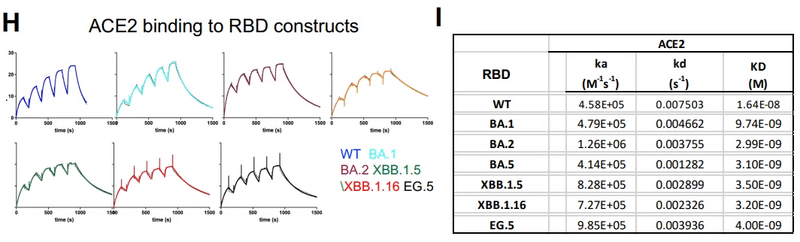
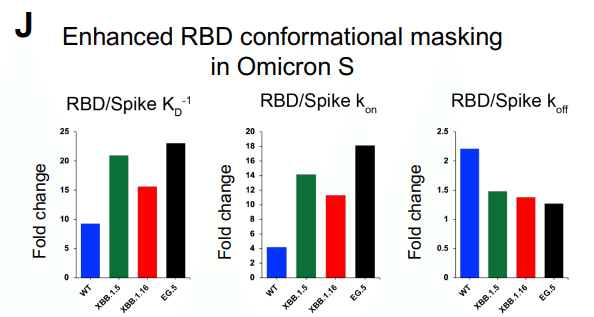
より高い会合率(kon)は、RBD のみの構築物における ACE2 親和性の強化の主な原因でした。 これは、RBD が S タンパク質の一部である場合、生産的な ACE2-RBM との遭遇が減少することと一致しています。
RBD のみの構築物と S 外部ドメインの間の ACE2 結合親和性と反応速度の違いは、S タンパク質との関連で RBM が受ける立体構造マスキングの尺度です。
我々のデータは、RBM の立体構造マスキングとシールドが WT RBD よりも オミクロンRBD の方が優れていることを示しています。
したがって、 D614G S タンパク質と比較した Omicron S タンパク質の ACE2 結合親和性の増加(図 2G)は、Omicron S タンパク質における RBM の立体構造マスキングの強化に対抗する、Omicron RBD に対する ACE2 の固有の親和性の増加によって達成されました (図 2H ~ J)。
Cryo-EM structures of XBB lineage S ectodomains
XBB 系統の細胞外部ドメインのクライオ EM 構造
それらの構造と立体構造を視覚化するために、XBB.1.5、XBB.1.16、EG.5、および EG.5.1 の S タンパク質細胞外ドメインの低温 EM 構造を決定しました (図 3-5、データ S3-S4 および表 S1)。我々の S-GSAS プラットフォームを利用します (図 S2 およびデータ S1)。
XBB.1.16 については、S-RRAR-XBB.1.16 の構造も決定しました。この構築物は、S1 サブユニットと S2 サブユニットの間に天然のフューリン切断部位を含み、タンパク質発現中に S タンパク質がフューリンで切断される構造です (図 4、S2、データ S1、S3、S4、および表 S1)。
Cryo-EM structures of the XBB.1.5 S protein ectodomain
XBB.1.5 S タンパク質外部ドメインのクライオ EM 構造
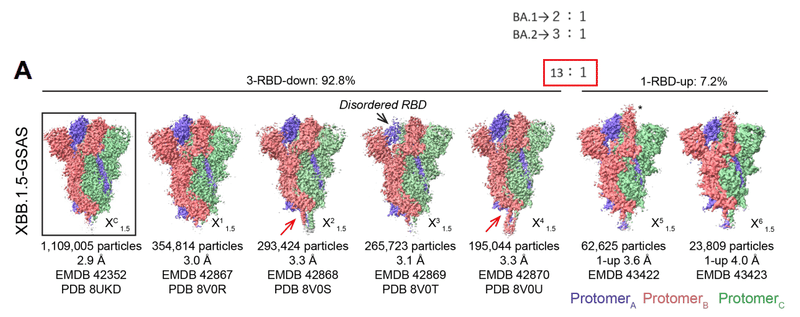
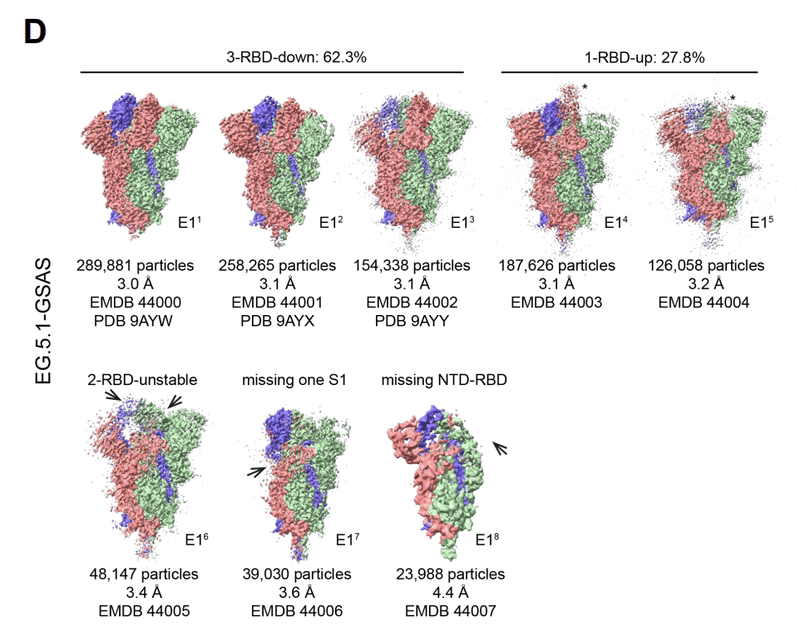
S-GSAS-XBB.1.5 (図 3、データ S3 ~ S4 および表 S1) については、私たちは、3-RBD-down (閉じた) および 1-RBD-up (開いた) 外部ドメイン粒子集団を、閉じたものと開いたものの比率がおよそ 13:1 であることを確認しました。 これは、BA.2 S と BA.1 S でそれぞれ観察されたクローズド S とオープン S の比率 3:1 および 2:1 よりも大幅に高かった。
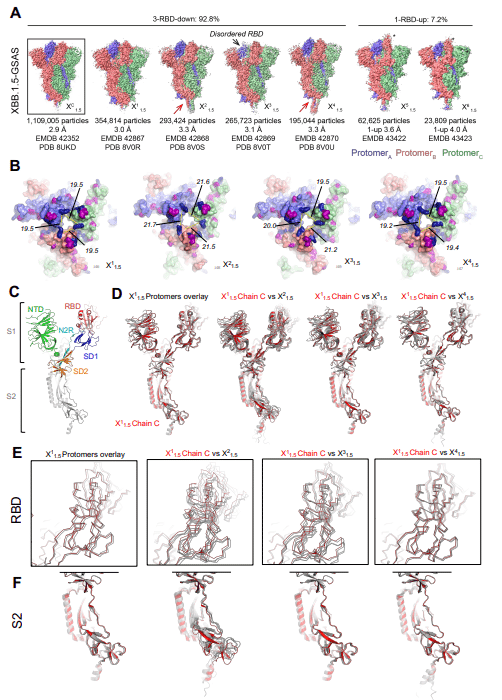
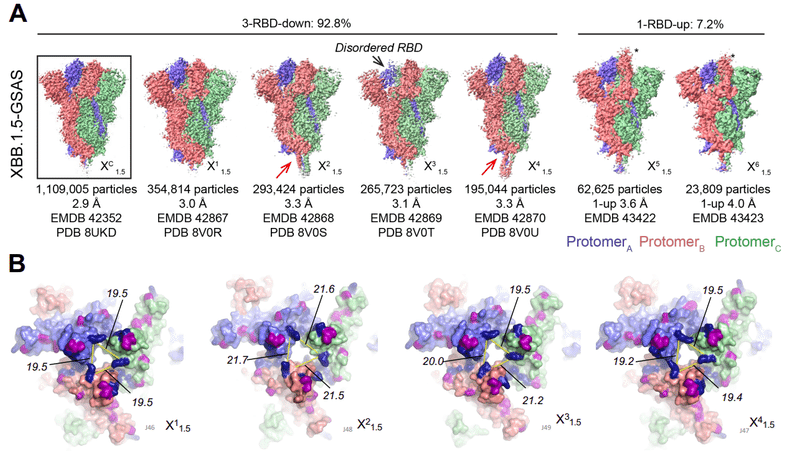
優勢な 3-RBD-down 集団は、ここでは XDC1.5 と名付けられた 2.9 Å の再構築をもたらしました (PDB: 8UKD; EMDB: 42352) (図 3A)。
さらに細分類すると、3-RBD-down 集団と 1-RBD-up 集団についてそれぞれ 4 クラスと 2 クラスが得られました。
これらは X1-1.5 (PDB: 8V0R、EMDB: 42867) 、X2-1.5 (PDB: 8V0S、EMDB: 42868)、X3-1.5 (PDB: 8V0T、EMDB: 42869)、X4-1.5 (PDB: 8V0U、EMDB: 42870)、X5-1.5 (EMDB: 43422)、X6-1. 5 (EMDB: 43423)と指定されました。
XBB.1 S タンパク質の低温 EM 構造決定を報告する研究では、S タンパク質の 3-RBD ダウンの閉じた状態のみが低温 EM データセットで特定されました。これは、3-RBD-down 閉鎖状態を採用する強い傾向を示唆しています。
実際、この研究では当初、クライオ EM データ分析では XBB.1.5 外部ドメインのクライオ EM データセットの 3-RBD-down 状態のみが得られ、1-RBD-up 状態は広範な分類によって明らかになりました。
コンセンサス 1-RBD-up 集団 XUC1.5 は、密度が欠落しているか密度が低いことを特徴とする高度に無秩序な up RBD を示しました (図 S4)。
XUC1.5 集団の障害を解決するためにさらに分類すると、ここでは X5-1.5 および X6-1.5 と名付けられた 2 つのサブクラスが、主に上向き RBD の位置によって異なることが明らかになりました。X6-1.5 の RBD は、中心三量体軸により傾いた中間構造を採用し、隣接する 2 つのプロトマーからの RBD および NTD との接触を維持します。X5-1.5 では、アップ RBD は三量体の中心軸から一方向に傾いたより直立した構造を採用しました。そして、隣接する RBD に向かって、隣接する NTD から遠ざかる直交方向に横に傾いています(図 3A および S4)。
したがって、コンセンサス構造における無秩序または欠落した「アップ」RBD 密度は、柔軟性の結果でした。RBD は、集団内の S タンパク質分子内の標準的な「アップ」配置を中心にさまざまな性質を採用しています。
4つの XBB.1.5 3-RBD-down S タンパク質サブクラス間では、明らかな違いが識別できました (図 3A ~ B)。
X1-1.5 サブクラスは最大数の粒子で構成され、コンセンサス構造 XDC1.5 に最もよく似ていました。2 つの構造間の全体的な C アルファ二乗平均平方根偏差 (Cα RMSD) は 0.26 Å です。
最も多様な 3-RBD-down サブクラスは X2-1.5 で、XDC1.5 および X1-1.5 と比較して、全体の Cα RMSD がそれぞれ 2.16 Å および 2.26 Å でした。
他の 2 つの 3-RBD-down 亜集団 X3-1.5 および X4-1.5 は、コンセンサス XDC1.5 と比較して、それぞれ 0.32 Å および 0.37 Å の全体的な Cα RMSD を示しました。X1-1.5 サブクラスと比較した場合は、それぞれ 0.19 Å と 0.22 Å です。
全体的な Cα RMSD には反映されていませんが、X3-1.5 集団と X4-1.5 集団を相互に、また他のサブクラスと区別する局所的な差異が観察されました。
X3-1.5 では、3 つの RBD のうち 1 つのクライオ EM マップ密度が他の 2 つよりも弱く見えました。RBD の位置にばらつきがあることを示唆しています。
X2-1.5 および X4-1.5 の C 末端の残基は、通常の外部ドメイン構造よりも規則的でした。これらは、SARS-CoV-2 スパイクのクライオ ET 構造で説明されているヒンジ動作を彷彿とさせる方法で、スパイクの本体に対してある角度でヒンジで取り付けられていました。
3-RBD-down サブクラス間で観察された顕著な違いは、RBD の相対的性質にありました。サブクラス X1-1.5 および X4-1.5 の 2 つの RBD は、他の 2 つのサブクラス (X2-1.5 および X3-1.5) よりもコンパクトにパックされています (図 3B)。
RBD間の分離は、各構造の異なるプロトマーにおける残基K440のCα原子間の距離を測定することによって定量化されました。
X1-1.5 では 3 つの距離すべてが 19.5 Å でした。X4-1.5 の場合は 19.2 ~ 19.5 Å です。
これらの距離は、X2-1.5 で 21.5 ~ 21.7 Å の間で測定されました。 X3-1.5 の場合は 19.5 ~ 21.2 Å です。
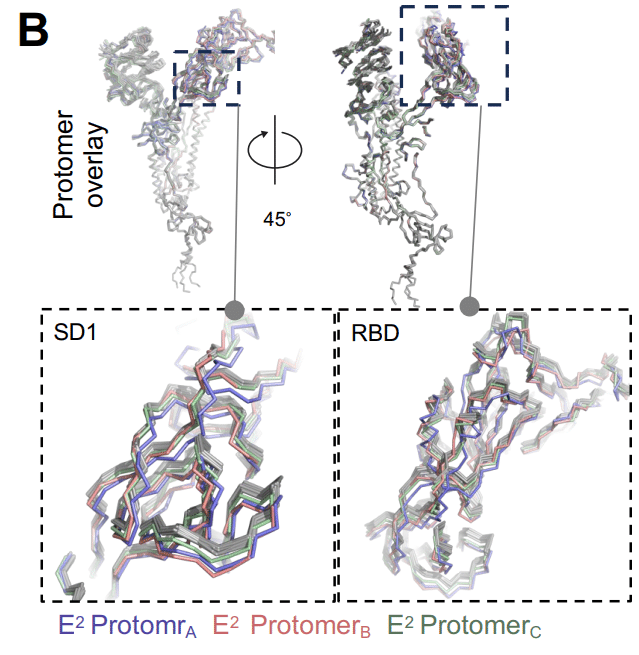
SD2 サブドメインが重なった X1-1.5 の 3 つのプロトマーは、NTD 領域で予想されるわずかな変化を除いて、全体にわたって良好な重複を示しました (図 3C および D)。これは、再構成中に対称性が適用されていないにもかかわらず、ほぼ対称的な構造を示しています。
次に、X1-1.5 プロトマーの 1 つを、それぞれ X2-1.5、X3-1.5、および X4-1.5 の 3 つのプロトマーとオーバーレイしました。
3 つの X4-1.5 プロトマーの RBD は、X1-1.5 の RBD とよく重なっていました (図 3D および E)。
プロトマー間の RBD 位置の最も大きな相違は X2-1.5 で見られ、次に X3-1.5 が続きました。
X3-1.5 では、RBD の位置は分岐していましたが、異なるプロトマーの S2 領域は相互に、また X1-1.5 の S2 とよく重なっていました。一方、X2-1.5 構造では、それ自体のプロトマー間および X1-1.5 プロトマーとの両方で、S2 領域の違いが全体にわたって見られました。
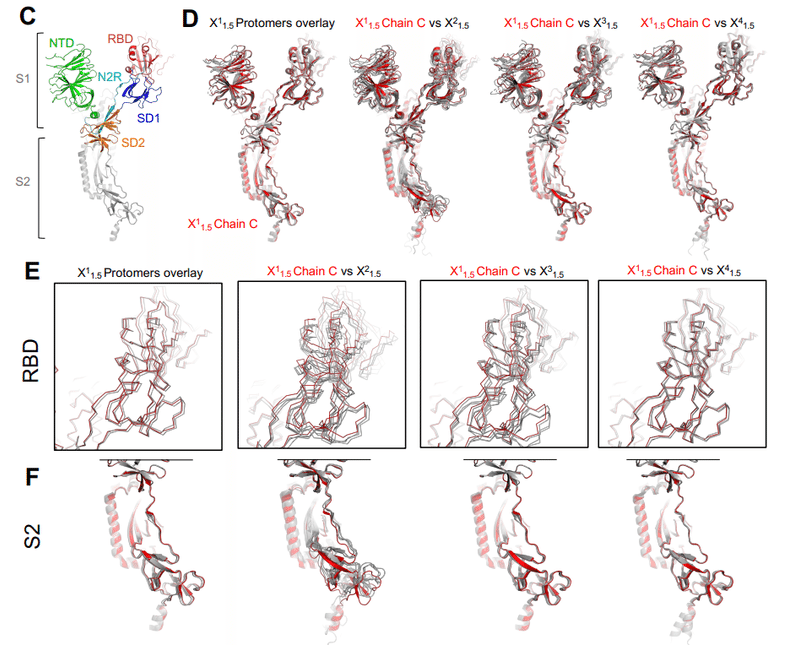
要約すると、XBB.1.5 外部ドメインのクライオ EM データセットは、RBD の性質に観察される立体構造の多様性を伴う、その優勢な 3-RBD ダウン集団のかなりの多様性を明らかにしました。 同様に、典型的には不変の S2 サブユニットを含む S タンパク質の他の領域にも多様性は存在します。
Cryo-EM structures of the XBB.1.16 S protein ectodomain
XBB.1.16 S タンパク質外部ドメインのクライオ EM 構造
XBB.1.16 S タンパク質は、NTD の E180V と RBD の K478R という 2 つの残基置換によって XBB.1.5 S タンパク質と異なります (図 1A および S2)。
XBB.1.5 および XBB.1.16 のベースライン フォームは、XBB.1 と同様に体液性免疫および治療用抗体を回避する能力を示しました。ただし、両方とも XBB.1 と比較してシュードウイルス アッセイにおいて感染力の増加を示し、XBB.1.16 は XBB.1 および XBB.1.5 と比較してヒト集団におけるより大きな増殖利点を示しました。
S-GSAS-XBB.1.16 クライオ EM データセット (図 4、データ S3 ~ S4、表 S1) では、3-RBD-down と 1-RBD-up S 集団が約 9:1 の比率で特定されました。
また、フーリンで切断された S-RRAR-XBB.1.16 コンストラクトの構造も決定し、閉じた S 構造と開いた S 構造の比率が約 14:1 であることが観察されました。
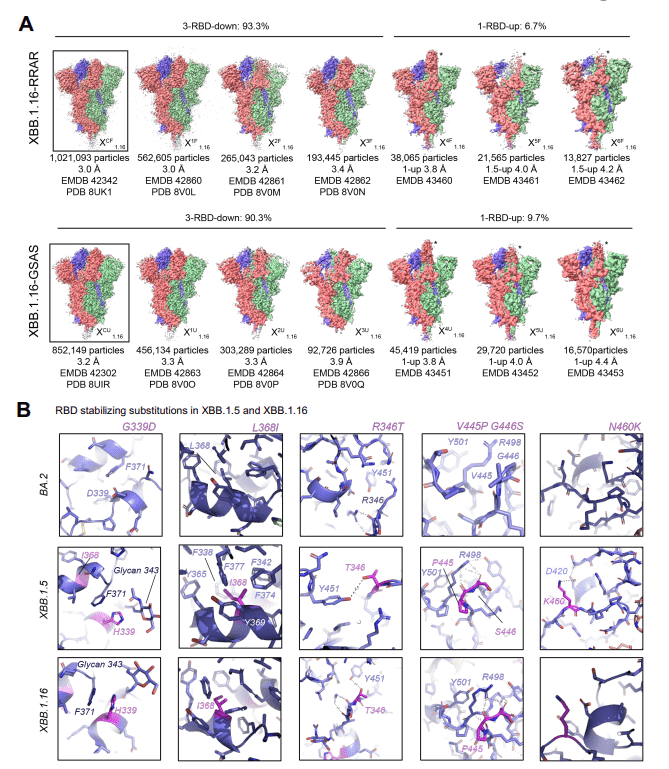
切断されていない S-GSAS-XBB.1.16 外部ドメインの 1-RBD-up 集団は、up RBD の性質に予想される変動性を示しました。これは、X4-1.16、X5-1.16、および X6-1.16 という名前の 3 つのサブクラスでサンプリングされました。
フューリンで切断された S-RRAR-XBB.1.16 細胞外部ドメインのクライオ EM データセットでは、私たちは、1-RBD-up S細胞外部ドメインだけでなく、2番目のRBDが部分的に上の位置に出現する集団も特定しました。これは、フューリンで切断された S タンパク質が、RBD アップまたはオープン状態において、切断されていない XBB.1.16 S タンパク質よりも柔軟性がある可能性があることを示唆しています。
XBB.1.16 S外部ドメインの未切断バージョンとフリン切断バージョンの3-RBDダウン構造は類似しており、全体のCα RMSDは0.3 Åでした。
3-RBD-down 集団は、XBB.1.16 S 外部ドメインの非切断データセットとフリン切断データセットごとに 3 つの集団に下位分類されました。
XBB.1.5 S タンパク質で観察されたものと比較すると、集団はより均質であり、サブクラス間の差異はわずかでした。すべての XBB.1.16 構造間の全体的な Cα RMSD は 0.20 ~ 0.40 Å の範囲にあります。
F486P 置換により、ACE2 結合が強化され、伝達性が高まり、中和抗体からの回避が促進されました。
デルタ VOC およびラムダ VOI で以前に観察された RBM の別の置換 (残基 483 ~ 506) である F490S は、中和抗体に対する耐性に関与していました。
XBB.1.5 および XBB.1.16 S 外部ドメインについては、BA.2 S タンパク質について以前に報告した RBD-RBD ダウン状態パッキングを観察しました (図 S5A)。
XBB.1.5 および XBB.1.16 S タンパク質の RBD-RBD ダウン状態プロトマー間接触部位では追加の変異は獲得されず、これらの部位で以前に獲得された変異はいずれも復帰または変化しませんでした (図 1A-B)。
私たちは、新しい RBD 変異が RBD 上の最も免疫優勢な部位を標的とすることで免疫回避を改善するか、または RBD 内の相互作用を促進することを発見しました。これにより、RBD の安定性が向上します (図 1A ~ C)。
Omicron BA.1 クレードと BA.2 クレードはどちらも G339D 置換を有しており、BA.2 では追加の S371F が出現し、それが後続の系統で保存されました。G339D 置換はプロトマー内 RBD 安定化部位に位置しており、BA.2 S371F 置換は 2 つの RBD 内のらせんストレッチ 339 ~ 342 および 367 ~ 371 の密な充填を促進します (図 4B)。
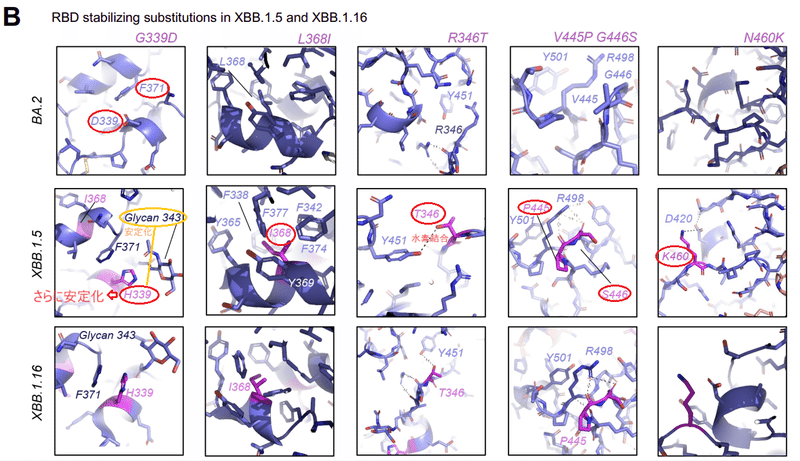
XBB.1.5 では、ヒスチジンが残基 339 に出現し、π-π スタッキング相互作用を介して F371 の側鎖に対してパッキングすることでこの領域をさらに安定化しました。したがって、この領域が RBD 安定化のホットスポットとして強調されます。
さらに、H339 は残基 N343 および N343 グリカンに対して積み重ねて水素結合するように配置され、ヘリックス内の安定性を高め、グリカン 343 を安定化および配向させました。
さらに、XBB.1.5 に見られる L368I 置換は、残基 F371、Y365、F377、F374、F342、F338、および V367 を含む芳香族/疎水性クラスターの中心の 367-371 螺旋ストレッチ内で発生します。
したがって、Omicronがプロトマーダウン状態のRBD-RBD接触を行いながらRBDを安定化するように進化するにつれて、S371F、G339H、およびL368Iの周囲の領域が最適化されているようです。
XBB.1.5 および XBB.1.16 の他の安定化変異には、RBD 表面ループの R346T 置換が含まれます。 これには、BQ.1.1、BA.4.6、BA.5.2.6、XBF、FU.1 などの拡張された複数の系統にも現れました。
346 位の側鎖をアルギニンからスレオニンに短くすると、ヒドロキシル基が Y451 側鎖ヒドロキシルとの水素結合距離内に収まります (図 4B)。
XBB.1.5 と XBB.1.16 では、2 つの連続する残基 V455P と G446S の置換が発生しました。ここで、G446S は BA.1 で取得され、BA.2 で G に戻り、XBB.1.5 で再び S に変更されました。
これらは一緒になって、Y501とπカチオンペアを形成することを以前に示した残基R498に対してパックする表面ループのターンを安定化します。どちらの残基も Q498R および N501Y 置換を介して Omicron BA.1 に取得され、後のバリアントにも保持されています。
R498-Y501 ペアは、ダウン状態の RBD 間のプロトマー間接触の安定化に寄与するループを安定化します。
N460K 置換は表面ループで発生し、D420 と塩橋を形成します。
要約すると、XBB 系統には、3-RBD ダウン状態の優位性を維持しながら RBD の安定性を向上させる RBD 置換が組み込まれています。これは、免疫優勢な RBD 領域の構造的マスキングによってその免疫回避特性に寄与している可能性があります。
Cryo-EM structures of EG.5 and EG.5.1 S ectodomains
EG.5 および EG.5.1 細胞外ドメインのクライオ EM 構造
EG.5 S タンパク質の構造と立体構造を視覚化するために、S-GSAS-EG.5 のクライオ EM データセットを取得しました (図 5、データ S3 ~ S4 および表 S1)。
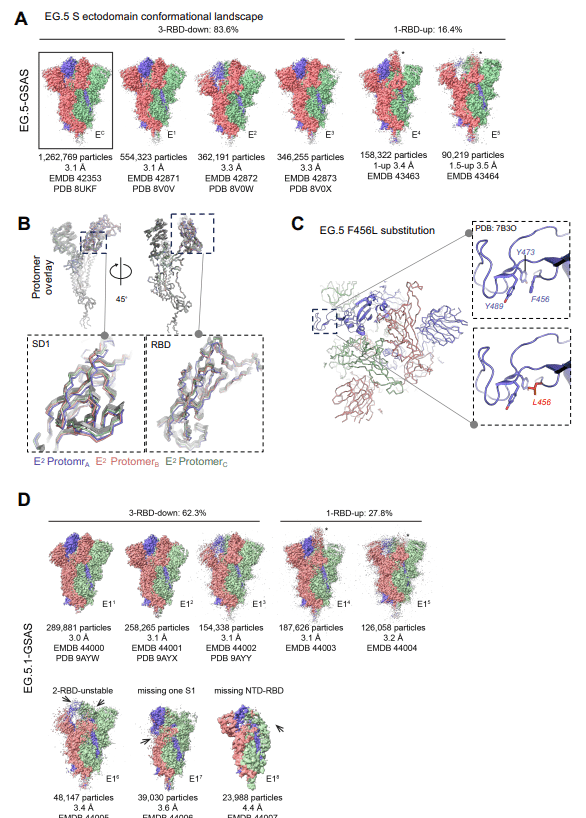
EG.5 には、RBD に位置する XBB.1.5 と比較して F456L 置換が 1 つあります (図 1A および S2)。
3-RBD-down 集団は依然として EG.5 S クライオ EM データセットで優勢ですが、RBD-up 集団に対するその割合 (約 5:1) は、XBB.1.5 および XBB.1.16 外部ドメインのクライオ EM データセットで観察されたものと比較して小さかった。
EG.5 S タンパク質 3-RBD ダウン コンセンサス構造 EC (PDB: 8UKF; EMDB: 42353) は、E1 (PDB: 8VOV; EMDB: 42871)、E2 (PDB: 8VOW; EMDB: 42872)、および E3 (PDB: 8VOX; EMDB: 42873) に下位分類されました。
3-RBD-down サブクラス間のかなりのばらつきが S1 サブユニットで観察されました。E1 は十分に詰め込まれた RBD で最も順序付けられており、E2 は最も小さくなっています (図 S5B)。
E2 の 3 つの RBD は非対称に配置され、さまざまな密度を示し、1 つの RBD とそれに接触する NTD は最も弱い密度を示し、高い無秩序性を示しました。
この再構成のガウス フィルター マップを視覚化すると、この RBD のダウン状態の割り当てが確認されました。
同様に、E3 では 1 つの RBD-NTD ペアが他の 2 つよりも乱れていましたが、E2 ほど乱れていませんでした。
3-RBD-down 構造のフィッティング座標を比較すると、最大数の粒子 E1 を持つサブクラスが、全体の Cα RMSD が 0.30 Å のコンセンサス構造 EC に最も類似していることがわかりました。続いて、EC と比較して、Cα RMSD がそれぞれ 0.48 Å および 0.64 Å の E3 と E2 が続きます。
すべての構造において、S2 領域は非常に類似しており、C 末端で何らかの立体構造の変動が観察された点を除いて、よく重なっていました (図 5B)。
E2 プロトマーを除くすべての S1 領域は、SARS-CoV-2 S タンパク質構造で通常見られる NTD 領域の位置変動を除けば、同様でした。
E2 構造では、プロトマーのうち 2 つが SD1 サブユニットと RBD の位置のシフトを示しました (図 5B)。
これらのシフトには、これらのプロトマーの RBD が中央の垂直三量体軸から離れて傾いていることが含まれます。まだ下位置にある間に、この RBD が傾き、接触している NTD に変化を引き起こします。
EG.5 クライオ EM 構造における EG.5 F456L 置換の周囲の領域の解像度が原子レベルの可視化を妨げたため、私たちは、F456L 置換によって引き起こされる効果の構造的基礎を理解するために、RBD (PDB: 7B3O) の結晶構造に依存しました (図 5C)。
残基F456は、そのフック状の外観から以前に命名されたRBDフックに隣接する領域に存在します。 ここで、F456 は残基 Y473 および Y489 を持つ芳香族クラスターの一部です。
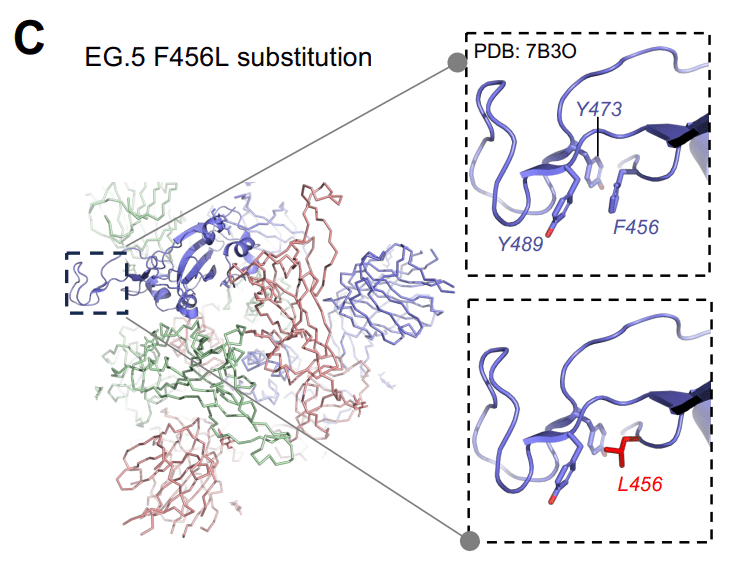
F456L 置換は、依然として疎水性であるにもかかわらず、フェニルアラニンをより嵩張らない非芳香族残基に置き換えることにより、この領域を不安定化します。
この領域は中和抗体の重要なエピトープであるため、F456L によって誘導される構造変化は、報告されているその免疫回避特性に寄与している可能性があります。
F456L 置換による F456/Y473/Y489 芳香族クラスターの不安定化は、XBB.1.5 と比較して EG.5 RBD の熱安定性が低下する構造的根拠を提供します。この変異は、これらの変異体の S タンパク質間で唯一異なる残基であるためです。
これは、もともと XBB.1.9 系統内から出現した EG.1 系統の間で保存されていましたが、XBB.1.16.6 亜系統でも独立して見つかりました。これは、EG.1 の導入前に北米で XBB.1.16 を置き換え始めていました。XBB.1.5 サブリネージ GK.1 (Pango alias XBB.1.5.70.1) および JD.1 (Pango エイリアス XBB.1.5.102.1) は、EG.1 が世界の他の地域で拡大していたように、南米でも有力なリネージとなりました (図S1)。
EG.5 は、NTD に単一残基置換 Q52H を取得して、最も急速に普及した亜系統である EG.5.1 になりました。
EG.5 と同様に、EG.5.1 S 外部ドメインのクライオ EM 構造 (図 5D および S5C) は、S1 サブユニット障害の亢進を示しました。RBD up 集団の割合が増加し、NTD および RBD 領域の一部が電子密度で見えなくなる程度に乱れた小さな粒子集団も発生しました。
この無秩序な個体群は、人間とミンクの間で感染したスパイクで観察された同様の無秩序な構造を彷彿とさせました。
まとめると、我々の結果は、1 つまたは 2 つの S1 サブユニット残基の置換が S タンパク質の構造にどのように局所的および全体的な大きな影響を与える可能性があるかを明らかにします。
Omicron の進行中の進化の文脈に置くと、私たちの結果は、さまざまな S タンパク質の特性が同時に最適化されている可能性が高いことを示しています。
したがって、Omicron の進化中に RBD の安定性が増加する傾向があるにもかかわらず、RBD を不安定化する EG.5 の単一残基置換が選択され、その後の亜系統の拡大に保持されました。 おそらく免疫優勢部位での免疫回避への寄与によるものと考えられます。
Antigenicity of the XBB lineage S proteins
XBB系統Sタンパク質の抗原性
抗原性に対する獲得変異の影響を評価するために、S タンパク質のさまざまな領域に結合するヒト抗 S 抗体のパネルへの結合を ELISA でテストしました。
これらには、S1 抗体 DH1050.1 (NTD 指向)、DH1042 (RBD ACE2 結合部位指向)、CR3022、DH1047 および S2X259 (RBD 内面指向)、DH1044、DH1193 および S309 (RBD 外面指向) が含まれます (図6A〜CおよびS6)。
また、S2 抗体 DH1058.1 および DH1294 (融合ペプチド指向)、DH1057.1 (ステムヘリックスエピトープ指向)、および 3 つの S2 グリカン部分の四次クラスターを標的とする Fab 二量体化グリカン反応性抗体のパネルも含めました (図6D、FおよびS8)。
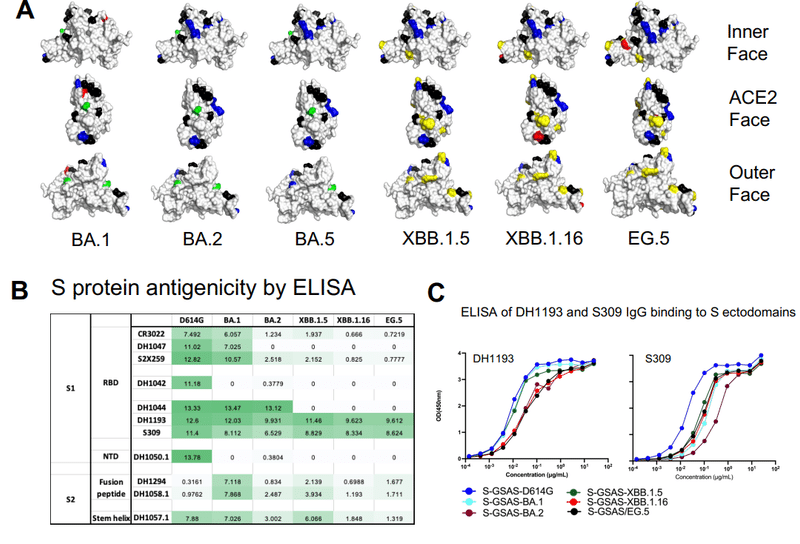
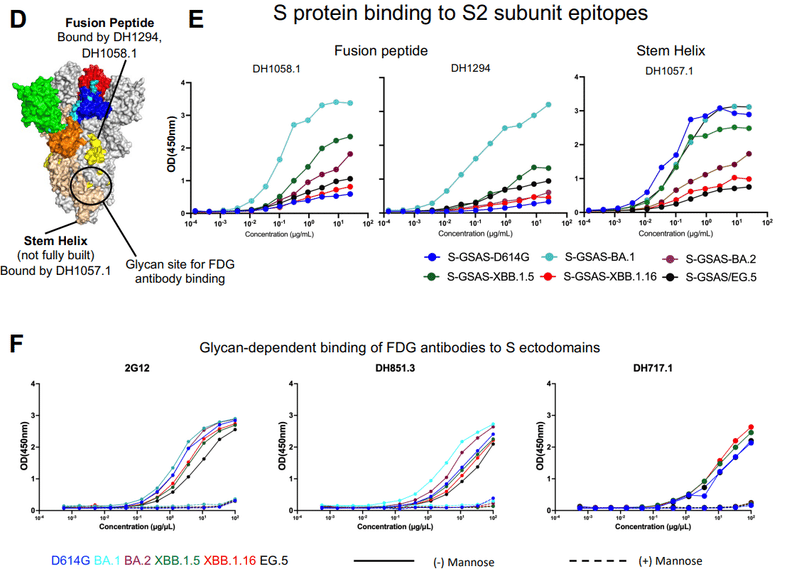
テストされたすべての S1 向け抗体のうち、 ELISA で XBB.1.5、XBB.1.16、および EG.5 S 外部ドメインへのかなりの結合を示した 2 つは、DH1193 と S309 でした。これは、重複しているにもかかわらず、RBD 外面上の別個のエピトープに結合します (図 6A ~ C、S6)。
DH1193 および S309 の ELISA 結合は、シュードウイルス中和アッセイにおける中和能力に変換されます。
最近の研究では、DH1193 エピトープ領域に結合する中和抗体がワクチン接種によって非ヒト霊長類で誘発されています。SARS-CoV-2の進化中に獲得された変異に対する耐性が低い可能性がある中和抗体を誘発するためのワクチン標的として、RBDのこの領域を強調しています。
EG.5 および EG.5.1 S 外部ドメインは、抗体 S309 および DH1193 を標的とする RBD 外面への強い結合が保持され、同様の抗原特性を示しました (図 S7)。
S2 サブユニットでは、四次 S2 グリカンクラスターを標的とする Fab 二量体化グリカン反応性 (FDG) 抗体のパネルが、グリカン依存的に XBB.1.5、XBB.1.16、および EG.5 S 外部ドメインに結合しました (図 6F)。
以前の観察と一致して、我々は、フーリンで切断された S 外部ドメインが、切断されていないバージョンよりも FDG 抗体に対して強い結合を示すことを発見しました。 S2 構造およびグリカンエピトープの提示に対するフリン切断のアロステリック効果を示しています (図 S8)。
構造モデリングにより、融合ペプチドを標的とする抗体 DH1058.1 および DH1294、およびステムヘリックスを標的とする抗体 DH1057.1 のエピトープは、融合前の SARS-CoV-2 S タンパク質への抗体結合に完全にはアクセスできないことが示されました。
それにもかかわらず、SARS-CoV-2 Sタンパク質とこれらの抗体との測定可能な結合はSPRおよびELISAによって観察でき、これは抗体誘導性のSタンパク質構造の局所的なリフォールディングが抗体エピトープを露出させることを示唆しています。
融合ペプチド指向性抗体については、我々は以前に、D614G S 外部ドメインと比較して BA.1 S 外部ドメインに対する ELISA 結合の増加を報告しました。そして、これは、BA.1 Sタンパク質のこの領域の柔軟性が増加し、抗体の存在下で融合ペプチドへのアクセスが向上することを示していると解釈しました。
BA.2 Sは、ELISAにおいてBA.1 Sと比較してDH1058.1およびDH1294への結合の減少を示し、融合前S立体構造におけるこの領域の安定化を示しました。
ここでの我々の結果は、これらの傾向を再現しており、BA.1 S が最高レベルの結合を示し、D614G S が最低、BA.2 S がその 2 つの中間であることを示しています (図 6E)。
XBB.1.16 および EG.5 S タンパク質は、BA.2 S よりも DH1058.1 への結合が低く、一方、XBB.1.5 は BA.1 S よりも大幅に低いものの、BA.2 S よりも高い結合を示します。この S タンパク質領域は、最初に同定された Omicron 変異体 BA.1 で不安定化された後、Omicron 進化時に融合前立体構造で全体的に安定化することが実証されました。
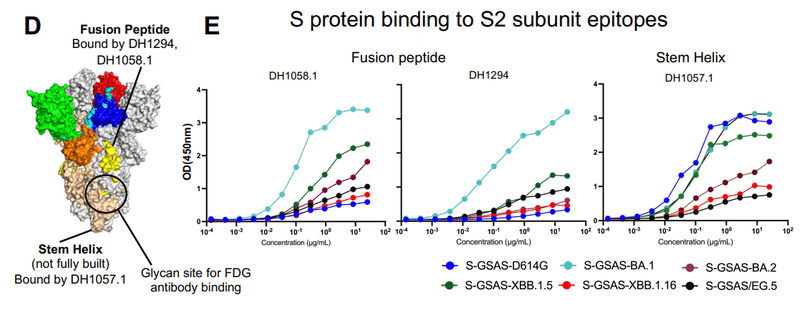
XBB.1.5 と XBB.1.16 の間で観察された ELISA 結合の違いは注目に値し、XBB.1.5 クライオ EM 亜集団で観察された S2 サブユニット構造のより多様性と一致しています。 これは、XBB.1.16 S よりも XBB.1.5 S タンパク質の方が S2 の構造的展性が大きいことを示唆しています。
2 番目に試験した融合ペプチド指向性抗体 DH1294 も同様の傾向を示しましたが、違いは DH1058.1 ほど顕著ではありませんでした (図 6E)。
DH1057.1 (ステムヘリックス) 結合で観察された傾向は、融合ペプチド指向性抗体で観察された傾向とは異なりました。D614G、BA.1 S、および XBB.1.5 S 外部ドメインでは最も強い結合が観察され、BA.2、XBB.1.16、および EG.5 S 外部ドメインでは実質的に低いレベルで結合しました (図 6E)。
融合ペプチド指向性抗体とステムヘリックス抗体の間で観察された傾向の違いは、それらのエピトープの露出機構の違いを示唆しています。
XBB.1.16またはEG.5と比較してXBB.1.5で観察された実質的に強い結合は、S2サブユニットのステムヘリックスエピトープに対する1つまたは2つのS1サブユニット変異のアロステリック効果をさらに実証しています。
まとめると、我々の抗原性データは、S1 サブユニットと S2 サブユニットのエピトープが SARS-CoV-2 の進化によってどのように異なる影響を受けたかを明らかにしています。
S1 指向性抗体では広範な結合喪失が観察され、選択されたエピトープのみが残されました。たとえば、S309 や DH1193 などの抗体に結合する RBD 外面上のものは、最初の Wu.1 スパイクに対して誘発された抗体に依然として結合することができます。
S2 サブユニットのエピトープも、S1 サブユニット残基の変更から生じる立体構造効果により、大部分が影響を受けています。
XBB.1.5 と XBB.1.16 S タンパク質間の S1 サブユニット残基の違いは 2 つだけであるにもかかわらず、融合ペプチド指向性抗体とステムヘリックスループ指向性抗体との結合の違いは、S2 サブユニットに対する S1 残基置換のアロステリック効果を示しています。
XBB.1.5 および XBB.1.16 S タンパク質の場合、S2 サブユニットの抗体結合におけるこれらの違いは、クライオ EM 集団で観察された S2 サブユニットの違いと一致していました (図 3-4)。
同様に、XBB.1.5 と EG.5 S タンパク質間の単一 RBD 残基置換にも関わらず、2 つの細胞外ドメインは融合ペプチド指向性抗体とステムヘリックス指向性抗体に対して異なる結合プロファイルを示します。
Vector analysis
ベクトル解析
次に、以前に説明したプロトマー間ベクターベースの分析を利用して、Omicron 亜系統 S タンパク質ドメインの配置を調べました (図 7)。
この分析では、ドメイン重心とアンカー残基を使用して各鎖にわたるドメイン間のベクトルを生成し、S タンパク質ドメインの四次構造を定義します (図 7A)。
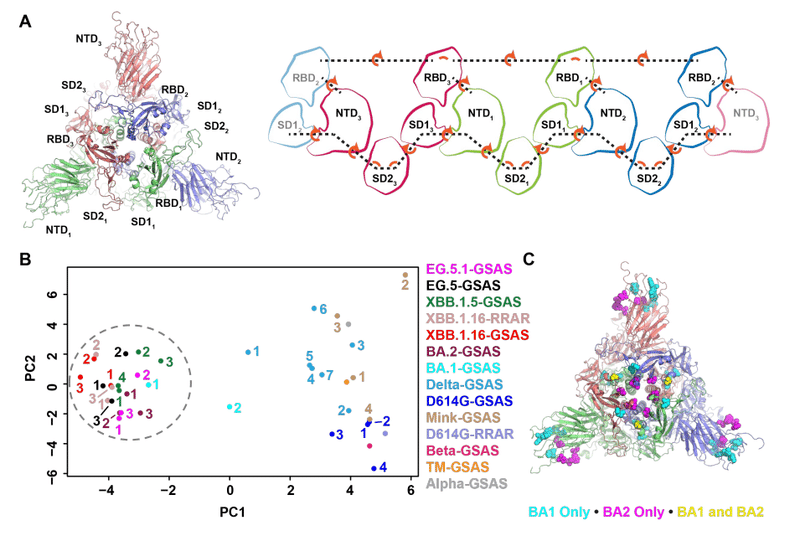
我々は以前、この幾何学形状がMERS、SARS-CoV-1、SARS-CoV-2の間で著しく異なり、SARS-CoV-2が進化するにつれてこれらの幾何学形状が変化したことを示しました。
ここで決定されたオミクロン亜系統の外部ドメイン構造、ベータおよびガンマ VOC に出現する D614G、ミンク、アルファ、ベータ、デルタ変異体および K417N-E484K-N501Y 三重変異体 (TM) の以前に決定された構造からのベクターで構成されるデータセットは、第一主成分に明確な「オミクロンクラスター」を示しました (図7B)。
このクラスターは、第 1 主成分と第 2 主成分の両方において、以前のバリアントよりも大幅に低い変動を示しました。
第 1 主成分に対するベクトルベースの寄与を調べると、初期の変異体と Omicron 変異体およびサブ系統の間の差異の主な要因は、(1) RBD 間の二面体の配向、(2) RBD 間の距離、および (3) サブドメイン 1 (SD1) から S2 の中心ヘリックスまでの距離であることが示されました。
私たちが以前に決定した単一の BA.1 構造 (以前の名前は O1BA.1、PDB: 7TF8) は、Omicron とそれ以前の亜種を分離する 2 つの主主成分クラスターの中間であるように見えました。一方、BA.2 バリアント構造は Omicron クラスターのみに存在しました。
この違いは、BA.1 と BA.2 の間で異なる突然変異が、Omicron 亜系統を安定したドメイン形状に定着させ、その後の変異体でも維持されることを示唆しています。
Omicron 亜系統におけるドメイン間の変動性の減少の推進要因を特定するために、BA.1 と BA.2 の間で、BA.1 のみ、BA.2 のみ、または BA.1 と BA.2 の両方に発生するが同一性が異なる変異位置を比較しました (図 7C)。
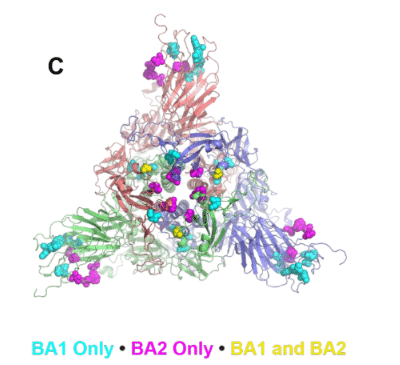
まとめると、我々のベクター解析の結果は、Omicron 亜系統における RBD ドメインの緊密な結合により、立体的に制限された S1 ドメインが作成されることを示しています。
Discussion
オミクロン株の出現は、COVID-19パンデミックにおける劇的な転換点を示しました。
オミクロンを以前の亜種と区別する主な特徴は、免疫回避が大幅に改善されたことでした。
公衆衛生上のリスクが低下したためパンデミックは終息しましたが、新たな免疫回避性亜変異体が出現し続けているため、オミクロンは依然として公衆衛生上の懸念事項であり、継続的な監視と最新のワクチンブースターが必要です。
現在最も更新されたブースターは、XBB.1.5 S タンパク質に基づく一価ワクチンです。
この研究では、我々は、XBB.1.5 S 外部ドメインとそれに続く 2 つの XBB 系統メンバー、XBB.1.16 EG.5、および EG.5.1 の S 外部ドメインの構造、受容体結合、および抗原性を特徴付けました。
私たちのデータは、XBB.1.5 RBD が以前の亜種の RBD や、その後の世界的大流行を支配した EG.5/EG.5.1 亜系統の RBD と比較してより安定していることを示しています。
より耐久性のある RBD は、現在のブースターにとって良い前兆となる可能性があります。
さらに、テストした他の オミクロン株と比較して、XBB.1.5 S タンパク質はサブドミナント S2 サブユニット エピトープへの抗体のアクセス可能性が増加したことを示しました。 特にステムヘリックス領域では、これらの領域を標的とする潜在的な防御抗体の誘発がより効果的に可能になる可能性があります。
初期の Omicron S タンパク質変異体 BA.1 および BA.2 の構造研究により、ダウン状態の RBD 間のプロトマー間相互作用の安定化につながる変異の蓄積が明らかになりました。 その結果、RBDを閉じた受容体がアクセスできない立体構造に保持する3-RBD-down立体構造を好む傾向が生じます。
この傾向は、BA.1 で初めて観察され、BA.2 ではさらに微調整され、追加の突然変異により RBD-RBD ダウン状態パッキングが最適化されると同時に、RBD 自体も安定化されました。
これは、RBD-up コンフォメーションを好む傾向があった以前の VOC とはまったく対照的であり、初期の変異体の間では伝達性が支配的な選択力であったことを示しています。
この変化により、ワクチン接種、自然感染、あるいはその両方によってかなりの集団レベルの免疫が達成されていた環境において、SARS-CoV-2が引き続き関連することが可能になりました。
ここで研究した XBB 系統の外部ドメインは、BA.2 S タンパク質の全体的なドメイン構成を保持しています。Omicron S タンパク質構造は、ベクター分析で明確なクラスターを形成しており、Omicron 以前の VOC から十分に分離されています (図 7)。
ベクター分析により、BA.1 S タンパク質構造が Omicron クラスター内に存在する 1 つの BA.1 S 外部ドメイン構造と、Omicron 分布と Omciron 以前の分布の間の中間に存在するもう 1 つの BA.1 S 外部ドメイン構造による移行点であることが強調されます。
S タンパク質構造のこの漸進的遷移パターンは、2 つの構造を重ね合わせずに比較する方法を提供する差分距離行列 (DDM) 解析でも実証されています (データ S5-S6)。
したがって、XBB.1.5、XBB.1.16、およびEG.5 S 外部ドメインは、BA.2 S タンパク質により類似し、BA.1 S とはそれほど類似せず、D614 GS タンパク質とは最も類似しません。
RBD の「ダウン」構造は、最も免疫優勢で保存されている RBD 領域を隠すため、効果的な免疫回避戦略となります。
しかし、受容体の結合と伝達にはRBD-up立体構造が必要であるため、この3-RBD-down状態の安定化には明らかな難題が存在します。
Omicron は、3-RBD ダウン状態の占有率を維持および改善しながら、どのようにして高親和性受容体結合と伝達性を維持するのでしょうか?
初期の Omicron BA.1 バリアントについては、受容体の存在下で開いた立体構造に移行する準備が整っていると想定されるひずんだ構造について説明しました。
我々の構造的結論は、BA.1 スパイクが ACE2 受容体の存在下でより容易に開放状態に移行できることを明らかにしたその後の HDX-MS 研究によって裏付けられました。
Omicron が BA.1 から BA.2 に進化するにつれて、構造上の歪みの一部が解決されたように見えましたが、XBB.1.5 S 3-RBD-down 集団で観察された構造の多様性(S2 サブユニットの構造の多様性を含む)は、内部の不安定化またはプレプライミングが依然として進化する Omicron 系統が最適化を続けているパラメーターである可能性があることを示唆しています。
Omicron が高親和性の受容体結合を維持できるようにした 2 つ目の要因は、RBD を安定化させるドメイン内の変異による RBD 自体の進化であり、また ACE2 に対する固有の親和性も強化しました。
最後に、より電気陽性の宿主膜に面する表面により、オミクロン S タンパク質とヘパラン硫酸などの宿主細胞表面共受容体との相互作用が促進され、宿主細胞近くのウイルスの蓄積が増加し、スパイクの開口が促進された可能性があります。
Omicron BA.1 S タンパク質では、宿主膜に面する側の陽性表面の増加が認められました。
実際、オミクロン株は、現在流行しているコロナウイルスの戦略の 1 ページを奪っている可能性があります。それらコロナウイルスは、 通常、宿主の糖衣との相互作用により、RBD のアップ位置への移行を促進する 3-RBD ダウン構造(したがって、宿主受容体に結合できる立体構造への必要な移行を確実にしながら、最適な免疫回避が可能)を採用してます。
オミクロン株スパイクの継続的な進化には、主な要因が免疫回避であるマルチパラメータの最適化が含まれています。
同時に最適化する必要がある他の特性には、融合前の立体構造安定性、宿主受容体結合、宿主細胞への侵入に必要な立体構造変化を受ける能力などがあります。
私たちの研究は、XBB 系統がスパイクの安定性と受容体結合を改善しながら免疫回避特性をどのように獲得したか、そしてそれがどのように進化し続けてきたかを示しています。
わずか 1 つまたは 2 つの残基置換によって影響を受ける実質的な構造的、立体構造的、および抗原性の影響は、SARS-CoV-2 S タンパク質におけるアロステリック ネットワークの有効性を強調し、宿主免疫から逃れるためのその継続的な進化を予告しています。
以下省略。
この記事が気に入ったらサポートをしてみませんか?
