
Nishikaコンペ:材料の物性予測 ~機械学習で材料の研究開発を推進しよう~その3:特徴量エンジニアリング編
1.概要
Nishikaで2023/03/10まで開催中のコンペ「材料の物性予測 ~機械学習で材料の研究開発を推進しよう~」に参加してみました。本記事では下記記事の続きになります。
前回の記事では「AutoML」を使用しましたが十分な性能は出ませんでした。結果を見るとTree系で比較的性能が高く出ており、特徴量エンジニアリングで性能が上がりそうです。
本記事では「CIF情報を解析(Parse)して重要な特徴量を抽出する(特徴量エンジニアリング)ことで性能向上」を試してみます。
2.前回記事の振り返り
前回記事の中で今回使用する情報を抽出しました。
2-1.データ概要の確認
データは化合物の物性・組成情報から「生成エネルギー(eV/atom):標準状態の元素に対する化合物のエネルギーを原子単位に正規化した値」を求める回帰分析の問題です。
基本的にはテーブルデータのため、数値データをGBDT(XGBoost)のような強いモデルに与えれば条件次第でそれなりの結果は出そう
原子数のデータはスパース(下表の通りほとんどが0の値)のため、そのまま使用するのは効率が悪そう
データを1個抽出しましたが、データ情報は下記の通りです。
id: 材料のid
Ac, Ag, ..., Zn, Zr: などの89種類の原子が構造式に含まれる数量
nsites: 組成式に含まれる原子の合計数
nelements: 組成式に含まれる原子の種類数
formula_pretty: 正規化された化学式
cif: Crystallographic Information File形式の結晶構造情報
CIFの詳細については参考文献[1][3]をご参照下さい。formation_energy_per_atom: 生成エネルギー(目的変数)
2-2.事前のコードまとめ
前回記事で記載済みの前処理の部分までのコードをまとめておきました。
[IN]
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import japanize_matplotlib
import seaborn as sns
import os
import glob
import shutil
from requests import get
import re
import itertools
from tqdm import tqdm
import missingno as msno
import random
import joblib
import pickle
import lightgbm as lgbm
from sklearn.metrics import mean_squared_error
from sklearn.model_selection import KFold
import torch
import torch.nn as nn
import torch.nn.functional as F
from torch.utils.data import Dataset, DataLoader
from torch.optim.lr_scheduler import StepLR
from pymatgen.core import Structure
class HorizontalDisplay:
def __init__(self, *args):
self.args = args
def _repr_html_(self):
template = '<div style="float: left; padding: 10px;">{}</div>'
return ''.join(template.format(a._repr_html_()) for a in self.args)
#データの中身確認
datafiles = glob.glob('data/*')
file_explain, file_submit, file_test, file_train = datafiles
df_train = pd.read_csv(file_train) #訓練用データ(train.csv)
df_test = pd.read_csv(file_test) #評価用データ(test.csv)
df_submit = pd.read_csv(file_submit) #投稿データフォーマット(sample_submission.csv)
print(f'データ形状(train):{df_train.shape}、データ形状(test):{df_test.shape}、データ形状(submit):{df_submit.shape}')
display(df_train.head(3))
display(df_train.describe())
excel_pd = pd.ExcelFile(file_explain) #データ説明(data_explanation.xlsx)
sheetnames = excel_pd.sheet_names
df_explain = pd.read_excel(file_explain, sheet_name=sheetnames) #データ説明(data_explanation.xlsx)
display(df_explain['カラム説明'].T)
[OUT]
データ形状(train):(71729, 95)、データ形状(test):(28691, 94)、データ形状(submit):(28691, 2)3.特徴量エンジニアリング:CIF解析
前回記事では使用しなかったCIF情報を使用して特徴量(記述子)を増やしていきます。当然ですが意味のない特徴量を増やしても計算負荷が上がるだけのため、なるべくドメイン知識(専門知識)を使用してデータ抽出していきます。
※大学で無機化学を習ってはいますが、正直ほとんど覚えていません。
3-1.結晶構造とは
金属は結晶構造を持っており規則的に原子が配列してます。構造の基礎単位が単位格子(unit lattice)であり、この構造には3種類あります。細かく分けると更なる結晶学的点群が存在します。
参考ですが、結晶構造を持たない物質の状態は”アモルファス”とよび、ガラスなどが該当します。
【金属の結晶構造】
面心立方格子 (face centered cubic lattice, FCC)
体心立方格子 (body centered cubic lattice, BCC)
六方最密格子 (hexagonal close-packed lattice, HCP)
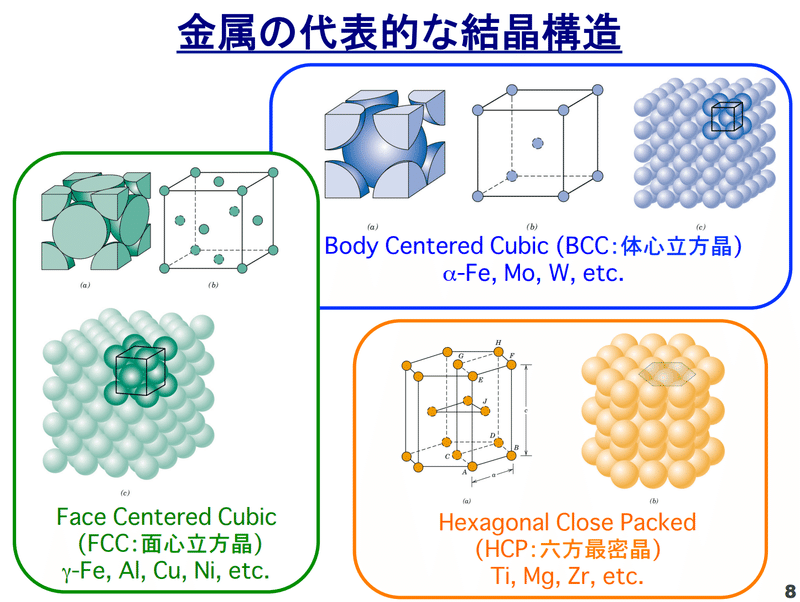

上記より結晶構造で”格子定数 (lattice constant)”と”角度”は重要そうな特徴量であると推測できます。
3-2.CIFデータの理解
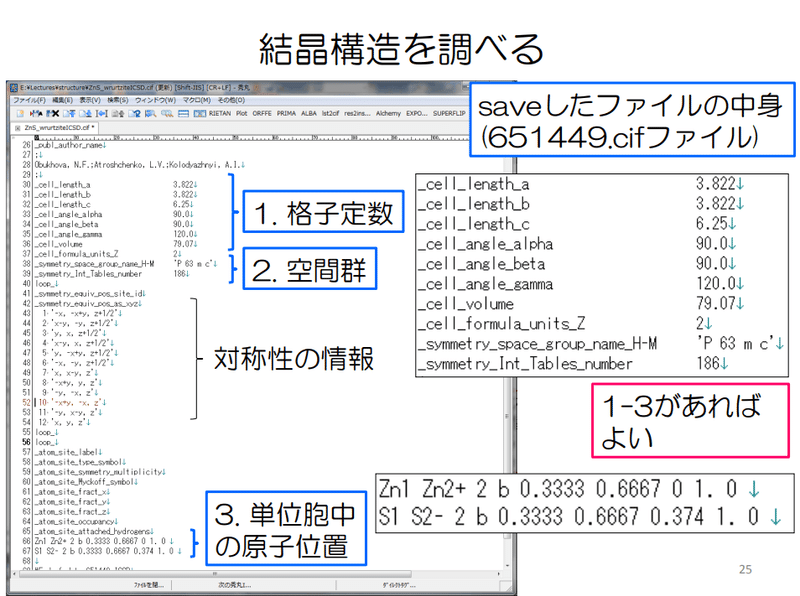
CIF(Crystallograhic Information File)とは”国際結晶学連盟が推奨している結晶学情報共通データ・フォーマット”であり、結晶構造データと特定の形で保存したものです。今回のCIFデータを抽出して中身を確認してみます。結果は下記の通りです。
CIFデータそのものはString型である。
頭に"# generated using pymatgen"があり、これはCIFデータではなくただのコメント
格子定数(_cell_length)、角度(cell_angle)、体積(_cell_volume)はすぐに使用できそう
単位格子中の非対称単位数(_symmetry_Int_Tables_number)も使ってよさそう
空間群の情報(_symmetry_space_group_name_H-M)はあるのでこれは使えそう。ただしカテゴリカルデータであり種類も多いのでどう扱うかが困る(one-hotだとスパースだし、ラベルにすると大小に意味がでるのでtree系以外だと邪魔になる可能性がある)_symmetry_Int_Tables_numberは空間群情報?
周期境界条件(pbc:Periodic boundary conditions)は何かに使えそうな気がするけど、データの意味が分からん。
[IN]
import pymatgen as mg
from pymatgen.core.structure import Structure
from pymatgen.io.cif import CifParser
data_cif = df_train['cif'].values
raw_cif = data_cif[0]
print(type(raw_cif))
print(raw_cif)
[OUT]
<class 'str'>
# generated using pymatgen
data_K2MgMo2(H2O5)2
_symmetry_space_group_name_H-M 'P 1'
_cell_length_a 6.00896235
_cell_length_b 6.55026940
_cell_length_c 7.95639355
_cell_angle_alpha 111.39242719
_cell_angle_beta 96.77130034
_cell_angle_gamma 109.07338782
_symmetry_Int_Tables_number 1
_chemical_formula_structural K2MgMo2(H2O5)2
_chemical_formula_sum 'K2 Mg1 Mo2 H4 O10'
_cell_volume 265.39072919
_cell_formula_units_Z 1
loop_
_symmetry_equiv_pos_site_id
_symmetry_equiv_pos_as_xyz
1 'x, y, z'
loop_
_atom_site_type_symbol
_atom_site_label
_atom_site_symmetry_multiplicity
_atom_site_fract_x
_atom_site_fract_y
_atom_site_fract_z
_atom_site_occupancy
K K0 1 0.33066327 0.70285292 0.24630803 1
K K1 1 0.66933673 0.29714708 0.75369197 1
Mg Mg2 1 -0.00000000 -0.00000000 -0.00000000 1
Mo Mo3 1 0.33274891 0.66534980 0.76077634 1
Mo Mo4 1 0.66725109 0.33465020 0.23922366 1
H H5 1 0.92202881 0.86654805 0.28439073 1
H H6 1 0.07797119 0.13345195 0.71560927 1
H H7 1 0.12465733 0.12755969 0.37626561 1
H H8 1 0.87534267 0.87244031 0.62373439 1
O O9 1 0.36066563 0.82312200 0.62054963 1
O O10 1 0.63933437 0.17687800 0.37945037 1
O O11 1 0.23666938 0.34989125 0.61648940 1
O O12 1 0.76333062 0.65010875 0.38351060 1
O O13 1 0.62693462 0.76248819 0.92136504 1
O O14 1 0.37306538 0.23751181 0.07863496 1
O O15 1 0.10929347 0.70888614 0.89009389 1
O O16 1 0.89070653 0.29111386 0.10990611 1
O O17 1 0.05337462 0.97851338 0.25767835 1
O O18 1 0.94662538 0.02148662 0.74232165 1【参考:変数名の意味】


3-3.pymatgenでCIFを解析:Structure
pymatgenでのCIF解析は"Structure"を使用します。なお別の手法として”CifParser.from_string(raw_cif).get_structures()[0]”でも同じ結果が得られます。
[IN]
import pymatgen as mg
from pymatgen.core.structure import Structure
from pymatgen.io.cif import CifParser
data_cif = df_train['cif'].values
raw_cif = data_cif[0]
#CIFから構造を取得
structure = Structure.from_str(raw_cif, fmt='cif')
display(structure)
[OUT]
Structure Summary
Lattice
abc : 6.008962350000001 6.5502694 7.95639355
angles : 111.39242719 96.77130034 109.07338782
volume : 265.39072946673986
A : 5.9670479898753515 0.0 -0.7084961610637405
B : -2.4392110195731824 5.589976226156952 -2.389235150747871
C : 0.0 0.0 7.95639355
pbc : True True True
PeriodicSite: K (0.2587, 3.9289, 0.0462) [0.3307, 0.7029, 0.2463]
PeriodicSite: K (3.2692, 1.6610, 4.8125) [0.6693, 0.2971, 0.7537]
PeriodicSite: Mg (0.0000, 0.0000, 0.0000) [0.0000, 0.0000, 0.0000]
PeriodicSite: Mo (0.3626, 3.7193, 4.2276) [0.3327, 0.6653, 0.7608]
PeriodicSite: Mo (3.1652, 1.8707, 0.6311) [0.6673, 0.3347, 0.2392]
PeriodicSite: H (3.3881, 4.8440, -0.4609) [0.9220, 0.8665, 0.2844]
PeriodicSite: H (0.1397, 0.7460, 5.3196) [0.0780, 0.1335, 0.7156]
PeriodicSite: H (0.4327, 0.7131, 2.6006) [0.1247, 0.1276, 0.3763]
PeriodicSite: H (3.0951, 4.8769, 2.2580) [0.8753, 0.8724, 0.6237]
PeriodicSite: O (0.1443, 4.6012, 2.7152) [0.3607, 0.8231, 0.6205]
PeriodicSite: O (3.3835, 0.9887, 2.1435) [0.6393, 0.1769, 0.3795]
PeriodicSite: O (0.5588, 1.9559, 3.9014) [0.2367, 0.3499, 0.6165]
PeriodicSite: O (2.9691, 3.6341, 0.9573) [0.7633, 0.6501, 0.3835]
PeriodicSite: O (1.8811, 4.2623, 5.0648) [0.6269, 0.7625, 0.9214]
PeriodicSite: O (1.6468, 1.3277, -0.2061) [0.3731, 0.2375, 0.0786]
PeriodicSite: O (-1.0770, 3.9627, 5.3108) [0.1093, 0.7089, 0.8901]
PeriodicSite: O (4.6048, 1.6273, -0.4521) [0.8907, 0.2911, 0.1099]
PeriodicSite: O (-2.0683, 5.4699, -0.3255) [0.0534, 0.9785, 0.2577]
PeriodicSite: O (5.5961, 0.1201, 5.1842) [0.9466, 0.0215, 0.7423][IN]
#CIFから構造を取得
CifParser.from_string(raw_cif).get_structures()[0]
[OUT]
同上Structureのメソッドを確認すると体積などの格子情報は取得できますが、情報量が多くどれを取得してよいか分かりません。よって次節で格子情報と空間群の情報を分けてとるメソッドを使用します。
[IN]
structure = Structure.from_str(raw_cif, fmt='cif')
[i for i in dir(structure) if not i.startswith('_')]
[OUT]
['DISTANCE_TOLERANCE', 'REDIRECT', 'add_oxidation_state_by_element', 'add_oxidation_state_by_guess', 'add_oxidation_state_by_site',
'add_site_property', 'add_spin_by_element', 'add_spin_by_site', 'append', 'apply_operation', 'apply_strain', 'as_dataframe',
'as_dict', 'atomic_numbers', 'cart_coords', 'charge', 'clear', 'composition', 'copy', 'count', 'density', 'distance_matrix',
'extend', 'extract_cluster', 'formula', 'frac_coords', 'from_dict', 'from_file', 'from_magnetic_spacegroup', 'from_prototype',
'from_sites', 'from_spacegroup', 'from_str', 'get_all_neighbors', 'get_all_neighbors_old', 'get_all_neighbors_py', 'get_angle',
'get_dihedral', 'get_distance', 'get_miller_index_from_site_indexes', 'get_neighbor_list', 'get_neighbors', 'get_neighbors_in_shell',
'get_neighbors_old', 'get_orderings', 'get_primitive_structure', 'get_reduced_structure', 'get_sites_in_sphere', 'get_sorted_structure',
'get_space_group_info', 'get_symmetric_neighbor_list', 'group_by_types', 'index', 'indices_from_symbol', 'insert', 'interpolate',
'is_3d_periodic', 'is_ordered', 'is_valid', 'lattice', 'make_supercell', 'matches', 'merge_sites', 'ntypesp', 'num_sites', 'pbc',
'perturb', 'pop', 'relax', 'remove', 'remove_oxidation_states', 'remove_site_property', 'remove_sites', 'remove_species', 'remove_spin',
'replace', 'replace_species', 'reverse', 'rotate_sites', 'scale_lattice', 'set_charge', 'site_properties', 'sites', 'sort', 'species',
'species_and_occu', 'substitute', 'symbol_set', 'to', 'to_json', 'translate_sites', 'types_of_specie', 'types_of_species', 'unsafe_hash',
'validate_monty', 'volume'] 【参考:structure.as_dict()】
下記ではlatticeとget_space_group_info()を使用しましたが"as_dict()"メソッドでも比較的多くの情報が抽出できます。
sitesキーだけリストに入っており情報量も多いため別途ループで中身を確認しました。
[IN]
display(structure.as_dict())
for i in structure.as_dict()['sites']:
print(i)
[OUT]
{'@module': 'pymatgen.core.structure',
'@class': 'Structure',
'charge': 0.0,
'lattice': {'matrix': [[5.9670479898753515, 0.0, -0.7084961610637405],
[-2.4392110195731824, 5.589976226156952, -2.389235150747871],
[0.0, 0.0, 7.95639355]],
'pbc': (True, True, True),
'a': 6.008962350000001,
'b': 6.5502694,
'c': 7.95639355,
'alpha': 111.39242719,
'beta': 96.77130034,
'gamma': 109.07338782,
'volume': 265.39072946673986},
'sites': [{'species': [{'element': 'K', 'occu': 1.0}],
'abc': [0.33066327, 0.70285292, 0.24630803],
'xyz': [0.25867701297592216, 3.928931113284994, 0.04616906153564204],
'label': 'K',
'properties': {}},
{'species': [{'element': 'K', 'occu': 1.0}],
'abc': [0.66933673, 0.29714708, 0.75369197],
'xyz': [3.269159957326247, 1.6610451128719579, 4.812493176652746],
'label': 'K',
'properties': {}},
以下省略
'properties': {}},
{'species': [{'element': 'O', 'occu': 1.0}],
'abc': [0.94662538, 0.02148662, 0.74232165],
'xyz': [5.596148670616609, 0.12010969498046849, 5.184186152615091],
'label': 'O',
'properties': {}}]}
{'species': [{'element': 'K', 'occu': 1.0}], 'abc': [0.33066327, 0.70285292, 0.24630803], 'xyz': [0.25867701297592216, 3.928931113284994, 0.04616906153564204], 'label': 'K', 'properties': {}}
{'species': [{'element': 'K', 'occu': 1.0}], 'abc': [0.66933673, 0.29714708, 0.75369197], 'xyz': [3.269159957326247, 1.6610451128719579, 4.812493176652746], 'label': 'K', 'properties': {}}
{'species': [{'element': 'Mg', 'occu': 1.0}], 'abc': [0.0, 0.0, 0.0], 'xyz': [0.0, 0.0, 0.0], 'label': 'Mg', 'properties': {}}
{'species': [{'element': 'Mo', 'occu': 1.0}], 'abc': [0.33274891, 0.6653498, 0.76077634], 'xyz': [0.3626001505179013, 3.7192895640782826, 4.2276075095323975], 'label': 'Mo', 'properties': {}}
{'species': [{'element': 'Mo', 'occu': 1.0}], 'abc': [0.66725109, 0.3346502, 0.23922366], 'xyz': [3.165236819784268, 1.8706866620786693, 0.6310547286559913], 'label': 'Mo', 'properties': {}}
{'species': [{'element': 'H', 'occu': 1.0}], 'abc': [0.92202881, 0.86654805, 0.28439073], 'xyz': [3.3880966047680094, 4.843982998322666, -0.4609163632954014], 'label': 'H', 'properties': {}}
{'species': [{'element': 'H', 'occu': 1.0}], 'abc': [0.07797119, 0.13345195, 0.71560927], 'xyz': [0.1397403655341597, 0.7459932278342863, 5.319578601483789], 'label': 'H', 'properties': {}}
{'species': [{'element': 'H', 'occu': 1.0}], 'abc': [0.12465733, 0.12755969, 0.37626561], 'xyz': [0.4326912688983892, 0.7130556345159507, 2.6006279375708576], 'label': 'H', 'properties': {}}
{'species': [{'element': 'H', 'occu': 1.0}], 'abc': [0.87534267, 0.87244031, 0.62373439], 'xyz': [3.09514570140378, 4.876920591641001, 2.2580343006175303], 'label': 'H', 'properties': {}}
{'species': [{'element': 'O', 'occu': 1.0}], 'abc': [0.36066563, 0.823122, 0.62054963], 'xyz': [0.1443408696555104, 4.601232411226762, 2.7151748435503618], 'label': 'O', 'properties': {}}
{'species': [{'element': 'O', 'occu': 1.0}], 'abc': [0.63933437, 0.176878, 0.37945037], 'xyz': [3.383496100646659, 0.9887438149301894, 2.1434873946380266], 'label': 'O', 'properties': {}}
{'species': [{'element': 'O', 'occu': 1.0}], 'abc': [0.23666938, 0.34989125, 0.6164894], 'xyz': [0.5587589555418105, 1.9558837692403386, 3.901380465192923], 'label': 'O', 'properties': {}}
{'species': [{'element': 'O', 'occu': 1.0}], 'abc': [0.76333062, 0.65010875, 0.3835106], 'xyz': [2.969078014760359, 3.6340924569166133, 0.957281772995465], 'label': 'O', 'properties': {}}
{'species': [{'element': 'O', 'occu': 1.0}], 'abc': [0.62693462, 0.76248819, 0.92136504], 'xyz': [1.881079368711857, 4.262290854825445, 5.064798504365415], 'label': 'O', 'properties': {}}
{'species': [{'element': 'O', 'occu': 1.0}], 'abc': [0.37306538, 0.23751181, 0.07863496], 'xyz': [1.646757601590312, 1.3276853713315069, -0.20613626617702718], 'label': 'O', 'properties': {}}
{'species': [{'element': 'O', 'occu': 1.0}], 'abc': [0.10929347, 0.70888614, 0.89009389], 'xyz': [-1.0769635038406957, 3.962656669652169, 5.310807597800098], 'label': 'O', 'properties': {}}
{'species': [{'element': 'O', 'occu': 1.0}], 'abc': [0.89070653, 0.29111386, 0.10990611], 'xyz': [4.6048004741428645, 1.6273195565047833, -0.45214535961170943], 'label': 'O', 'properties': {}}
{'species': [{'element': 'O', 'occu': 1.0}], 'abc': [0.05337462, 0.97851338, 0.25767835], 'xyz': [-2.0683117003144402, 5.469866531176483, -0.3255239144267019], 'label': 'O', 'properties': {}}
{'species': [{'element': 'O', 'occu': 1.0}], 'abc': [0.94662538, 0.02148662, 0.74232165], 'xyz': [5.596148670616609, 0.12010969498046849, 5.184186152615091], 'label': 'O', 'properties': {}}3-4.格子定数の情報:structure.lattice
格子情報は”structure.lattice”で取得します。下記の通り属性を使用することで情報を抽出できます。
[IN]
#CIFから構造を取得
structure = Structure.from_str(raw_cif, fmt='cif')
lattice = structure.lattice #格子定数
display(lattice)
print([i for i in dir(lattice) if not i.startswith('_')])
[OUT]
Lattice
abc : 6.008962350000001 6.5502694 7.95639355
angles : 111.39242719 96.77130034 109.07338782
volume : 265.39072946673986
A : 5.9670479898753515 0.0 -0.7084961610637405
B : -2.4392110195731824 5.589976226156952 -2.389235150747871
C : 0.0 0.0 7.95639355
pbc : True True True
['REDIRECT', 'a', 'abc', 'alpha', 'angles', 'as_dict', 'b', 'beta', 'c', 'copy',
'cubic', 'd_hkl', 'dot', 'find_all_mappings', 'find_mapping', 'from_dict',
'from_parameters', 'gamma', 'get_all_distances', 'get_brillouin_zone', 'get_cartesian_coords',
'get_distance_and_image', 'get_frac_coords_from_lll', 'get_fractional_coords', 'get_lll_frac_coords',
'get_lll_reduced_lattice', 'get_miller_index_from_coords', 'get_niggli_reduced_lattice', 'get_points_in_sphere',
'get_points_in_sphere_old', 'get_points_in_sphere_py', 'get_recp_symmetry_operation',
'get_vector_along_lattice_directions', 'get_wigner_seitz_cell', 'hexagonal', 'inv_matrix',
'is_3d_periodic', 'is_hexagonal', 'is_orthogonal', 'lengths', 'lll_inverse', 'lll_mapping',
'lll_matrix', 'matrix', 'metric_tensor', 'monoclinic', 'norm', 'orthorhombic', 'parameters',
'pbc', 'reciprocal_lattice', 'reciprocal_lattice_crystallographic', 'rhombohedral', 'scale', 'selling_dist',
'selling_vector', 'tetragonal', 'to_json', 'unsafe_hash', 'validate_monty', 'volume']参考として何個か情報を抽出しました。
[IN]
print(f'格子間距離:{lattice.abc}')
print(f'格子角度:{lattice.angles}')
print(f'格子体積:{lattice.volume}')
print(f'pbc: {lattice.pbc}')
print(f'格子定数(ベクトル):{lattice.matrix}')
[OUT]
格子間距離:(6.008962350000001, 6.5502694, 7.95639355)
格子角度:(111.39242719, 96.77130034, 109.07338782)
格子体積:265.39072946673986
pbc: (True, True, True)
格子定数(ベクトル):
[[ 5.96704799 0. -0.70849616]
[-2.43921102 5.58997623 -2.38923515]
[ 0. 0. 7.95639355]]3-5.空間群の情報:structure.get_space_group_info()
空間群の情報は”structure.get_space_group_info()”を使用します。こちらは特に属性はなく出力をそのまま使用します。
[IN]
space_group = structure.get_space_group_info() #空間群
display(space_group)
print([i for i in dir(space_group) if not i.startswith('_')])
[OUT]
('P-1', 2)
['count', 'index']3-6.Pythonによる実装
今回の実装での思想は下記の通りです。
条件として重要そうであり分かりやすい格子定数(距離)、角度、体積の情報のみを使用
元のDataFrameに結晶情報を追加後に機械学習の入力値とする。
処理の流れとしては下記の通りです。サンプルとして1行だけで入出力しました。
pymatgenの”Structure.lattice”でCIFからlatticeクラスを出力する関数作成
DataFrameの"cif"カラムにapply(<latticeを出力する関数>)を適用して格子クラスを出力
getattr()で各情報の値を属性名から取得
元のDataFrameと抽出したDataFrameを統合して出力
[IN]
def Parse_CIF(text):
#CIFから構造を取得
structure = Structure.from_str(text, fmt='cif')
lattice = structure.lattice #格子定数
return lattice
#Latticeから格子定数をDataFrameに格納
def Parse_df(df, col='cif'):
#取得する属性
names_attr = ['a', 'b', 'c', 'alpha', 'beta', 'gamma', 'volume'] #格子定数、角度、体積
_df = pd.DataFrame(columns=names_attr) #空のDataFrameを作成
df_lattice = df[col].apply(Parse_CIF) #CIFから構造を取得
for name in names_attr:
_df[name] = df_lattice.apply(lambda x: getattr(x, name))
df_output = pd.concat([df, _df], axis=1) #元のDataFrameに結合
return df_output
_df1 = df_train.iloc[:1, :]
display(_df1)
Parse_df(_df1, col='cif')
[OUT]
上記関数で学習データを処理します。なお処理には時間がかかるため出力値はPickle化しておくと別モデルに適用時に高速で読み込めます。
[IN]
#CIFをParseして保存
df_train_cif = Parse_df(df_train, col='cif')
df_train_cif.to_pickle('tempdata/df_train_cif.pkl')
# df_train_cif_all = pd.read_pickle('tempdata/df_train_cif.pkl') #保存したものを読み込み
targets = df_train_cif_all['formation_energy_per_atom'].values #目的変数(Numpy配列に変換)
df_train_cif_all.head()
[OUT]
4.モデル実装:LightGBM
ハイパーパラメータを指定したLightGBMで実装します。各種詳細は前回の記事通りです。
4-1.学習・検証
学習・検証Phaseの大きな流れは下記の通りです。
交差検証の分割数(fold)を設定
LightGBMで使用するハイパーパラメータを設定
入力値(Pandasでカラムを指定)、ラベルを作成
ループ処理においてenumerate()のindexからfold番号を抽出できるよう、事前に4-1節で作成したfoldデータをNumpy型で用意しておく(val_fold)。
"val_fold=index番号"でmask(boolのリスト)を作成できるため、maskをデータに渡すことで学習用・検証用データに分割
モデルを作成・学習・推論を実施
各foldでモデルをPickleファイルで保存し、modelsリストに格納(次節でアンサンブルによる各モデルの平均値を推論値とします)
検証用データの性能(RMSE)を確認+各foldの平均値を算出
[IN]
#交差検証Params
n_folds = 5 #分割数
kf = KFold(n_splits=n_folds, random_state=42, shuffle=True) #KFoldの設定
#DataFrameに交差検証時のfold番号(何番目か)を追加
for i, (train_idx, val_idx) in enumerate(kf.split(targets)):
df_train_cif_all.loc[val_idx, 'val_fold'] = i #foldの列にfold番号を格納
#学習用のDataFrameを作成
df_train_cif = df_train_cif_all.drop(columns=['id', 'formula_pretty', 'cif', 'formation_energy_per_atom', 'val_fold'])
#LightGBMのParams
lgbm_params = {
"objective": "regression", #回帰問題
"learning_rate": 0.2, #学習率
"reg_alpha": 0.1, #L1正則化
"reg_lambda": 0.1, #L2正則化
"random_state": 42, #乱数シード
"max_depth": 5, #木の深さ
"n_estimators": 1000000, #学習回数
"colsample_bytree": 0.9,
}
#LightGBMによるモデル作成
X = df_train_cif.values #説明変数
targets = df_train_cif_all['formation_energy_per_atom'].values #目的変数
print(f'形状確認 X:{X.shape} targets:{targets.shape}')
models = [] #モデルを格納するリスト
oof = np.zeros(targets.shape[0]) #Out Of Foldの予測値を格納する配列
val_fold = df_train_cif_all['val_fold'].values #foldの列を取得(0~4):Numpy配列に変換
for i_fols in tqdm(range(n_folds)):
print(f'Fold {i_fols + 1} / {n_folds}')
X_train, y_train = X[val_fold != i_fols], targets[val_fold != i_fols] #fold番号がi_folsでないデータを学習データとして取得
X_val, y_val = X[val_fold == i_fols], targets[val_fold == i_fols] #fold番号がi_folsのデータを検証データとして取得
#モデルの学習
model = lgbm.LGBMRegressor(**lgbm_params) #モデルの作成
model.fit(X_train, y_train,
eval_set = [(X_val, y_val)],
eval_metric='rmse',
callbacks=[lgbm.early_stopping(stopping_rounds=20, verbose=True),# early_stopping_rounds()の代わりにcallbacksを使う
lgbm.log_evaluation(400)]) #400回ごとに検証データのRMSEを表示
#モデルの予測
pred = model.predict(X_val)
#Out Of Foldの予測値を格納
oof[val_fold == i_fols] = model.predict(X_val) #fold番号がi_folsのデータの予測値を格納
cv_fold_rmse = np.sqrt(mean_squared_error(y_val, pred)) #fold番号がi_folsのデータのRMSEを計算
print(f'CV-fold {i_fols + 1} RMSE: {cv_fold_rmse:.4f}')
#各foldのモデルを保存
filename_model = f'model/Case1_fold{i_fols}.pkl' #モデルの保存先
with open(filename_model, 'wb') as f:
pickle.dump(model, f) #モデルの保存
models.append(model) #モデルをリストに追加
cv_rmse = np.sqrt(mean_squared_error(targets, oof)) #Out Of FoldのRMSEを計算
print(f'CV RMSE: {cv_rmse:.4f}')
[OUT]
形状確認 X:(71729, 98) targets:(71729,)
0%| | 0/5 [00:00<?, ?it/s]Fold 1 / 5
Training until validation scores don't improve for 20 rounds
[400] valid_0's rmse: 0.324471 valid_0's l2: 0.105281
[800] valid_0's rmse: 0.286322 valid_0's l2: 0.0819802
[1200] valid_0's rmse: 0.270831 valid_0's l2: 0.0733494
[1600] valid_0's rmse: 0.262334 valid_0's l2: 0.0688193
[2000] valid_0's rmse: 0.256666 valid_0's l2: 0.0658773
[2400] valid_0's rmse: 0.252293 valid_0's l2: 0.0636516
[2800] valid_0's rmse: 0.249706 valid_0's l2: 0.0623529
Early stopping, best iteration is:
[3149] valid_0's rmse: 0.24792 valid_0's l2: 0.0614642
20%|██ | 1/5 [00:05<00:22, 5.55s/it]CV-fold 1 RMSE: 0.2479
Fold 2 / 5
Training until validation scores don't improve for 20 rounds
[400] valid_0's rmse: 0.305192 valid_0's l2: 0.093142
[800] valid_0's rmse: 0.27101 valid_0's l2: 0.0734464
[1200] valid_0's rmse: 0.258094 valid_0's l2: 0.0666128
[1600] valid_0's rmse: 0.250051 valid_0's l2: 0.0625254
[2000] valid_0's rmse: 0.245732 valid_0's l2: 0.0603842
Early stopping, best iteration is:
[2081] valid_0's rmse: 0.244983 valid_0's l2: 0.0600167
40%|████ | 2/5 [00:09<00:13, 4.47s/it]CV-fold 2 RMSE: 0.2450
Fold 3 / 5
Training until validation scores don't improve for 20 rounds
[400] valid_0's rmse: 0.309237 valid_0's l2: 0.0956276
[800] valid_0's rmse: 0.274798 valid_0's l2: 0.0755139
[1200] valid_0's rmse: 0.260065 valid_0's l2: 0.0676337
[1600] valid_0's rmse: 0.252385 valid_0's l2: 0.063698
[2000] valid_0's rmse: 0.247534 valid_0's l2: 0.0612733
[2400] valid_0's rmse: 0.244393 valid_0's l2: 0.0597281
Early stopping, best iteration is:
[2771] valid_0's rmse: 0.241975 valid_0's l2: 0.0585518
60%|██████ | 3/5 [00:13<00:09, 4.54s/it]CV-fold 3 RMSE: 0.2420
Fold 4 / 5
Training until validation scores don't improve for 20 rounds
[400] valid_0's rmse: 0.312802 valid_0's l2: 0.0978449
[800] valid_0's rmse: 0.277853 valid_0's l2: 0.0772023
[1200] valid_0's rmse: 0.264054 valid_0's l2: 0.0697244
[1600] valid_0's rmse: 0.256016 valid_0's l2: 0.065544
[2000] valid_0's rmse: 0.250851 valid_0's l2: 0.0629262
[2400] valid_0's rmse: 0.24747 valid_0's l2: 0.0612414
[2800] valid_0's rmse: 0.245072 valid_0's l2: 0.0600603
[3200] valid_0's rmse: 0.243315 valid_0's l2: 0.0592022
[3600] valid_0's rmse: 0.241605 valid_0's l2: 0.0583731
Early stopping, best iteration is:
[3644] valid_0's rmse: 0.241497 valid_0's l2: 0.0583209
80%|████████ | 4/5 [00:19<00:05, 5.05s/it]CV-fold 4 RMSE: 0.2415
Fold 5 / 5
Training until validation scores don't improve for 20 rounds
[400] valid_0's rmse: 0.320652 valid_0's l2: 0.102818
[800] valid_0's rmse: 0.284949 valid_0's l2: 0.0811961
[1200] valid_0's rmse: 0.269645 valid_0's l2: 0.0727085
[1600] valid_0's rmse: 0.260937 valid_0's l2: 0.0680879
[2000] valid_0's rmse: 0.256067 valid_0's l2: 0.0655702
[2400] valid_0's rmse: 0.252495 valid_0's l2: 0.0637536
Early stopping, best iteration is:
[2738] valid_0's rmse: 0.249983 valid_0's l2: 0.0624915
100%|██████████| 5/5 [00:24<00:00, 4.87s/it]CV-fold 5 RMSE: 0.2500
CV RMSE: 0.24534-2.可視化
決定木はどのパラメータ(入力値)の影響が強いかを可視化することが出来ます。重要なこととして結晶構造の情報が特徴量として重要であることが示唆されています。
[IN]
plt.style.use('seaborn-whitegrid')
df_imp = pd.DataFrame(models[0].feature_importances_,
index = list(df_train_cif.columns),
columns=['importance']).sort_values('importance', ascending=False)
plt.subplots(figsize=(20, 10))
plt.bar(df_imp.index, df_imp.importance)
plt.grid()
plt.xticks(rotation=90)
plt.ylabel("importance")
plt.tight_layout()
plt.show()
[OUT]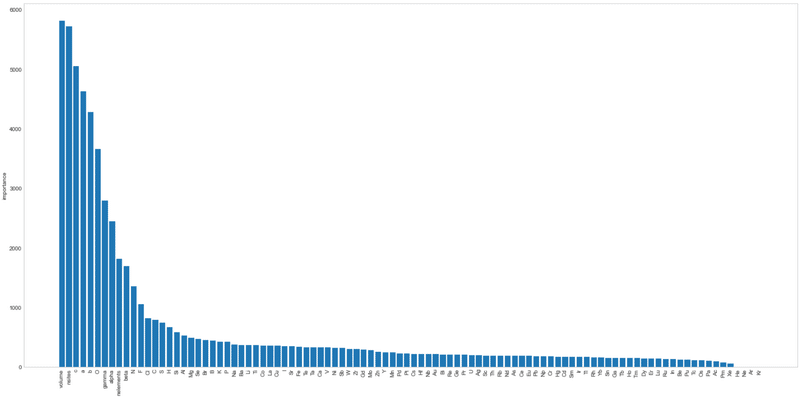

4-3.テストデータの推論・提出・結果確認
テストデータを同様の前処理をして推論後に所定のCSVの形にして提出しました。
[IN]
#CIFをParseして保存
# X_test_cif = Parse_df(df_test, col='cif')
# X_test_cif.to_pickle('tempdata/df_test_cif.pkl')
X_test_cif = pd.read_pickle('tempdata/df_test_cif.pkl')
X_test_cif = X_test_cif.drop(columns=['id', 'formula_pretty', 'cif'])
X_test_cif = X_test_cif.astype('float32')
preds = np.zeros(X_test_cif.shape[0]) #予測値を格納する配列
print(X_test_cif.shape, preds.shape)
#アンサンブル学習:全モデルの結果を平均化
for model in models:
pred_fold = model.predict(X_test_cif) #各foldのモデルでテストデータの予測値を計算
preds += pred_fold / n_folds #各foldの予測値を平均化
#提出用ファイルの作成
output = df_submit.copy() #提出用のデータフレームを作成
output['formation_energy_per_atom'] = preds #予測値を置換
output.to_csv('output/submit_LightGBM(特徴エンジ1)_R2.csv', index=False) #提出用のデータフレームをCSVファイルに出力
[OUT]
(28691, 98) (28691,) 前回提出結果から大きく改善(0.3764->0.2377)しました。これより特徴量エンジニアリングが非常に重要であることが分かります。
しかし順位としてはまだまだであり最低でもRMSE<0.2出ないと話にならなさそうです。

5.モデル実装:PyCaret(AutoML)
PyCaretで最低モデルの検証をします。各種詳細は前回の記事通りです。AutoMLの流れは下記の通りです。
回帰用のライブラリをインポート:”from pycaret.regression import *”
前処理:setup(<data>, target='<カラム名>')+必要な条件を入力
モデルの一括学習:"best=compare_models()"で交差検証(KFold)も含めて複数のモデルを学習
学習後のベストモデルの確認:"evaluate_model()"で最高性能のモデルのグラフを確認できる(※plot_model()でも同じ内容を確認可能)
個別のモデル作成:”create_model('compare_models()のindex')”で学習済みモデルのインスタンスを作成(※作成後はpredict_model()で推論して結果がcompare_models()と同じかを念のために確認)
ハイパーパラメータ調整:tune_model(model)で各モデルのハイパーパラメータ
テスト用データの推論:predict_model(data=<テストデータ>)で提出用のデータ作成
5-1.学習・検証
setup()で前処理、compare_models()で学習します。結果として「傾向は前回同様にTree系が強くRMSEは前回より改善されている」です。
なお前回はハイパーパラメータ調整で大した成果が出なかったため今回は飛ばしました。
[IN]
#ラベル付きデータの作成
dataset = df_train_cif_all.drop(columns=['id', 'formula_pretty', 'cif', 'val_fold'])
dataset = dataset.astype('float32')
from pycaret.regression import *
#PyCaretによるAutoML
#前処理:Setup
exp = setup(dataset,
target='formation_energy_per_atom', #目的変数
train_size=0.8, #学習データの割合
session_id=123, #乱数シード
log_experiment=True, #get_logs()でログを取得
silent=False, #ログを表示
use_gpu=True, #GPUを使用
fold=5, #交差検証の分割数
experiment_name='exp2')
[OUT]
[IN]
best = compare_models() #モデルの比較
[OUT]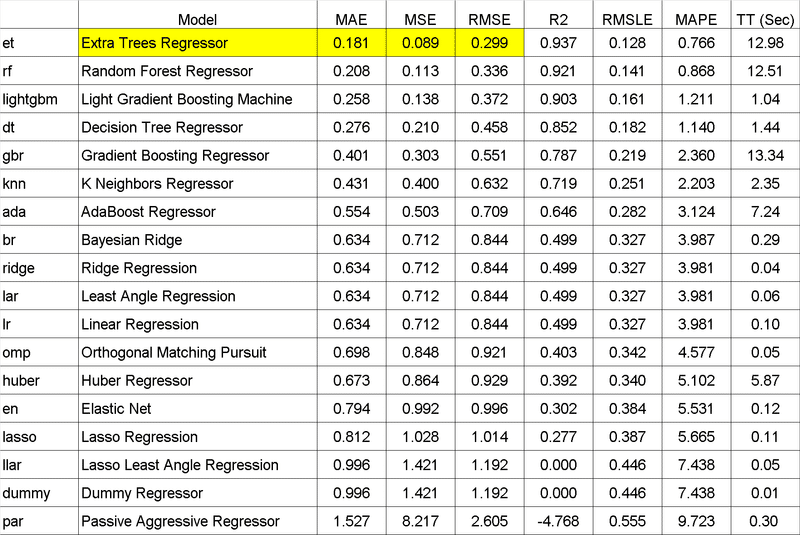
5-2.可視化・モデルの保存
可視化は”evaluate_model(<model>)”、推論は”predict_model(<model>)”で実施できます。
念のため学習済みモデルは保存もしておきます。
[IN]
#モデルの評価
# evaluate_model(best)
#推論
predict_model(best)
#ハイパーパラメータチューニング
# best_tuned = tune_model(best)
#モデルの保存
save_model(best, 'model/et_PyCaret(特徴エンジ)R2')
[OUT]
Model MAE MSE RMSE R2 RMSLE MAPE
0 Extra Trees Regressor 0.1675 0.0801 0.2830 0.9430 0.1208 0.5967
(Pipeline(memory=None,
steps=[('dtypes',
DataTypes_Auto_infer(categorical_features=[],
display_types=True, features_todrop=[],
id_columns=[], ml_usecase='regression',
numerical_features=[],
target='formation_energy_per_atom',
time_features=[])),
('imputer',
Simple_Imputer(categorical_strategy='not_available',
fill_value_categorical=None,
fill_value_numerical=Non...
ExtraTreesRegressor(bootstrap=False, ccp_alpha=0.0,
criterion='mse', max_depth=None,
max_features='auto', max_leaf_nodes=None,
max_samples=None,
min_impurity_decrease=0.0,
min_impurity_split=None,
min_samples_leaf=1, min_samples_split=2,
min_weight_fraction_leaf=0.0,
n_estimators=100, n_jobs=-1,
oob_score=False, random_state=123,
verbose=0, warm_start=False)]],
verbose=False),
'model/et_PyCaret(特徴エンジ)R2.pkl')5-3.テストデータの推論・提出・結果確認
前回は複数モデルで提出しましたが「学習・検証時でのスコアはテストデータと大きな差が無い」ため、今回はbestモデルだけで提出しました。
[IN]
#CIFをParseして保存
X_test_cif = pd.read_pickle('tempdata/df_test_cif.pkl')
X_test_cif = X_test_cif.drop(columns=['id', 'formula_pretty', 'cif'])
X_test_cif = X_test_cif.astype('float32')
print(X_test_cif.shape, preds.shape)
#提出用ファイルの作成
output = df_submit.copy() #提出用のデータフレームを作成
output['formation_energy_per_atom'] = predict_model(best, data=X_test_cif)['Label'] #予測値を置換
output.to_csv('output/submit_Et(特徴エンジ)_R2.csv', index=False) #提出用のデータフレームをCSVファイルに出力
[OUT]
(28691, 98) (28691,) 前回よりは結果は改善しており学習時の結果と同等スコアですが、ハイパラ調整済みのLightGBMには及びませんでした。
同系統のTree系ならハイパラ調整済みの方が性能は高そうです。

6.モデル実装:全結合
全結合モデルを作成していきたいと思います。記法に関しては下記記事の通りです。
6-1.モデルの作成
全結合のモデルを作成しました。フローは下記の通りです。
nn.Moduleを継承して全結合を定義した。構造は簡単に"入力次元数->256->128->64->32->1"とした。
今回はDropoutやBatchNormalizationなどの過学習防止手法は未設定
乱数は固定値に設定
入力値をStandarScalerで標準化(重みの誤差逆伝搬を均一化するため)
EarlyStoppingを設定(記事の通りGitHubからモジュール取得)
ホールドアウト法を行うため、訓練と検証用データに分割
GPUを使用するためにTensor化してDevice(cuda)に乗せる
ミニバッチ学習にするためにDatasetを作成して、DataLoaderに渡した。バッチ学習(全データ)を渡すと(私のPCでは)CUDAのメモリに乗りませんでした。
[IN]
import torch
import torch.nn as nn
import torch.nn.functional as F
import torch.optim as optim
from torchinfo import summary
from tqdm import tqdm
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
from pytorchtools import EarlyStopping
#データ読み込み
df_train_cif_all = pd.read_pickle('tempdata/df_train_cif.pkl') #保存したものを読み込み
targets = df_train_cif_all['formation_energy_per_atom'].values #目的変数(Numpy配列に変換)
#学習用のDataFrameを作成
# df_train_cif = df_train_cif_all.drop(columns=['id', 'formula_pretty', 'cif', 'formation_energy_per_atom', 'val_fold'])
class Net(nn.Module):
def __init__(self, input_dim, output_dim):
super(Net, self).__init__()
self.fc1 = nn.Linear(input_dim, 256) #入力層
self.fc2 = nn.Linear(256, 128) #中間層
self.fc3 = nn.Linear(128, 64) #中間層
self.fc4 = nn.Linear(64, 32) #中間層
self.fc5 = nn.Linear(32, output_dim) #出力層
def forward(self, x):
x = F.relu(self.fc1(x))
x = F.relu(self.fc2(x))
x = F.relu(self.fc3(x))
x = F.relu(self.fc4(x))
x = self.fc5(x)
return x
#乱数値の固定
def seed_everything(seed=42):
np.random.seed(seed%(2**32-1)) #Numpyの乱数
random.seed(seed) #Pythonの乱数
torch.manual_seed(seed) #PyTorchの乱数
torch.cuda.manual_seed(seed) #PyTorch(CUDA)
torch.backends.cudnn.deterministic =True #PyTorch(CUDA)
torch.backends.cudnn.benchmark = False #PyTorch(CUDA)
seed_everything()
#データ読み込み
df_train_cif = df_train_cif_all.drop(columns=['id', 'formula_pretty', 'cif', 'formation_energy_per_atom'])
df_target = df_train_cif_all['formation_energy_per_atom']
X, y = df_train_cif.values, df_target.values #Numpy配列に変換
#前処理(標準化)
scaler = StandardScaler()
scaler.fit(X)
X = scaler.transform(X) #平均値0、標準偏差1に変換
# display(pd.DataFrame(X).describe()) #確認
#テンソルに変換
X, y = torch.tensor(X, dtype=torch.float32), torch.tensor(y, dtype=torch.float32) #PyTorchのテンソルに変換
#データの分割
X_train, X_val, y_train, y_val = train_test_split(X, y, test_size=0.2, random_state=42)
print(f'X_train: {X_train.shape}, y_train: {y_train.shape}, X_val: {X_val.shape}, y_val: {y_val.shape}')
print(f'データ型 X_train: {X_train.dtype}, y_train: {y_train.dtype}, X_val: {X_val.dtype}, y_val: {y_val.dtype}')
#学習時のハイパラなど
device = torch.device('cuda' if torch.cuda.is_available() else 'cpu') #GPUが使えるか確認
epochs = 1000 #エポック数
lr = 0.005 #学習率
early_stopping = EarlyStopping(patience=10, verbose=True)
#Logging用
def logging_epoch(logs, epoch, loss): #学習結果を辞書に格納する関数
logs['epoch'].append(epoch) #学習回数を格納
logs['loss'].append(loss) #損失関数を格納
logs = {'train':{'epoch':[], 'loss':[]},
'val':{'epoch':[], 'loss':[]}} #学習結果を格納する辞書
#モデルの定義
net = Net(input_dim=X_train.shape[1], output_dim=1) #モデルの定義
critetion = nn.MSELoss() #損失関数
optimizer = optim.Adam(net.parameters(), lr=lr) #最適化手法
#GPUへの転送
X_train, y_train, X_val, y_val = X_train.to(device), y_train.to(device), X_val.to(device), y_val.to(device)
net = net.to(device)
#Datasetの作成
class TrainDataset(Dataset):
def __init__(self, X, y):
self.X = X
self.y = y
def __len__(self):
return len(self.X)
def __getitem__(self, idx):
return self.X[idx], self.y[idx]
train_dataset = DataLoader(TrainDataset(X_train, y_train), batch_size=32, shuffle=True)
for _ in train_dataset: break
print(f'size: {_[0].shape}, type: {_[0].dtype}')
[OUT]
X_train: torch.Size([57383, 98]), y_train: torch.Size([57383]), X_val: torch.Size([14346, 98]), y_val: torch.Size([14346])
データ型 X_train: torch.float32, y_train: torch.float32, X_val: torch.float32, y_val: torch.float32
size: torch.Size([32, 98]), type: torch.float326-2.学習・検証
学習・検証Phaseの大きな流れは下記の通りです。
事前に設定したepochsの回数だけ学習させるが、検証データのスコアが上昇しなくなったらEarly stoppingを実施
1epoch内で訓練と検証を1回ずつ実施
訓練データでは(バッチ学習だとCUDAにメモリが乗らないため)、ミニバッチ学習で学習させるためDataloaderで作成したオブジェクトをループで使用
特定のepoch回数ごとにlogを取得+Early stoppingの判定確認
モデルの保存(※Early stoppingモジュールによる自動保存)
[IN]
#学習
for epoch in tqdm(range(epochs)):
for phase in ['train', 'val']:
if phase == 'train':
net.train() #モデルを訓練モードに
else:
net.eval() #モデルを評価モードに
if phase == 'train':
for X_train, y_train in train_dataset:
outputs = net(X_train) #モデルの出力
optimizer.zero_grad() #勾配の初期化
loss = critetion(outputs, y_train.view(-1, 1)) #損失関数の計算
loss.backward() #勾配の計算
optimizer.step() #パラメータの更新
if epoch % 10 == 0:
if phase == 'train':
logging_epoch(logs['train'], epoch, loss.item())
elif phase == 'val':
y_pred_val = net(X_val)
loss_val = critetion(y_pred_val, y_val.view(-1, 1))
logging_epoch(logs['val'], epoch, loss_val.item())
early_stopping(loss_val.item(), net)
if early_stopping.early_stop:
print('Early stopping')
break
[OUT]
0%| | 1/1000 [00:03<53:58, 3.24s/it]Validation loss decreased (0.048042 --> 0.046796). Saving model ...
1%| | 11/1000 [00:34<52:06, 3.16s/it]Validation loss decreased (0.046796 --> 0.041543). Saving model ...
2%|▏ | 21/1000 [01:06<51:51, 3.18s/it]Validation loss decreased (0.041543 --> 0.037175). Saving model ...
3%|▎ | 31/1000 [01:38<51:08, 3.17s/it]EarlyStopping counter: 1 out of 10
4%|▍ | 41/1000 [02:09<50:38, 3.17s/it]EarlyStopping counter: 2 out of 10
5%|▌ | 51/1000 [02:41<50:08, 3.17s/it]Validation loss decreased (0.037175 --> 0.036907). Saving model ...
6%|▌ | 61/1000 [03:13<49:36, 3.17s/it]EarlyStopping counter: 1 out of 10
7%|▋ | 71/1000 [03:45<49:01, 3.17s/it]EarlyStopping counter: 2 out of 10
8%|▊ | 81/1000 [04:16<49:01, 3.20s/it]EarlyStopping counter: 3 out of 10
9%|▉ | 91/1000 [04:49<48:47, 3.22s/it]EarlyStopping counter: 4 out of 10
10%|█ | 101/1000 [05:21<48:35, 3.24s/it]EarlyStopping counter: 5 out of 10
11%|█ | 111/1000 [05:54<48:00, 3.24s/it]EarlyStopping counter: 6 out of 10
12%|█▏ | 121/1000 [06:27<47:49, 3.26s/it]EarlyStopping counter: 7 out of 10
13%|█▎ | 131/1000 [06:59<46:59, 3.24s/it]EarlyStopping counter: 8 out of 10
14%|█▍ | 141/1000 [07:31<46:09, 3.22s/it]EarlyStopping counter: 9 out of 10
15%|█▌ | 150/1000 [08:03<45:42, 3.23s/it]EarlyStopping counter: 10 out of 10
Early stoppingなお「Early Stoppingの記事」で実行すると”checkpoint.pt”にモデルの重みが保存されます。こちらは後ほど読み込み可能です。
6-3.可視化
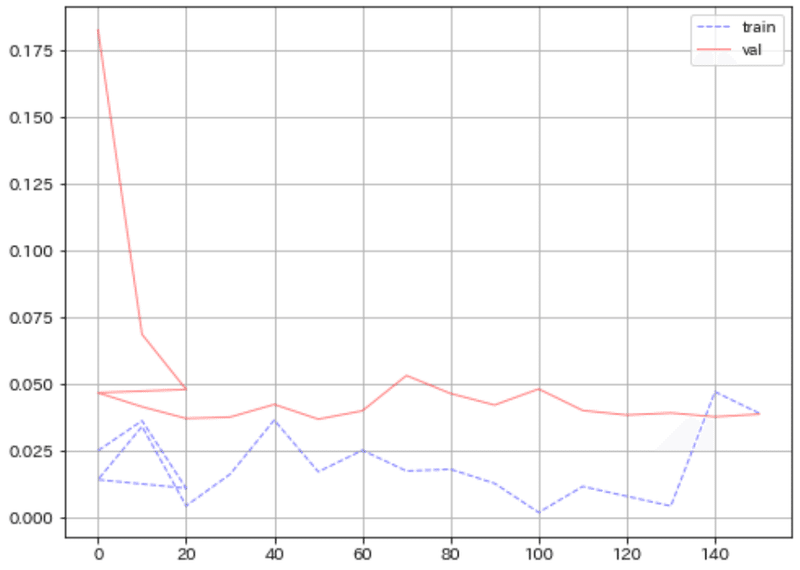
今回作成したモデルの中身と学習時の結果を可視化しました。
[IN]
summary_model = summary(net, (X_train.shape[1],)) # モデルの概要を表示
print(summary_model)
#学習時・検証時の損失の推移を可視化
fig, ax = plt.subplots(1, 1, figsize=(8, 6))
ax.plot(logs['train']['epoch'], logs['train']['loss'],
label='train', color='blue', linewidth=1, alpha=0.5, linestyle='--')
ax.plot(logs['val']['epoch'], logs['val']['loss'],
label='val', color='red', linewidth=1, alpha=0.5, linestyle='-')
plt.grid(), plt.legend()
plt.show()
[OUT]
==========================================================================================
Layer (type:depth-idx) Output Shape Param #
==========================================================================================
Net [1] --
├─Linear: 1-1 [256] 25,344
├─Linear: 1-2 [128] 32,896
├─Linear: 1-3 [64] 8,256
├─Linear: 1-4 [32] 2,080
├─Linear: 1-5 [1] 33
==========================================================================================
Total params: 68,609
Trainable params: 68,609
Non-trainable params: 0
Total mult-adds (M): 11.29
==========================================================================================
Input size (MB): 0.00
Forward/backward pass size (MB): 0.00
Params size (MB): 0.27
Estimated Total Size (MB): 0.28
==========================================================================================
6-4.テストデータの推論・提出・結果確認
今回は下記3パターンで提出してみました。結果は1,2,3の順で性能が良くなりました。
Early Stoppingなし、学習率lr=0.01
Early Stoppingあり、stop確認のepoch単位:50回毎、学習率lr=0.01
Early Stoppingあり、stop確認のepoch単位:10回毎、学習率lr=0.005
Early Stoppingを使用する場合”checkpoint.pt”にモデルの重みが保存されているため上書きでモデルに重みを読み込みます。
[IN]
print(early_stopping.path)
net.load_state_dict(torch.load(early_stopping.path))#ベストモデルの読み込み(上書き)
[OUT]
-後は他と同様に①テストデータの読み込み、②学習時と同じ前処理、③提出の形にそろえて推論・ファイル化します。
[IN]
#テストデータ作成
X_test_cif = pd.read_pickle('tempdata/df_test_cif.pkl')
X_test_cif = X_test_cif.drop(columns=['id', 'formula_pretty', 'cif'])
#前処理
X_test_cif = scaler.transform(X_test_cif) #標準化
X_test_cif = torch.tensor(X_test_cif, dtype=torch.float32) #テンソル化
X_test_cif = X_test_cif.to(device) #GPUへの転送
#提出用ファイルの作成
output = df_submit.copy()
output['formation_energy_per_atom'] = net(X_test_cif).detach().cpu().numpy() #予測値の計算
print(output.shape)
output.to_csv('output/submit_NN(特徴エンジ+lr調整)_Earlystopping_R2.csv', index=False)
[OUT]結果は下記の通りです。LightGBMよりも高い性能を得ることができました。データが豊富とはいえ、離散値を使用しているためモデルの適用範囲が広がっているのではと思います。


7.所感
「特徴量エンジニアリングがめっちゃ重要」ということは身に染みて理解できました。詰まる所、モデルが凄くても重要なデータが無いと何も結果がでない(Garbage In, Garbage Out)ということですね。
後はもう少し体系的に実施できたらよいがいかんせん経験が少なすぎるのがつらい。
Googleが深層学習のノウハウを公開して話題だけど、実際読んで想像以上に価値を感じた。
— goto@meta翻訳開発者 (@goto_yuta_) January 20, 2023
例えば「バッチサイズはバリデーションセットでの性能に影響
せず、むしろバッチサイズを下げてサンプルがばらついて正則化の効果を持ったりする」みたいの詳細知識が満載だった...https://t.co/LZNiRxiOgW
参考資料
あとがき
次の方針は下記の通り。
全結合モデルにDropoutやBatchNormalizationを追加
”CIF情報からまだ使っていない情報を使用”か”外部データで使えそうなやつを使用”
単位格子中の非対称単位数(_symmetry_Int_Tables_number)と空間群の情報(_symmetry_space_group_name_H-M)はすぐに適用可能:空間群は学習・テストをまとめてユニークで取った後にDataFrameに追加(多分40-50次元くらい増加
交互作用特徴量もありだと思うけど、スパースなデータに対してとると次元数がものすごく増えて実用的ではないため最終手段
この記事が気に入ったらサポートをしてみませんか?
