【NISHIKAコンペ】健診データによる肝疾患判定
今回NISIKAの練習問題をサブミットしてみました!
from google.colab import drive
drive.mount('/content/drive/')Colaboratoryで行うのでまずはデータのマウントから
import pandas as pd
import numpy as np
from matplotlib import pyplot as plt
from sklearn.model_selection import train_test_split
from sklearn.linear_model import LinearRegression as LR
from sklearn.metrics import mean_squared_error as MSE
from pydrive.auth import GoogleAuth
from pydrive.drive import GoogleDrive
from google.colab import auth
from oauth2client.client import GoogleCredentials
import pandas as pd
import numpy as np
from matplotlib import pyplot as plt
from sklearn.model_selection import train_test_split
from sklearn.linear_model import LinearRegression as LR
import seaborn as sns ひひとまず、必要そうなやつを入れる
train.tail()
test.head()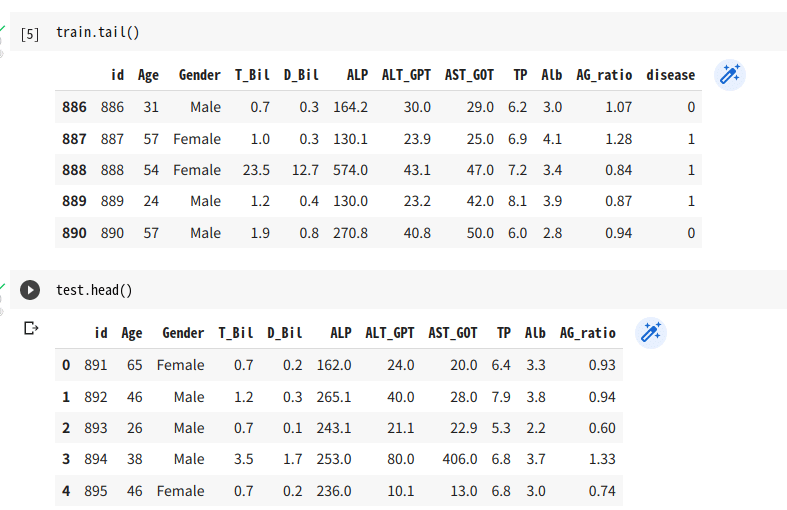
test.isnull().sum()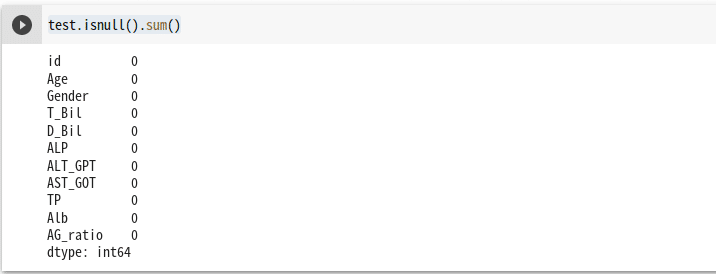
testは欠損はなさそう。
trainはあった。
train_test = pd.concat([train,test],ignore_index = True) #Genderラベリング
dummy_df = pd.get_dummies(train_test["Gender"])
train_test = pd.concat([train_test,dummy_df],axis = 1)
train_test = train_test.drop(columns=['Gender'])
train_test = train_test.drop(columns=['id'])train,testを一旦結合。
Genderをワンホットにする。
obujectのGenderを消す。idも消す。
train_test.info()ここで一旦確認 obujectはなし。
train_test.info()
結合した分に欠損あり、testになかったのでtrainにある。
このAG_ratioは計算で求められるもの
train_test["AG_ratio"].fillna(train_test["Alb"]/(train_test["Alb"]*train_test["TP"]),inplace = True)
train_test.isnull().sum()ここれでnullをなくす。
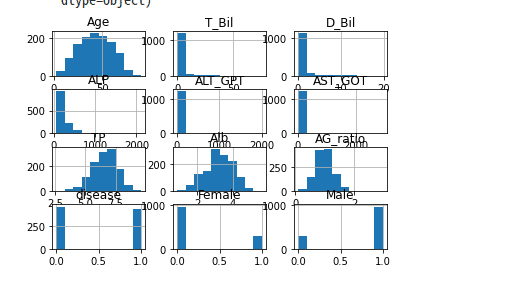
train_test.hist()ひヒストグラムを確認
#列名をリスト化して
prods = train_test.columns.tolist()
prods
#疾患のありなしで確認する 。
disease_0= train_test.query("disease== 0")[prods[2]]
disease_1 = train_test.query("disease== 1")[prods[2]]
sns.distplot(disease_0)
sns.distplot(disease_1)
plt.show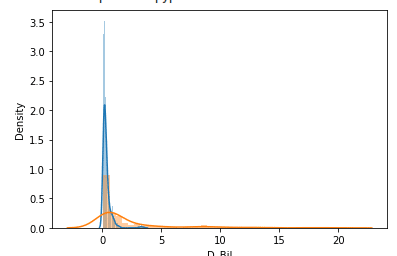
これから、疾患なしの人はだいたい4以上は存在しなさそう。あと、マイナスの方にも多くは存在しない。この辺りで、特徴量をつくる。
#bining区切りをつける 。
# 上記から
bins_D_Bil = [0, 0.5, 1,1.5, 2]
# T_Bil列を分割し、0始まりの連番でラベル化した結果を、X_cutに格納する
X_cut, bin_indice = pd.cut(train_test["D_Bil"], bins=bins_D_Bil, retbins=True, labels=False)
# bin分割した結果をダミー変数化 (prefix=X_Cut.nameは、列名の接頭語を指定している)
X_dummies = pd.get_dummies(X_cut, prefix=X_cut.name)
# 元の説明変数のデータフレーム(X)と、ダミー変数化の結果(X_dummies)を横連結
train_test_bins_T_D = pd.concat([train_test_bins, X_dummies], axis=1)ここれから、特徴量をたくさん作る、そして、l1正則化で特徴量を減らす。
LogisticRegressionの引数をsolver='liblinear'とする。
この記事が気に入ったらサポートをしてみませんか?
