R言語による統計解析~ggplot(ggpaired)にはレイヤーが存在する~
R言語を用いた統計解析において、ggplot(と、付随したパッケージ達)は必須といっても過言ではないだろう。データの可視化、論文に即用いることの出来るfigure作成、間違いなく役立つパッケージだ。
付随したパッケージの中でとても素敵だなと思ったのがggpaired、である。ggpairedの簡単な紹介に加え、いじっていく中でみつけたレイヤーの考え方について、備忘録として記しておく。どなたかの参考になれば幸いである。
ggpairedの簡単な解説
ggplotに付随したパッケージであり、「治療前→治療後」のように対応する二群を比較したりするのに有用な可視化を提供してくれる。基本的な構造はggplotとほとんど同じなので、プロフェッショナルな方々なら楽勝だろう(私はめちゃくちゃ苦労した)
試しにggpariedを描画してみる
サンプルデータを作る。
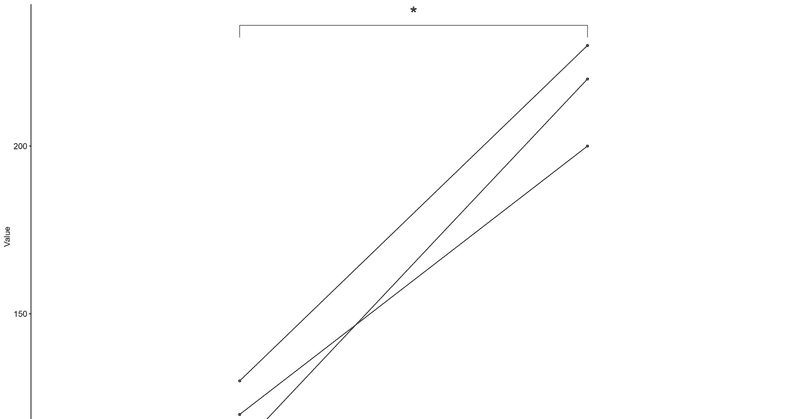
library(tidyr)
sampledata<-data.frame(row.names=c("subject1", "subject2", "subject3"),before=c(130,110,120),after=c(200,230,220))
print(sampledata)
before after
subject1 130 200
subject2 110 230
subject3 120 220
プロットしてみる。・・・・と行きたいところだが、ggplot系の落とし穴があるのだ。ggplot用にデータ形式を変更しないといけない。そこで役に立つのがpivot_longerだ。
long_data <- pivot_longer(sampledata, cols = c(before, after), names_to = "time", values_to = "value")
print(long_data)
# A tibble: 6 × 2
time value
<chr> <dbl>
1 before 130
2 after 200
3 before 110
4 after 230
5 before 120
6 after 220longerが縦長になり、widerが横長になる仕組みだ。cols=を用いて、縦長にしたい列名を指定している。
いよいよプロットを描画する。
library(GGally)
p <- ggpaired(data = long_data, x = "time", y = "value")
ggsave("Figure1.png", p, width = 16.2, height =10 , dpi = 300)これで出力されたのがこちら。ggsaveもぜひ使って欲しい。

さらにすごいのがこちら。
p2 <- ggpaired(data = long_data, x = "time", y = "value") +
stat_compare_means(comparisons = list(c("before", "after")),
method = "t.test",label = "p.signif", paired=TRUE, size=10,
size = 10)
ggsave("Figure2.png", p2, width = 16.2, height =10 , dpi = 300)
このように、検定まで出来てしまう。細かな記載方法についてはマニュアルも参照してほしいが、これだけでも超便利なのが伝わると思う。
Boxplotが邪魔だなあ、そう思ったあなたへ。layersをいじる
ここからが少し手を加えた部分だ。検体数も少ないし、boxplotいらなくね?って思った方の役に立ちたいと思い、記載したい。
names(p2)
[1] "data" "layers" "scales" "mapping" "theme" "coordinates" "facet" "plot_env"
[9] "labels" 他の記事でも記載したが、とりあえずnames関数を使ってみることで見えてくるものがある。layersという怪しい要素が格納されている。
p2$layers
[[1]]
mapping:
geom_boxplot: outlier.colour = NULL, outlier.fill = NULL, outlier.shape = 19, outlier.size = 1.5, outlier.stroke = 0.5, outlier.alpha = NULL, notch = FALSE, notchwidth = 0.5, varwidth = FALSE, na.rm = FALSE, orientation = NA, width = 0.5
stat_boxplot: na.rm = FALSE, orientation = NA, width = 0.5
position_identity
[[2]]
mapping: group = ~id
geom_line: na.rm = FALSE, orientation = NA
stat_identity: na.rm = FALSE
position_identity
[[3]]
mapping:
geom_point: na.rm = FALSE
stat_identity: na.rm = FALSE
position_identity
[[4]]
geom_signif: na.rm = FALSE, extend_line = 0, parse = FALSE, orientation = NA
stat_signif: na.rm = FALSE, comparisons = list(c("before", "after")), test = t.test, test.args = list(paired = TRUE), annotations = NULL, map_signif_level = c(`****` = 1e-04, `***` = 0.001, `**` = 0.01, `*` = 0.05, ns = Inf), y_position = NULL, xmin = NULL, xmax = NULL, margin_top = 0.05, step_increase = 0.12, tip_length = 0.03, manual = FALSE, orientation = NA
position_identity これを見るとlayers[1]にboxplotが格納されていそうなことがわかる。boxplotを消すには・・・
p2$layers <- p2$layers[-1]これだけ!

ちなみ、再度layer構造を確認してみると、
p2$layers
[[1]]
mapping: group = ~id
geom_line: na.rm = FALSE, orientation = NA
stat_identity: na.rm = FALSE
position_identity
[[2]]
mapping:
geom_point: na.rm = FALSE
stat_identity: na.rm = FALSE
position_identity
[[3]]
geom_signif: na.rm = FALSE, extend_line = 0, parse = FALSE, orientation = NA
stat_signif: na.rm = FALSE, comparisons = list(c("before", "after")), test = t.test, test.args = list(paired = TRUE), annotations = NULL, map_signif_level = c(`****` = 1e-04, `***` = 0.001, `**` = 0.01, `*` = 0.05, ns = Inf), y_position = NULL, xmin = NULL, xmax = NULL, margin_top = 0.05, step_increase = 0.12, tip_length = 0.03, manual = FALSE, orientation = NA
position_identity [1]を抜くと他のlayersは繰り上がるようですね。
figure作りは解析の最終局面でしかないかもしれませんが、せっかくなら綺麗なものを作りたいですよね。どなたかの参考になれば幸いです。
この記事が気に入ったらサポートをしてみませんか?
